На главной странице Pfam ищем по ID (раздел JUMP TO). По полученным данным можно составить следующую таблицу:
Доменная структура белка SPSE_BACSU по данным Pfam
Cхема из Pfam: |
|||||
| Пояснения к схеме |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов |
Положение в последовательности белка GUAD_BACSU | Клан |
| 1. | PF03102 | NeuB | NeuB это прокариотическая N-ацетилнейраминовая кислота(Neu5Ac)синтаза. Она катализирует прямой синтез Neu5Ac (наиболее распространенной сиаловой кислоты)путем конденсации фосфоенолпирувата (PEP) и N-ацетилманнозамин (ManNAc). Эта реакция наблюдалась только у прокариот, эукариоты синтезируют 9-фосфат форму, Neu5Ac-9-P, и используют ManNAc-6-P вместо ManNAc. | 37-276 | Семейство принадлежит клану TIM_barrel (CL0036), в котором находится всего около 55 членов |
| 2. | PF08666 | SAF | Это семейство доменов включает в себя целый ряд различных белков. Такие, как белки-антифриз и белки жгутика FlgA, и CpaB белки пили. | 304-365 | Это семейство принадлежит клану AFP_III-like (CL0489), которое состоит из двух членов. |
2. Изучение домена PF03102 белка SPSE_BACSU
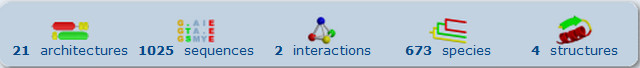
| Во сколько разных архитектур входит домен? | 21 |
| Для какого числа белков, содержащих домен, известна последовательность? | 1025 |
| Для какого числа разных белков, содержащих домен, определена пространственная структура (домена или всего белка)? | 4 |
| Выравнивание "seed" фрагментов белков, соответствующих домену | PF03102_seed.txt |
3. Описание доменной архитектуры, в которой присутствует два или более разных домена
Белок SPSE_BACSU включает два домена, поэтому возьмем именно эту архитектуру

В разделе Species доступна информация распределении числа последовательностей с рассматриваемым доменом по таксонам.
| Таксон | Количество находок в последовательностях с доменом PF03102 | Количество находок в последовательностях с доменом PF08666 | |
| Эукариоты | Зеленые растения (Viridiplantae) | 1 | 2 |
| Грибы (Fungi) | 0 | 0 | |
| Животные (Metazoa) | 52 | 78 | |
| Остальные эукариоты | 2 | 0 | |
| Археи (Archaea) | 15 | 37 | |
| Бактерии (Bacteria) | 953 | 3134 | |
| Вирусы (Viruses) | 0 | 0 | |
Домены совсем не представлены в таких таксонах, как грибы и вирусы, и единичны в зеленых растениях. Наиболее домены представлены в последовательностях белков бактерий, видимо, многим эта архитектура просто необходима для нормальной жизнедеятельности
4. Сравнение описания мотивов в разных банках семейств по данным InterPro
На главной странице InterPro ищем по Uniprot ID.

На основе полученной информации можно сделать следующие утверждения:
- Самый короткий мотив - G3DSA:3.90.1210.10. InterPro ID: IPR006190. Мотив описан в банке CATH (protein structure classification).
- Самый длинный мотив - PTHR23416. Но ему не присвоен ID и ни в каком банке не описан. Поэтому можно взять следующий по длине мотив - G3DSA:3.20.20.70 InterPro ID: IPR013785. Мотив описан в банке EMBL-EBI.
- В InterPro также интегрированы следующие структурные подписи: 1vliA, 1vliA01(3.20.20.70), 1vliA02(3.90.1210.10) и d1vlia1 (b.85.1.1), d1vlia2 (c.1.10.6)
- Границы структурных доменов практически совпадают с границами доменов Pfam (37-274 против 37-276, 308-364 против 304-365).