Задание 1.
Построить модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA
С помощью программы Putty :
Используем следующие команды для указания пути к 3DNA:
export X3DNA=/home/preps/golovin/progs/X3DNA
С помощью программы fiber пакета 3DNA получаем A-,B- и Z-формы дуплекса ДНК. Последовательность одной нити - "gatc" 5 раз. Ввод с клавиатуры
fiber -b gatc-b.pdb
fiber -z gatc-z.pdb
Структуры дуплекса:
A-форма
B-форма
Z-форма
Задание 2
Упражнение 1
Научиться выделять разные атомы и химические группировки, используя предопределенные подомножества RasMol
|
|
|
| Сахарофосфатный остов А-формы ДНК | Сахарофосфатный остов А-формы ДНК |
|
|
|
| Все нуклеотиды | Все аденины |
|
|
|
| Все гуанины с выделенным атомом N7 | Первый гуанин с выделенным атомом N7 |
|
|
|
| Сахарофосфатный остов B-формы ДНК | Сахарофосфатный остов B-формы ДНК |
|
|
|
| Все нуклеотиды | Все аденины |
|
|
|
| Все гуанины с выделенным атомом N7 | Первый гуанин с выделенным атомом N7 |
|
|
|
| Сахарофосфатный остов Z-формы ДНК | Сахарофосфатный остов Z-формы ДНК |
|
|
|
| Все нуклеотиды (G и C) | Все аденины (их нет) |
|
|
|
| Все гуанины с выделенным атомом N7 | Первый гуанин с выделенным атомом N7 |
Скрипт для получения картинок
Упражнение 2
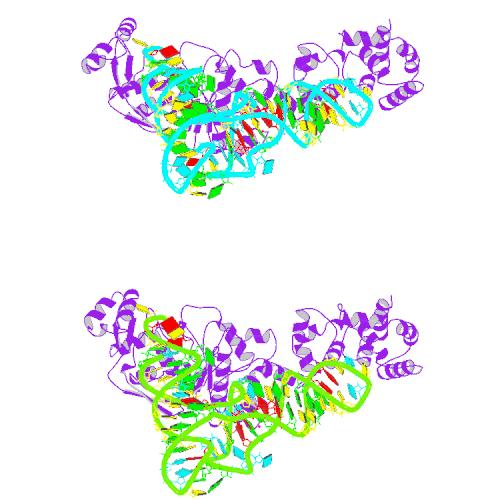
Thermus thermophilus glutamyl-tRNA synthetase complexed with tRNA(Glu) and glutamol-AMP
1N78
|
DNA-BINDING DOMAIN OF INTRON ENDONUCLEASE I-TEVI WITH ITS SUBSTRATE
1I3J
|
Упражнение 3
Разрывы в структурах нуклеиновых кислот из 1N78 и 1I3J не обнаружены.
|
|
|
| Изображение нуклеиновой кислоты из 1N78 в проволочной модели | Изображение нуклеиновой кислоты из 1I3J в проволочной модели |
Задание 3
Упражнение 1
Выбрала девятый гуанин. Красным отмечены атомы, обращенные в сторону большой бороздки, синим цветом - в сторону малой бороздки.
Молекула гуанина, построенная в программе ChemSketch. Обращенные атомы выделены тем же цветом.
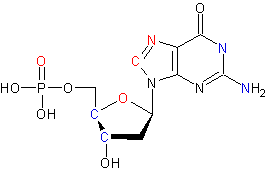
В таблице отмечены: (B) - атомы обращены в сторону большой бороздки(красный цвет), (b) - атомы обращены в сторону маленькой бороздки(синий цвет)
ATOM 165 P G A 9 -8.877 2.510 -25.152 ATOM 166 O1P G A 9 -10.072 2.945 -24.395 ATOM 167 O2P(B)G A 9 -9.120 1.561 -26.260 ATOM 168 O5' G A 9 -8.094 3.791 -25.705 ATOM 169 C5'(b)G A 9 -7.132 4.443 -24.854 ATOM 170 C4'(b)G A 9 -5.883 4.778 -25.645 ATOM 171 O4' G A 9 -4.919 3.686 -25.648 ATOM 172 C3' G A 9 -6.097 5.085 -27.127 ATOM 173 O3' G A 9 -5.197 6.115 -27.518 ATOM 174 C2' G A 9 -5.704 3.797 -27.850 ATOM 175 C1' G A 9 -4.491 3.471 -26.984 ATOM 176 N9 G A 9 -4.033 2.058 -27.101 ATOM 177 C8 (B)G A 9 -4.777 0.915 -27.294 ATOM 178 N7 (B)G A 9 -4.063 -0.183 -27.354 ATOM 179 C5 G A 9 -2.753 0.265 -27.190 ATOM 180 C6 G A 9 -1.535 -0.463 -27.165 ATOM 181 O6 (B)G A 9 -1.358 -1.672 -27.284 ATOM 182 N1 G A 9 -0.435 0.386 -26.975 ATOM 183 C2 G A 9 -0.504 1.758 -26.828 ATOM 184 N2 (b)G A 9 0.660 2.389 -26.656 ATOM 185 N3 (b)G A 9 -1.648 2.440 -26.851 ATOM 186 C4 G A 9 -2.724 1.631 -27.035
Упражнение 2
|
A-форма |
B-форма |
*Z-форма |
Тип спирали (правая или левая) |
правая |
правая |
левая |
Шаг спирали (A) |
28,03 |
33,75 |
|
Число оснований на виток |
11 |
10 |
|
Ширина большой бороздки |
16,97 |
17,91 |
|
Ширина малой бороздки |
7,98 |
11.69 |
|
|
|
|
| Большая бороздка А-формы | Малая бороздка А-формы |
|
|
|
| Большая бороздка B-формы | Малая бороздка B-формы |
Сравнение торсионных углов в структурах А- и В-форм.
| α | β | γ | δ | ε | ξ | χ |
| A-форма (презентация) | ||||||
| -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| А-форма (RasMol) | ||||||
| -51.7 | 174.79 | 41.71 | 79.07 | -147.78 | -75.05 | 22.76 |
| B-форма (презентация) | ||||||
| -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
| В-форма(RasMol) | ||||||
| -29.89 | 136.35 | 31.14 | 143.40 | -140.77 | -160.50 | 82.09 |
A-форма Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 174.8 41.7 79.0 -147.8 -75.1 -157.2 2 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 7 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 C -51.7 174.8 41.7 79.0 -147.8 -75.0 -157.2 9 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 C -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 13 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 16 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 17 G -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 18 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 19 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 20 C -51.7 174.8 41.7 79.1 --- --- -157.2 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -51.7 174.8 41.7 79.0 --- --- -157.2 2 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 7 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 9 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 13 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 16 G -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 17 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 18 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 19 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 20 G --- 174.8 41.7 79.1 -147.8 -75.1 -157.2 B-форма Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0 2 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 4 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 5 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 9 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 10 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 14 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 15 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 17 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 18 A -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 19 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 20 C -29.9 136.3 31.2 143.3 --- --- -98.0 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -29.9 136.4 31.1 143.4 --- --- -98.0 2 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 4 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 5 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 9 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 10 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 14 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 15 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 17 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 18 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 19 A -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 20 G --- 136.3 31.2 143.3 -140.8 -160.5 -98.0 Z-форма Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 179.0 -173.8 94.9 -103.6 -64.8 58.7 2 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 3 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 4 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 5 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 6 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 7 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 8 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 9 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 10 C -139.5 -136.8 50.8 137.6 --- --- -154.3 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -139.5 -136.7 50.9 137.6 --- --- -154.3 2 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 3 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 4 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 5 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 6 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 7 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 8 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 9 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 10 G --- 179.0 -173.8 94.9 -103.6 -64.8 58.7
Можно заметить, что во всех структурах углы при разных основаниях практически совпадают
| α | β | γ | δ | ε | ξ | χ |
| A-форма | ||||||
| -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.8 |
| B-форма | ||||||
| -29.9 | 136.4 | 31.1 | 143.4 | -140.8 | -160.5 | -98.0 |
| Z-форма (для гуанинов) | ||||||
| 51.9 | 179 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
| Z-форма (для цитозинов) | ||||||
| -139.5 | -136.7 | 50.9 | 137.6 | -96.5 | 81.9 | -154.3 |
Из таблицы видно, что:
В форме A от предыдущей таблицы сильно отличаются углы ε
В форме В сильно отличаются от предыдущей таблицы углы ε и ξ
Углы ε сходны по значению в формах А и В, но сильно отличаются в форме Z.
Форма Z вообще имеет разные значения углов для разных оснований (для гуанина и цитозина)
Торсионные углы тРНК 1N78
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 170.0 50.9 86.6 -146.4 -80.0 -172.7 2 G -79.4 -175.8 54.9 84.0 -147.5 -70.4 -161.8 3 C -66.9 169.8 54.4 82.7 -150.0 -72.7 -164.3 4 C -67.9 173.7 53.9 81.6 -152.4 -79.4 -161.7 5 C -61.6 169.7 54.8 81.2 -151.3 -72.3 -166.5 6 C -61.7 171.4 58.2 87.3 -156.2 -79.4 -164.9 7 A -51.5 174.8 59.9 137.6 --- --- -122.1 8 G --- 142.7 40.0 81.9 -148.5 -73.1 -173.8 9 G -61.7 -179.6 48.0 81.3 -153.7 -69.3 -170.1 10 G -59.4 -178.8 44.5 81.1 -171.6 -78.4 -159.2 11 G 160.5 -157.2 168.6 85.9 -107.1 -47.3 176.5 12 G -108.7 90.8 151.8 88.8 -117.5 -68.2 163.3 13 U -68.6 -180.0 45.5 82.5 -125.2 -77.6 -162.3 14 U -52.7 155.6 50.9 81.0 --- --- -153.5 15 A --- 167.5 48.8 85.3 -141.5 -63.3 -167.8 16 G -66.0 173.7 52.0 81.7 -152.2 -77.5 -164.9 17 G -59.7 174.8 48.9 82.0 -149.3 -71.5 -166.0 18 C -65.0 174.3 53.2 82.6 -155.0 -68.8 -159.4 19 C -70.3 174.5 57.1 78.3 -153.2 -69.3 -166.3 20 G -57.1 174.6 54.7 81.1 -155.7 -81.6 -164.7 21 A -63.2 173.9 50.5 82.4 --- --- -154.3 22 G --- 178.1 52.4 88.3 -148.4 -67.7 -178.6 23 U -70.4 -175.8 48.0 81.6 -154.2 -71.0 -164.7 24 C -65.9 -174.2 48.5 81.5 -152.9 -66.4 -159.7 25 U 140.4 -155.7 -167.0 83.4 -153.3 -77.5 -171.5 26 A -57.7 -174.8 45.3 79.8 -139.4 -66.7 -170.7 27 G -43.4 161.1 58.8 129.6 --- --- -119.6 28 G --- -131.3 48.7 114.2 --- --- -64.1 29 G --- -172.0 45.6 87.6 -148.4 -76.0 -168.5 30 G -72.6 -177.1 47.3 85.4 -149.1 -69.6 -163.9 31 C -72.8 172.1 55.8 84.4 -148.2 -75.4 -157.8 32 C -54.9 171.7 42.9 81.4 -152.6 -78.8 -158.0 33 C -60.9 170.1 54.8 79.6 -150.6 -70.8 -166.3 34 C -63.3 169.5 59.0 84.2 -160.7 -75.5 -160.5 35 A -42.2 -171.5 39.8 98.9 --- --- -115.4 36 G --- 147.1 33.3 82.1 -147.3 -73.4 -168.1 37 G -60.5 179.2 46.7 80.6 -154.6 -70.2 -167.0 38 G -58.3 177.2 45.9 82.3 -162.1 -89.1 -161.4 39 G 156.9 -149.8 166.7 85.5 -103.1 -52.8 175.5 40 G -101.2 90.6 151.4 88.2 -111.0 -74.5 160.1 41 U -68.1 176.7 44.9 84.2 -126.0 -77.3 -160.5 42 U -52.7 157.2 50.2 81.7 --- --- -152.3 43 A --- 164.4 53.8 85.4 -139.0 -60.9 -169.5 44 G -67.7 174.0 49.9 79.9 -153.5 -73.6 -161.3 45 G -60.5 173.5 48.6 79.9 -151.6 -72.9 -168.6 46 C -63.4 177.6 50.8 82.4 -155.8 -67.8 -157.5 47 C -67.4 171.7 59.1 79.4 -153.6 -70.3 -167.5 48 G -59.2 174.4 55.1 81.4 -152.5 -80.6 -164.1 49 A -65.8 170.3 54.6 79.7 --- --- -155.4 50 G --- 177.3 48.1 88.1 -154.0 -63.5 -172.9 51 U -66.6 -176.2 47.6 82.4 -152.0 -71.8 -165.9 52 C -68.6 -174.3 48.6 81.6 -154.7 -69.3 -158.6 53 U 144.9 -154.1 -169.6 84.6 -149.2 -81.2 -174.1 54 A -54.7 -176.8 43.7 78.4 -142.7 -62.5 -168.6 55 G -49.0 168.8 61.4 126.3 --- --- -121.4 56 G --- -130.7 46.6 113.9 --- --- -59.7 Strand II base alpha beta gamma delta epsilon zeta chi 1 C 157.9 -143.8 161.4 87.5 --- --- -168.9 2 U -70.0 176.4 52.4 83.8 -168.0 -94.2 -160.5 3 G -47.2 160.9 54.1 82.8 -149.9 -65.8 -174.1 4 G -64.4 -178.5 51.0 84.1 -153.2 -79.1 -169.2 5 G -67.4 178.8 47.4 79.5 -163.2 -65.5 -166.9 6 G -64.8 178.2 47.2 83.1 -160.2 -67.8 -159.0 7 U -73.6 179.7 54.5 83.4 -157.5 -67.0 -158.2 8 C -65.0 177.1 55.6 84.4 -156.9 -60.6 -163.4 9 C -63.4 168.9 55.7 82.2 -154.5 -65.1 -162.6 10 C -73.5 -169.5 49.6 84.4 -160.3 -76.4 -155.6 11 C -70.7 -175.7 51.4 83.8 -159.3 -67.7 -163.7 12 C --- -150.3 47.5 82.2 -158.3 -74.2 -165.5 13 A --- -96.6 179.1 81.6 --- --- -149.1 14 G --- -149.9 -64.9 108.9 --- --- -115.4 15 C -61.1 167.7 51.8 85.0 --- --- -162.5 16 C -63.2 173.9 53.7 82.8 -141.4 -70.0 -159.9 17 C -66.3 172.8 55.1 83.9 -154.5 -70.5 -164.9 18 G -64.4 178.8 53.5 83.2 -158.3 -72.1 -165.6 19 G -72.3 -179.5 52.5 79.0 -153.2 -71.9 -169.1 20 C -64.4 178.2 50.5 78.2 -146.9 -73.6 -159.7 21 G -67.1 169.9 50.2 81.8 -148.3 -65.8 -164.9 22 C -60.0 176.0 52.8 84.1 -144.6 -61.6 -163.0 23 A -63.5 176.3 50.3 81.0 -152.1 -76.5 -164.6 24 G -54.3 170.7 53.6 81.0 -153.0 -69.0 -166.0 25 G --- 142.1 45.6 80.2 -147.5 -81.7 -172.6 26 U --- -156.2 59.0 85.8 --- --- -152.1 27 C --- 108.0 172.3 133.5 --- --- -123.6 28 C --- 162.2 52.6 83.3 --- --- -160.1 29 C 157.5 -150.9 163.6 85.5 --- --- -162.5 30 U -63.1 171.0 52.5 86.4 -168.3 -90.1 -156.2 31 G -56.1 171.2 54.6 85.2 -143.8 -72.2 -169.4 32 G -54.3 169.2 49.6 81.1 -156.8 -73.5 -169.4 33 G -63.1 171.0 47.3 77.2 -151.0 -77.4 -172.0 34 G -73.4 -177.5 49.0 83.5 -154.9 -71.6 -159.9 35 U -77.4 -171.2 52.1 82.4 -160.5 -60.5 -160.0 36 C -62.9 176.1 57.2 85.2 -161.1 -55.5 -168.4 37 C -64.0 174.5 51.3 81.7 -157.4 -67.1 -171.4 38 C -73.2 -177.2 53.8 83.0 -157.3 -74.5 -161.6 39 C -75.2 -174.5 54.3 83.3 -156.2 -73.4 -167.2 40 C --- -145.8 56.5 86.1 -160.2 -72.0 -172.6 41 A --- -96.4 -179.2 81.5 --- --- -148.8 42 G --- -149.3 -60.2 106.3 --- --- -112.0 43 C -63.4 167.4 52.5 85.8 --- --- -157.1 44 C -63.7 175.3 54.6 83.3 -145.3 -67.5 -162.4 45 C -70.8 175.4 58.7 84.5 -154.5 -70.6 -164.0 46 G -56.5 172.3 51.3 80.5 -157.4 -68.9 -166.0 47 G -59.6 172.5 45.4 78.3 -150.3 -73.9 -167.9 48 C -74.0 -177.1 53.2 81.2 -141.6 -81.6 -161.3 49 G -70.8 176.4 51.0 81.3 -153.6 -61.3 -161.9 50 C -58.3 175.2 53.0 83.5 -144.5 -58.8 -167.0 51 A -72.6 178.9 53.6 81.1 -150.8 -76.8 -169.1 52 G -53.4 170.5 53.2 80.8 -152.7 -70.8 -169.6 53 G --- 143.1 42.4 81.2 -148.7 -79.8 -172.0 54 U --- -173.4 44.3 84.2 --- --- -154.1 55 C --- 109.0 170.8 132.1 --- --- -126.9 56 C --- 162.8 52.7 83.0 --- --- -154.3
Эта форма тРНК больше всего по значению углов похожа на А-форму ДНК (даже очень похожа)
Торсионные углы DNA-protein complex 1I3J
Strand I base alpha beta gamma delta epsilon zeta chi 1 T --- 166.7 60.3 146.6 -171.5 -102.4 -105.7 2 C -50.8 -174.0 31.0 150.4 175.8 -114.2 -85.3 3 T -16.9 -174.6 13.5 150.0 -161.2 -114.5 -99.4 4 T 18.2 -162.5 -58.8 151.2 -159.9 -95.2 -95.8 5 G -60.2 175.4 34.8 153.6 -122.6 -179.0 -65.5 6 G -67.3 137.9 49.4 147.5 -149.2 -150.8 -89.9 7 G -71.5 157.8 56.9 147.8 -174.3 -92.2 -122.1 8 T -55.9 -159.4 25.3 144.7 161.1 -82.0 -95.7 9 C 151.3 150.4 -171.3 135.6 -154.7 -113.1 -129.7 10 T -38.9 -174.8 29.9 144.0 -170.3 -109.5 -105.1 11 A -48.8 -178.3 40.5 147.1 -166.1 -113.6 -93.0 12 C 22.8 -169.9 -55.1 153.1 -178.1 -115.7 -91.8 13 C 27.5 -161.8 -61.8 149.8 -159.9 -93.1 -124.5 14 G -55.8 -169.5 37.6 148.3 170.5 -108.5 -99.3 15 T -43.8 -171.3 39.0 147.8 -168.4 -120.7 -103.4 16 T 26.1 -168.3 -53.3 154.4 179.4 -89.1 -107.1 17 T -50.1 -171.0 29.7 156.8 -166.8 -154.0 -84.6 18 A -44.9 159.2 47.7 146.3 177.3 -90.7 -92.8 19 A -37.0 -155.4 25.0 151.9 -162.2 -112.0 -86.8 20 T 24.7 -126.9 -75.8 161.3 --- --- -119.5 Strand II base alpha beta gamma delta epsilon zeta chi 1 A 16.3 -176.4 -51.6 159.8 --- --- -94.6 2 G -52.4 164.8 49.0 151.0 -161.1 -112.2 -107.6 3 A -38.5 160.2 37.6 152.6 -163.9 -149.8 -85.8 4 A -48.7 157.8 48.3 145.1 -168.7 -123.3 -83.3 5 C -41.4 175.4 31.3 144.7 -156.6 -144.8 -86.6 6 C -27.9 -172.7 17.8 142.9 -164.0 -105.2 -105.6 7 C 41.2 174.7 -57.0 151.5 -176.1 -98.0 -112.2 8 A -35.2 167.6 23.9 147.1 -172.2 -120.7 -111.5 9 G -49.4 -177.4 29.3 152.5 -166.5 -150.0 -85.4 10 A -33.9 175.0 46.1 149.0 -175.2 -99.2 -96.8 11 T -48.9 -167.7 35.9 146.1 -171.5 -123.0 -99.1 12 G 32.9 -177.8 -52.9 151.4 165.5 -100.4 -125.1 13 G -42.4 -161.1 33.1 154.1 177.5 -130.9 -94.6 14 C 32.0 -161.0 -57.9 150.1 -178.5 -93.9 -100.8 15 A -21.7 162.3 31.9 147.7 -170.7 -111.2 -96.5 16 A -35.0 -173.9 24.6 151.0 170.0 -131.3 -84.8 17 A -36.7 174.5 42.1 147.1 175.8 -101.8 -85.5 18 T -31.1 178.3 26.2 144.4 -176.8 -119.8 -93.6 19 T 6.0 -151.1 -80.2 143.3 -167.5 -112.0 -108.4 20 A --- 177.9 47.4 155.3 -132.9 -137.0 -85.2
Excel-файл
Самым "деформированным" нуклеотидом можно считать девятый нуклеотид первой цепи (С)
Упражнение 2.
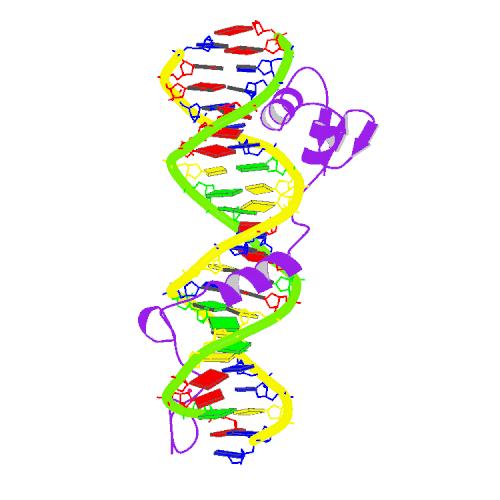
Для исследования была выбрана цепь C, представляющая глутамил-тРНК с последовательностью:
5'GGCCCCAUCGUCUAGCGGUUAGGACGCGGCCCUCUCAAGGCCGAAACGGGGGUUCGAUUCCCCCUGGGGU3'
На 3'-конце есть триплет GGU, к которому присоединяется аминокислота.
В выходном файле файле есть информация о водородные связях:
RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
1 (0.004) C:.501_:[..G]G-----C[..C]:.572_:C (0.005) |
2 (0.007) C:.502_:[..G]G-*---U[..U]:.571_:C (0.008) |
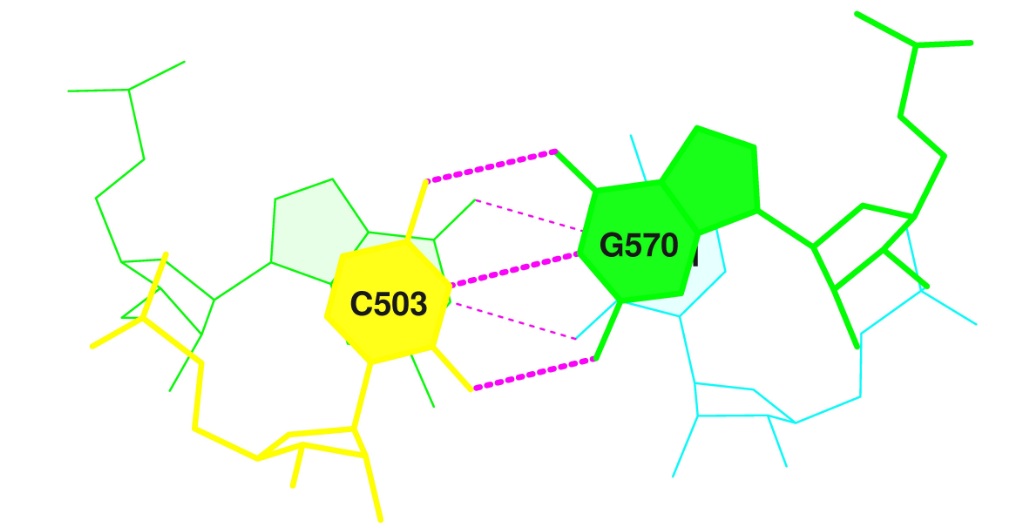
3 (0.003) C:.503_:[..C]C-----G[..G]:.570_:C (0.003) |
4 (0.003) C:.504_:[..C]C-----G[..G]:.569_:C (0.007) |
5 (0.005) C:.505_:[..C]C-----G[..G]:.568_:C (0.009) |
6 (0.005) C:.506_:[..C]C-----G[..G]:.567_:C (0.003) |
7 (0.004) C:.507_:[..A]Ax----U[..U]:.566_:C (0.006) |
8 (0.004) C:.549_:[..G]G-----C[..C]:.565_:C (0.002) |
9 (0.001) C:.550_:[..G]G-----C[..C]:.564_:C (0.003) |
10 (0.007) C:.551_:[..G]G-----C[..C]:.563_:C (0.002) |
11 (0.003) C:.552_:[..G]G-----C[..C]:.562_:C (0.003) |
12 (0.005) C:.553_:[..G]G----xC[..C]:.561_:C (0.004) |
13 (0.003) C:.554_:[..U]U-**-xA[..A]:.558_:C (0.008) |
14 (0.003) C:.555_:[..U]Ux**+xG[..G]:.518_:C (0.003) x
15 (0.003) C:.538_:[..A]A-*---C[..C]:.532_:C (0.005) |
16 (0.005) C:.539_:[..G]G-----C[..C]:.531_:C (0.004) |
17 (0.003) C:.540_:[..G]G-----C[..C]:.530_:C (0.004) |
18 (0.005) C:.541_:[..C]C-----G[..G]:.529_:C (0.005) |
19 (0.004) C:.542_:[..C]C-----G[..G]:.528_:C (0.005) |
20 (0.006) C:.543_:[..G]G-----C[..C]:.527_:C (0.003) |
21 (0.005) C:.544_:[..A]Ax*---G[..G]:.526_:C (0.004) |
22 (0.006) C:.510_:[..G]G-----C[..C]:.525_:C (0.004) |
23 (0.004) C:.511_:[..U]U-----A[..A]:.524_:C (0.003) |
24 (0.002) C:.512_:[..C]C-----G[..G]:.523_:C (0.011) |
25 (0.004) C:.513_:[..U]U-*--xG[..G]:.522_:C (0.007) |
26 (0.007) C:.514_:[..A]A-**-xU[..U]:.508_:C (0.004) |
27 (0.018) C:.515_:[..G]Gx**+xC[..C]:.548_:C (0.012) x
28 (0.004) C:.519_:[..G]Gx---xC[..C]:.556_:C (0.002) +
Note: This structure contains 16[8] non-Watson-Crick base-pairs.
Стебли
В соответствии с полученными данными можно определить:
- Акцепторный стебель состоит из участка 1-6 и комплементарного ему участка 67-72
Т-стебель – 49-53 и комплементарного ему участка 61-65
D-стебель – 10-13 и комплементарного ему участка 22-25
Антикодоновый стебель – 26-32 и комплементарного ему участка 38-44
Водородные связи у неканонических пар
Комплементарные пары, не имеющие отношение к стеблям и стабилизирующие третичную структуру тРНК:
13 (0.003) C:.554_:[..U]U-**-xA[..A]:.558_:C (0.008) | 14 (0.003) C:.555_:[..U]Ux**+xG[..G]:.518_:C (0.003) x 27 (0.018) C:.515_:[..G]Gx**+xC[..C]:.548_:C (0.012) x 28 (0.004) C:.519_:[..G]Gx---xC[..C]:.556_:C (0.002) +
взаимодействия между 54 и 58 - взаимодействия в Т-петле
водородные связи между 55 и 18, 19 и 56 - дополнительные связи между D- и Т-петелями, стабилизирующие третичную структуру тРНК, при этом пара 55-18 - неканоническая, 19-56 - каноническая.
Упражнение 3.
Нахождение стекинг-взаимодействий
В файле находим информацию о перекрывании азотистых оснований:
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/UC 1.21( 0.13) 0.00( 0.00) 0.00( 0.00) 0.89( 0.00) 2.10( 0.13)
2 GC/GU 7.23( 4.42) 0.00( 0.00) 0.00( 0.00) 6.56( 3.95) 13.79( 8.36)
3 CC/GG 0.10( 0.00) 0.00( 0.00) 0.37( 0.00) 3.01( 1.55) 3.47( 1.55)
4 CC/GG 0.00( 0.00) 0.00( 0.00) 0.27( 0.00) 3.72( 2.31) 3.98( 2.31)
5 CC/GG 0.46( 0.10) 0.00( 0.00) 0.24( 0.00) 2.81( 1.37) 3.52( 1.47)
6 CA/UG 0.57( 0.00) 0.00( 0.00) 1.85( 0.72) 0.36( 0.00) 2.77( 0.72)
7 AG/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.56( 0.89) 2.56( 0.89)
8 GG/CC 2.15( 0.70) 0.00( 0.00) 0.37( 0.00) 0.38( 0.11) 2.90( 0.82)
9 GG/CC 3.75( 2.42) 0.00( 0.00) 0.17( 0.00) 0.38( 0.00) 4.30( 2.42)
10 GG/CC 3.99( 2.66) 0.00( 0.00) 0.47( 0.00) 0.02( 0.00) 4.48( 2.66)
11 GG/CC 4.12( 2.56) 0.00( 0.00) 0.59( 0.00) 0.00( 0.00) 4.71( 2.56)
12 GU/AC 6.51( 3.65) 0.00( 0.00) 0.00( 0.00) 1.58( 0.17) 8.09( 3.82)
13 UU/GA 5.21( 2.66) 0.00( 0.00) 0.00( 0.00) 5.41( 2.58) 10.61( 5.25)
14 UA/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 AG/CC 4.30( 2.39) 0.00( 0.00) 0.00( 0.00) 5.43( 2.51) 9.72( 4.90)
16 GG/CC 2.49( 1.03) 0.00( 0.00) 0.55( 0.00) 0.42( 0.00) 3.47( 1.03)
17 GC/GC 6.65( 3.52) 0.00( 0.00) 0.00( 0.00) 6.36( 3.25) 13.01( 6.77)
18 CC/GG 0.15( 0.00) 0.00( 0.00) 0.83( 0.00) 3.03( 1.51) 4.01( 1.52)
19 CG/CG 0.00( 0.00) 0.00( 0.00) 5.17( 2.44) 0.00( 0.00) 5.17( 2.44)
20 GA/GC 1.65( 0.00) 0.00( 0.00) 0.00( 0.00) 5.14( 2.36) 6.79( 2.36)
21 AG/CG 0.00( 0.00) 0.00( 0.00) 0.75( 0.00) 1.72( 1.04) 2.46( 1.04)
22 GU/AC 4.94( 2.21) 0.00( 0.00) 0.00( 0.00) 3.50( 2.19) 8.44( 4.40)
23 UC/GA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 4.00( 2.53) 4.00( 2.53)
24 CU/GG 1.68( 0.17) 0.00( 0.00) 0.00( 0.00) 2.66( 1.14) 4.34( 1.31)
25 UA/UG 0.00( 0.00) 2.32( 0.09) 5.83( 4.42) 0.00( 0.00) 8.14( 4.51)
26 AG/CU 3.68( 1.43) 0.00( 0.00) 0.01( 0.00) 0.00( 0.00) 3.70( 1.43)
27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
28 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
29 GG/UC 1.18( 0.11) 0.00( 0.00) 0.00( 0.00) 1.35( 0.04) 2.52( 0.14)
30 GC/GU 6.96( 4.23) 0.00( 0.00) 0.00( 0.00) 6.25( 3.65) 13.21( 7.87)
31 CC/GG 0.38( 0.01) 0.00( 0.00) 0.33( 0.00) 3.05( 1.62) 3.77( 1.63)
32 CC/GG 0.00( 0.00) 0.00( 0.00) 0.40( 0.00) 3.60( 2.17) 4.00( 2.17)
33 CC/GG 0.70( 0.15) 0.00( 0.00) 0.14( 0.00) 3.18( 1.77) 4.02( 1.92)
34 CA/UG 0.44( 0.00) 0.00( 0.00) 1.57( 0.54) 0.69( 0.04) 2.69( 0.58)
35 AG/CU 0.00( 0.00) 0.00( 0.00) 0.47( 0.00) 0.79( 0.19) 1.26( 0.19)
36 GG/CC 2.45( 0.94) 0.00( 0.00) 0.69( 0.00) 0.04( 0.00) 3.18( 0.95)
37 GG/CC 4.14( 2.78) 0.00( 0.00) 0.21( 0.00) 0.00( 0.00) 4.34( 2.78)
38 GG/CC 3.75( 2.42) 0.00( 0.00) 0.26( 0.00) 0.00( 0.00) 4.00( 2.42)
39 GG/CC 3.77( 1.93) 0.00( 0.00) 0.95( 0.00) 0.00( 0.00) 4.72( 1.93)
40 GU/AC 6.53( 3.72) 0.00( 0.00) 0.00( 0.00) 1.49( 0.12) 8.02( 3.84)
41 UU/GA 5.30( 2.78) 0.00( 0.00) 0.00( 0.00) 5.02( 2.27) 10.32( 5.05)
42 UA/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
43 AG/CC 5.03( 2.49) 0.00( 0.00) 0.00( 0.00) 5.98( 2.59) 11.00( 5.09)
44 GG/CC 2.90( 1.44) 0.00( 0.00) 0.58( 0.00) 0.12( 0.00) 3.60( 1.44)
45 GC/GC 6.11( 3.06) 0.00( 0.00) 0.00( 0.00) 6.59( 3.46) 12.70( 6.52)
46 CC/GG 0.21( 0.01) 0.00( 0.00) 0.82( 0.00) 3.02( 1.52) 4.06( 1.53)
47 CG/CG 0.00( 0.00) 0.00( 0.00) 5.06( 2.36) 0.00( 0.00) 5.06( 2.36)
48 GA/GC 2.07( 0.11) 0.00( 0.00) 0.00( 0.00) 5.49( 2.61) 7.56( 2.72)
49 AG/CG 0.00( 0.00) 0.00( 0.00) 0.75( 0.00) 1.06( 0.59) 1.82( 0.59)
50 GU/AC 5.23( 2.50) 0.00( 0.00) 0.00( 0.00) 3.40( 2.25) 8.64( 4.75)
51 UC/GA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 3.50( 2.02) 3.50( 2.02)
52 CU/GG 1.37( 0.09) 0.00( 0.00) 0.00( 0.00) 2.67( 1.15) 4.04( 1.24)
53 UA/UG 0.00( 0.00) 2.39( 0.12) 5.97( 4.52) 0.00( 0.00) 8.37( 4.64)
54 AG/CU 3.93( 1.55) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 3.93( 1.55)
55 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Выбираем пару с наибольшим перекрыванием, например
30 GC/GU 6.96( 4.23) 0.00( 0.00) 0.00( 0.00) 6.25( 3.65) 13.21( 7.87)
Далее находим данные о таком взаимодействии в файле stacking.pdb. Вырезаем эту структуру в отдельный файл:
ex_str -30 stacking.pdb step30.pdb
Строим изображение:
stack2img -cdolt step30.pdb step30.ps
Если воспользоваться командой:
pdb2img -rc step7.pdb stdout | render -jpeg > step7.jpg
то получим это изображение в формате .jpg