FT CDS 163..435 FT /product="RSS" FT /note="Rat Stomach Serotonin receptor-related gene" FT /db_xref="GOA:P0A7B8" FT /db_xref="InterPro:IPR001353" FT /db_xref="InterPro:IPR022281" FT /db_xref="PDB:1E94" FT /db_xref="PDB:1G4A" FT /db_xref="PDB:1G4B" FT /db_xref="PDB:1HQY" FT /db_xref="PDB:1HT1" FT /db_xref="PDB:1HT2" FT /db_xref="PDB:1NED" FT /db_xref="UniProtKB/Swiss-Prot:P0A7B8" FT /protein_id="BAA14040.1" FT /translation="MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHY FT GIAQRGLTITSDDHMAVTAYAYYSCHELTPWLRIQSTNPVQKYGA" >D89965_5 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds. MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM AVTAYAYYSCHELTPWLRIQSTNPVQKYGAДанная запись EMBL ссылается на запись P0A7B8 в UniProtKB/Swiss-Prot:
FT /db_xref="UniProtKB/Swiss-Prot:P0A7B8"В этой записи (последовательность которой принадлежит кишечной палочке!) мы находим частичное соответствие девятой рамки считывания: она в точности соответствует 28-125 остаткам (их всего 176).
>sp|P0A7B8|HSLV_ECOLI ATP-dependent protease subunit HslV; MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGGTADAFTL FELFERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVADETASLIITGNGDVVQPE NDLIAIGSGGPYAQAAARALLENTELSAREIAEKALDIAGDICIYTNHFHTIEELSYKA >D89965_9 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGSСоответствие начинается с метионина (что подходит, как старт-кодон), но внезапно обрывается. Так как наши рамки считывания были найдены для крысы (!), а ему соответвует отрывок из генома кишечной палочки. Возможно, изначально предполагалось, что последовательность D89965 принадлежит крысе, но позже выяснилось, что произошла контаминация геномом палочки (ошибка эксперимента). Следовательно, становится понятно, почему у нас так странно соотносятся рамки считывания.
Задание 2. Поиск гомологов некодирующих последовательностей с помощью blastn
Запустим blastn с нужными параметрами на последовательностях тРНК:
blastn -query trna_bacsu.fasta -task blastn -db sa -out satrna.txt -outfmt 7 -evalue 0.01
получим результат.
Далее сначала вытащим список последовательностей:
grep -i '# Q' blastn_0.txt | sed -e 's/# Query: //' -e "s/ .*//" > list.txt
Потом для каждой найдем количество вхождений:
while read s; do grep -i $s blastn_0.txt | grep -v '# Query' | wc -l >> list0.txt; done < list.txt
Задание 3. Поиск гомологов при измененных параметрах blastn Прогоним BLAST с разными параметрами.
blastn_1.txt — -reward 5 -penalty -4 -gapopen 8 -gapextend 6
blastn_2.txt — -reward 5 -penalty -4 -gapopen 8 -gapextend 6 -word_size 4
blastn_3.txt — -word_size 4
Получим аналогичным образом файлы list1, list2, list3, подклеим их к таблице: окончательный вариант.
Анализ результатов
При уменьшении параметра word_size программе нужно обработать больше выравниваний,следовательно, процесс замедляется.
Изменение word_size на минимальное значени - 4 приводит к большему количеству выравниваний
При уменьшении значения параметра word_size программе приходится прорабатывать больше потенциальных выравниваний и процесс значительно замедляется.
Для анализа была выбрана пара BSn5_t20982 и embl|AL766855|AL766855. Она есть при выравнивании с параметрами №2, но ее нет при параметрах №3. Это связано с ослаблением параметров, связанных со штрафами.
Командой seqret -sask вырезала гомологичный участок. Ниже приведен результат команды needle.
# Aligned_sequences: 2
# 1: BSn5_t20982
# 2: AL766855
# Matrix: EDNAFULL
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 83
# Identity: 49/83 (59.0%)
# Similarity: 49/83 (59.0%)
# Gaps: 21/83 (25.3%)
# Score: 164.0
#
#
#=======================================
BSn5_t20982 1 gcttccatagctcagcaggtagagcact----------tccatggtaagg 40
||.|||.||||||||..|||||||||.| ||.|||
AL766855 1 gcctccttagctcagttggtagagcagtagactcttaatctatg------ 44
BSn5_t20982 41 aagaggtcagcggttcgagcccgcttggaagct 73
|||||..|||||||||||..|.||..||
AL766855 45 ----ggtcacaggttcgagccctgtagggggc- 72
В целом, выравнивание напоминает выравнивание тРНК-последовательностей. Проверим с помощью mfold.
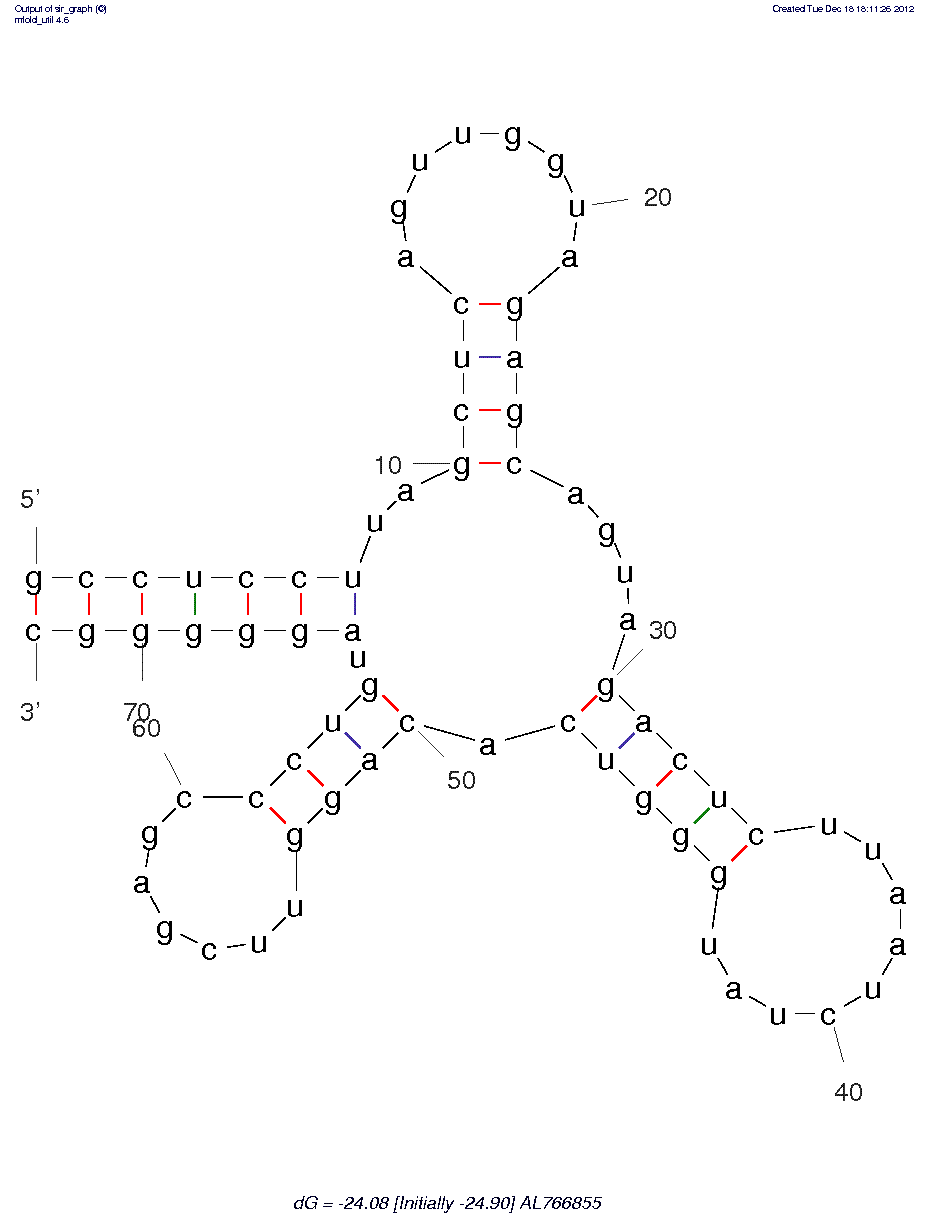
В поле FT EMBL-файла бактерии гомологичный учаток проаннотирован как т-РНК.
FT tRNA 17457..17529 FT /product="transfert RNA-Lys" FT /note="tRNAscan-SE vs 1.3 result - Cove score = 83.56"