0.Описание трансмембранных белков с известной 3D структурой
Для трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков были определены параметры,указанные ниже
| PDB код | Тип | Какая мембрана | Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 1zcd | Спираль | Внутренняя мембрана E.coli | 28.4 ± 1.4 A | 21 |
| 3lbw | Спираль | Мембрана Influenza virus | 31.1 ± 1.7 A | 19 |
| 3kp9 | Спираль | Внутренняя мембрана Synechococcus sp. | 29.8 ± 1.6 A | 20 |
| 2fgg | Баррель | Внешняя мембрана Comamonas acidovorans | 25.0 ± 0.9 A | 8 |
| 1af6 | Баррель | Внешняя мембрана E.coli | 25.1 ± 0.7 A | 9 |
| 3qlb | Баррель | Внешняя мембрана Pseudomonas fluorescens | 23.4 ± 0.8 A | 7 |
1. Отбор гомологов белка ASIC1_CHICK
Этот белок - Acid-sensing ion channel(ASIC); принадлежит организму Gallus gallus (Chicken - курица). Этот канал понтециал-независимый, активируемый протонами рецептор, который принадлежит к семейству эпителиальных натриевых каналов и вовлечен в процессы восприятия боли, ишемического инсульта, механосенситизации, обучения и памяти.
fasta-последовательность белка (только цепь А, вообще этот белок имеет 6 цепей)
Был произведен поиск гомологов белка с помощью PSI-BLAST (как в подсказках). При поиске гомологов были исключены белки из типа Chordata
После 4 итераций PSI-BLAST (порог на E-value 1e-10, максимальное количество хитов 1000) было отобрано 11 хитов из (по возможности) разных таксономических групп.
В группах отбирались белки по наибольшему Query cover. К сожалению, очень большого таксономического разнообразия отобрать не удалось, так как из многих организмов были предсказанные белки, которые я решила не брать. В основном белки брались из дрозофил, комаров и червей.
Затем названия последовательностей были отредактированы с сохранением указания на сам белок и соответствующий ему организм.
seq_hom.fasta
2. Анализ структуры белка ASIC1_CHICK
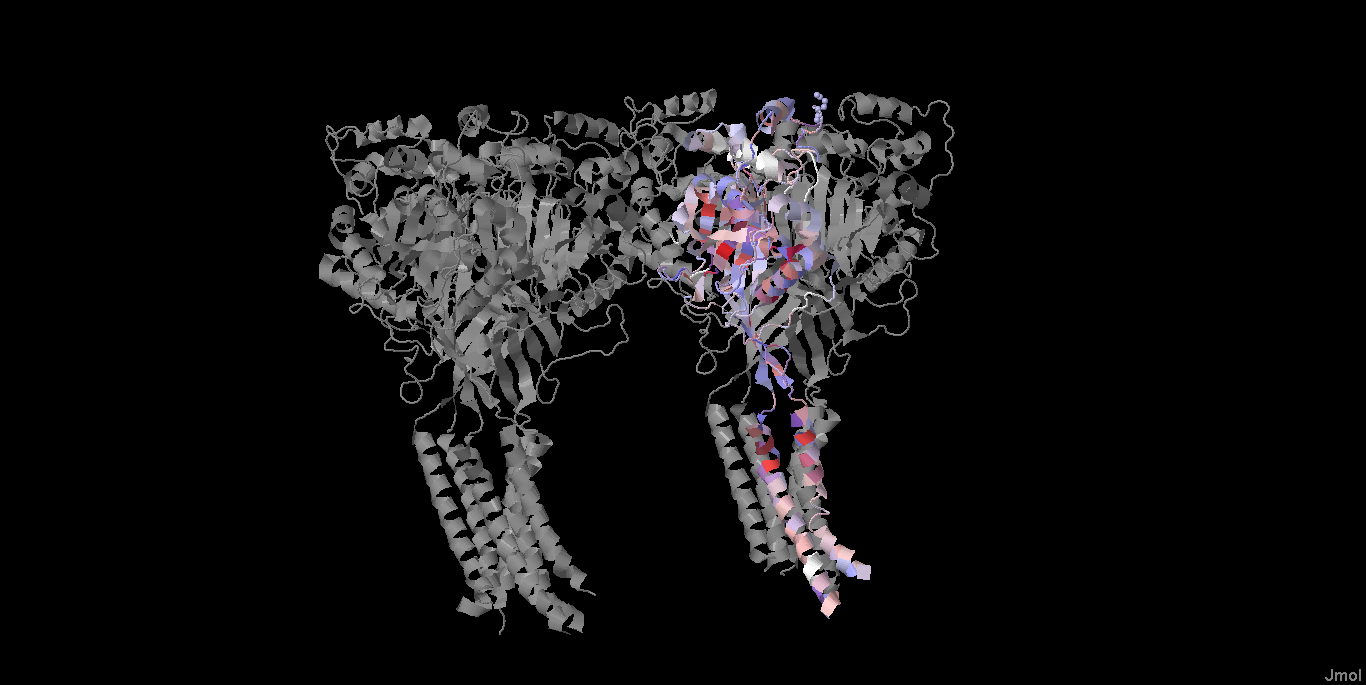
В базе данных OPM находим выданный белок 1QTS.
В базе данный TCBD структура этого белка, к сожалению, отсутствует. Зато, пройдя по ссылке из OPM, можно прочитать информацию про каналы на сайте TCBD: 1.A.6 The Epithelial Na+ Channel (ENaC) Family. Если искать в базе по названию в примерах белков, то наш белок также не находится.
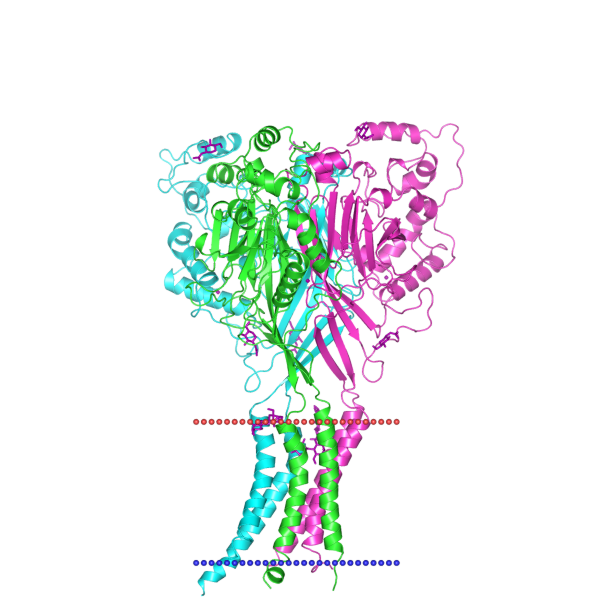
| PDB ID | Организм | Тип мембраны | TC-код | Наклон спиралей (бета-тяжей) к нормали | Количество трансмембранных втоичных структур | Название белка |
| 1QTS | Gallus gallus (курица) | Цитоплазматическая мембрана эукариот | 1.A.6.* | 15 ± 0 | 2 в цепи А | Acid-sensing ion channel |
3. Анализ множественного выравнивания трансмембранных белков
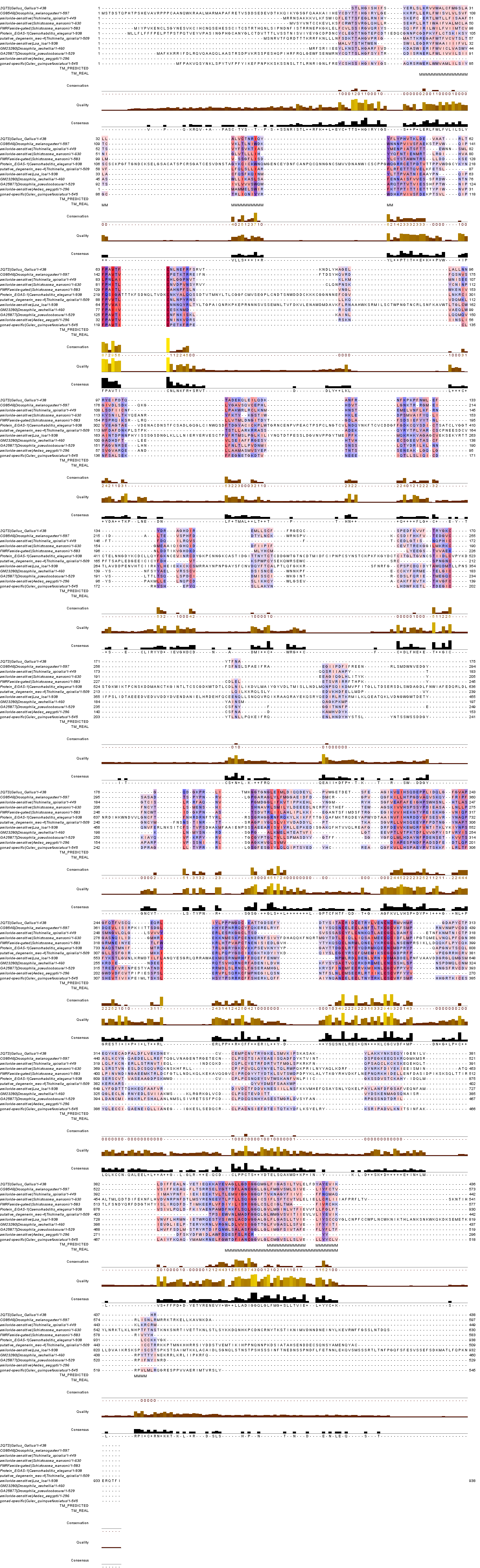
Построим множественное выравнивание отобранных гомологов с помощью Muscle (align.fasta):
muscle -in seqs.fasta -out align.fasta
Загрузим полученное множественное выравнивание в программу JalView. К выравниванию добавим аннотации TM_REAL и TM_PREDICTED. TM_REAL содержит указания на участки последовательности, отвечающие трансмембранным спиралям в данном белке (1QTS:A), которая была прикреплена к его последовательности ASIC1_CHICK в выравнивании в программе JalView. TM_PREDICTED содержит предсказанные с помощью TMHMM участки трансмембранных спиралей в гомологе из Drosophila melanogaster.
В поле TM_REAL получилось 2 спирали (mem.jar). В базе данных OPM также указывалось существование двух трансмембранных спиралей.
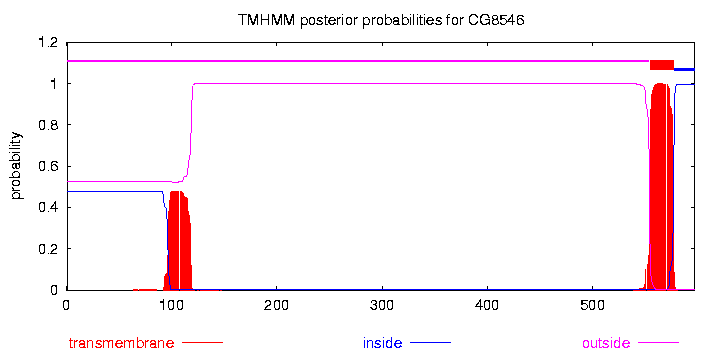
Предсказание с помощью TMHMM:
# CG8546 Length: 597 # CG8546 Number of predicted TMHs: 1 # CG8546 Exp number of AAs in TMHs: 33.26184 # CG8546 Exp number, first 60 AAs: 0.00021 # CG8546 Total prob of N-in: 0.47513 CG8546 TMHMM2.0 outside 1 554 CG8546 TMHMM2.0 TMhelix 555 577 CG8546 TMHMM2.0 inside 578 597
В JalView покрасили выравнивание по гидрофобности, чтобы различать гидрофобные и гидрофильные остатки (Color -> Hydrophobicity), консервативность - 15% (Color -> By Conservation 15%)
Основная часть белка (все, кроме трансмембранных спиралей) ориентирована в межклеточное пространство.
Участки, относящиеся к TM спиралям, в основном консервативны. Также консервативны участки между этими спиралями.
В ТМ, на мой взгляд, наиболее часто встречаются остатки валина, аланина, лейцина, изолейцина, глицина, фенилаланина - по большей части гидрофобные остатки. Менее часто встречаются консервативные позиции полярных остатков - тирозин, аргинин. Возможно, эти остатки имеют какое-то важное функциональное значение.
Результат программы TMHMM а реальная структура белка не очень совпадают (предсказанная спираль целиком входит состав реальной).
Может, это из-за того, что выбран неподходящий гомолог для сравнения.
Возможно, такое предсказание нельзя считать полностью неверным, так как оно попадает на достаточно консервативные участки последовательностей и входит в состав реальной спирали.
Но помимо этого есть спираль, полностью проигнорированная предсказанием. Это, как мне кажется, должно быть странным.
Но, возможно, это объясняется тем, что в теx участках спиралей, которые не были предсказаны, есть консервативные полярные остатки, которые не дают свернуть спираль (а в самой мембране они, возможно, чем-нибудь стабилизируются).