Задание 1
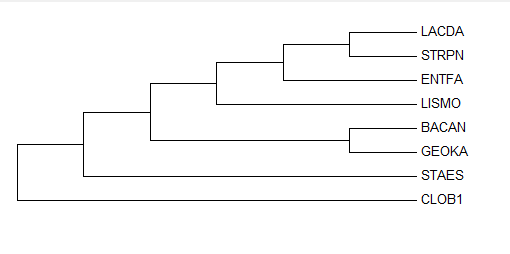
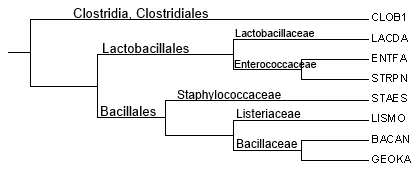
Пользуясь таксономическим сервисом NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/, определяем, к каким таксонам относятся отобранные в предыдущем задании бактерии.Все отобранные бактерии относятся к Cellular organisms, Bacteria и Firmicutes (грамположительные). Дальнейшие различия в таксономии представлены в таблице.
| Таксоны | Название | Мнемоника |
| Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group | Bacillus anthracis | BACAN |
| Clostridia; Clostridiales; Clostridiaceae; Clostridium | Clostridium botulinum | CLOB1 |
| Bacilli; Lactobacillales; Enterococcaceae; Enterococcus | Enterococcus faecalis | ENTFA |
| Bacilli; Bacillales; Bacillaceae; Geobacillus | Geobacillus kaustophilus | GEOKA |
| Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus | Lactobacillus delbrueckii | LACDA |
| Bacilli; Bacillales; Listeriaceae; Listeria | Listeria monocytogenes | LISMO |
| Bacilli; Bacillales; Staphylococcaceae; Staphylococcus | Staphylococcus epidermidis | STAES |
| Bacilli; Lactobacillales; Streptococcaceae; Streptococcus | Streptococcus pneumoniae | STRPN |
Определим на изображении филогенетического дерева, полученного в предыдущем задании, ветви, выделяющие какие-нибудь таксоны
Задание 2
Из Swiss-Prot получила последовательности белков - факторов высвобождение пептидной цепи 1(RF1) для выбранных бактерий.seqret sw:rf1_bacan
Поместила последовательности в один fasta-файл rf1_bacteria.fasta отредактировав названия последовательностей, оставив только мнемонику видов.
Далее выровняем последовательности с помощью JalView (импортировала выбранные невыровненные последовательности, воспользовалась Web Service, Muscle with Defaults)
Ниже представлено изображение выравнивания в блочной (Wrap) форме, раскраска по проценту идентичности

выравнивание в fasta-формате
JalView проект
Задание 4
Деревья были реконструированы четырьмя методами, доступными из JalView (меню Calculate > Calculate tree)Каждое дерево сохранено в Newick-формате в файл с соответствующим названием. Изображения получены с помощью программы Mega.
| Average distance tree using PID | 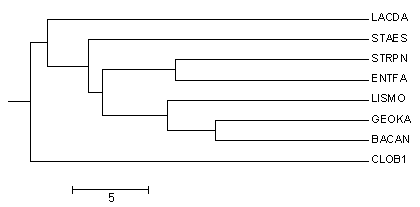 |
Правильно выделена ветвь "Clastridiales". Отсутствует правильное разделение ветвей "Bacillales" и "Lactobacillales" | Верные нетривиальные ветви (есть в правильном дереве):
(LISMO, BACAN, GEOKA) против (STAES, STRPN, ENIFA, LACDA, CLOB1) (STRPN, ENIFA) против (STAES, LASMO, BACAN, GEOKA, LACDA, CLOB1) Есть неверная нетривиальная ветвьЖ (LISMO, BACAN, GEOKA, STRPN, ENTFA) против (STAES, LACDA, CLOB1) |
| Neigbour joining tree using PID | 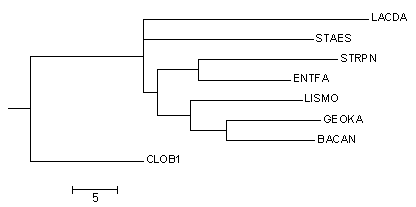 |
Отличается от 1 дерева тем, что от узла отходят три ветви после отделения CLOB1 от остального дерева | Верные и неверные ветви те же, что и в 1 |
| Average distance tree using BLOSUM62 | 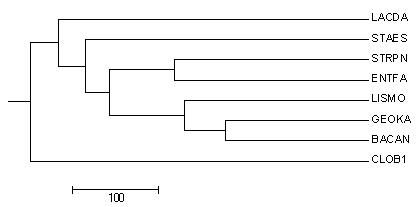 |
Абсолютно не отличается от дерева 1 | |
| Neigbour joining tree using BLOSUM62 |  |
От дерева 1 отличается топологией листьев STAES и LACDA (в первом дереве наоборот) | Верные и неверные ветви те же, что и в 1 |
Судя по всем этим деревьям, CLOB1 действительно далеко стоит от остальным организмам по белку RF1 (все деревья выделяют его в отдельную ветвь). Но STAES и LACDA находятся не на своем месте, по сравнению с топологией в правильном дереве, что говорит о сильных различиях по этому белку с кладой LISMO, BACAN, GEOKA, STRPN, ENTFA). Все деревья очень похожи.
Задание 5
Импортировано выравнивание в программу Mega (при импорте выбран "Analyze"). Реконструировано дерево методом "Maximum Parsimony" (кнопка Phylogeny). Укоренено дерево в наиболее правильную, с моей точки зрения, ветвь (меню Subtree > Root).