Выбор структуры белка
Для дальнейшей работы был выбран белок spsE из работы в первом семестре. Этот белок соответствует всем необходимым критериям, а именно:
Сервис EDS знает PDB код белка, что равносильно тому, что для данного белка доступны экспериментальные данные, а не только модель
Доступен файл со структурными факторами
Существуют подходящие структурные гомологи белка (Проверка PDBeFold => launch => Query - PDB код, в таблице с находками должно быть 5 или более разных белков с RMSD (оценка сходства) от 0,8 до 2,5 ангстрем и N_align(число сопоставленных C_alpha) от 50% до 90%)
Структурные факторы
Из PDB была загружена модель структуры
и
файл со структурными факторами
Каждый структурный фактор расположен в отдельной строке и представлен последовательностью чисел. Прямого соответствия атомам модели нет.
Число атомов можно определить по файлу .pdb (2739), число отражений - по файлу структурных факторов (520 245).
Для каждого структурного фактора присутствуют следующие поля:
Crystal_ID lambda_ID Scale_Group_ID h k l Status F_mean F_sigma F_calc phase_calc 1 1 1 0 0 9 o 827.1 16.8 1043.6 0.0 1 1 1 0 0 12 o 854.0 17.4 4247.5 0.0 1 1 1 0 0 15 o 227.4 4.9 345.1 180.0
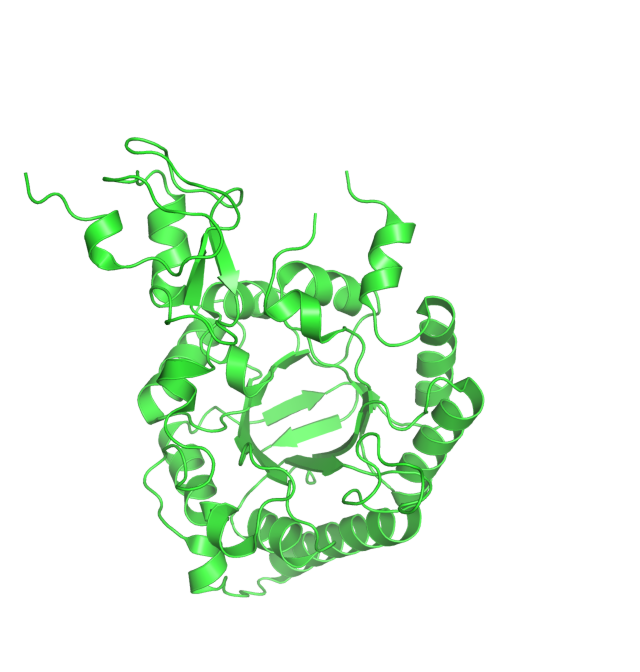
Изображение электронной плотности
Файл с электронной плотностью был скачан с сайта PDB в виде карты формата O и 2mFo-DFc (по умолчанию).
Нужно было оценить качество модели пространственной структуры, представленной в PDB файле.
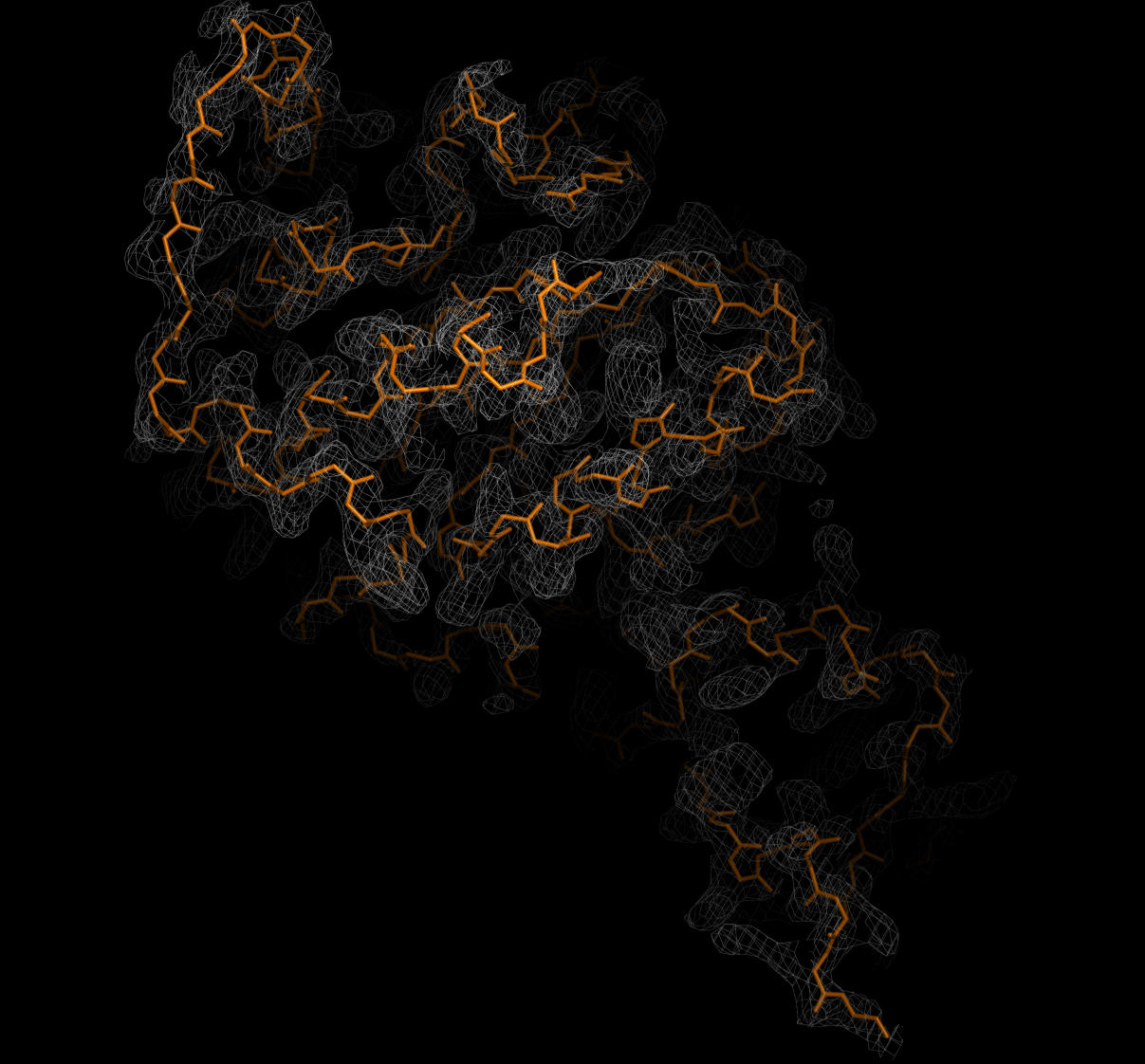
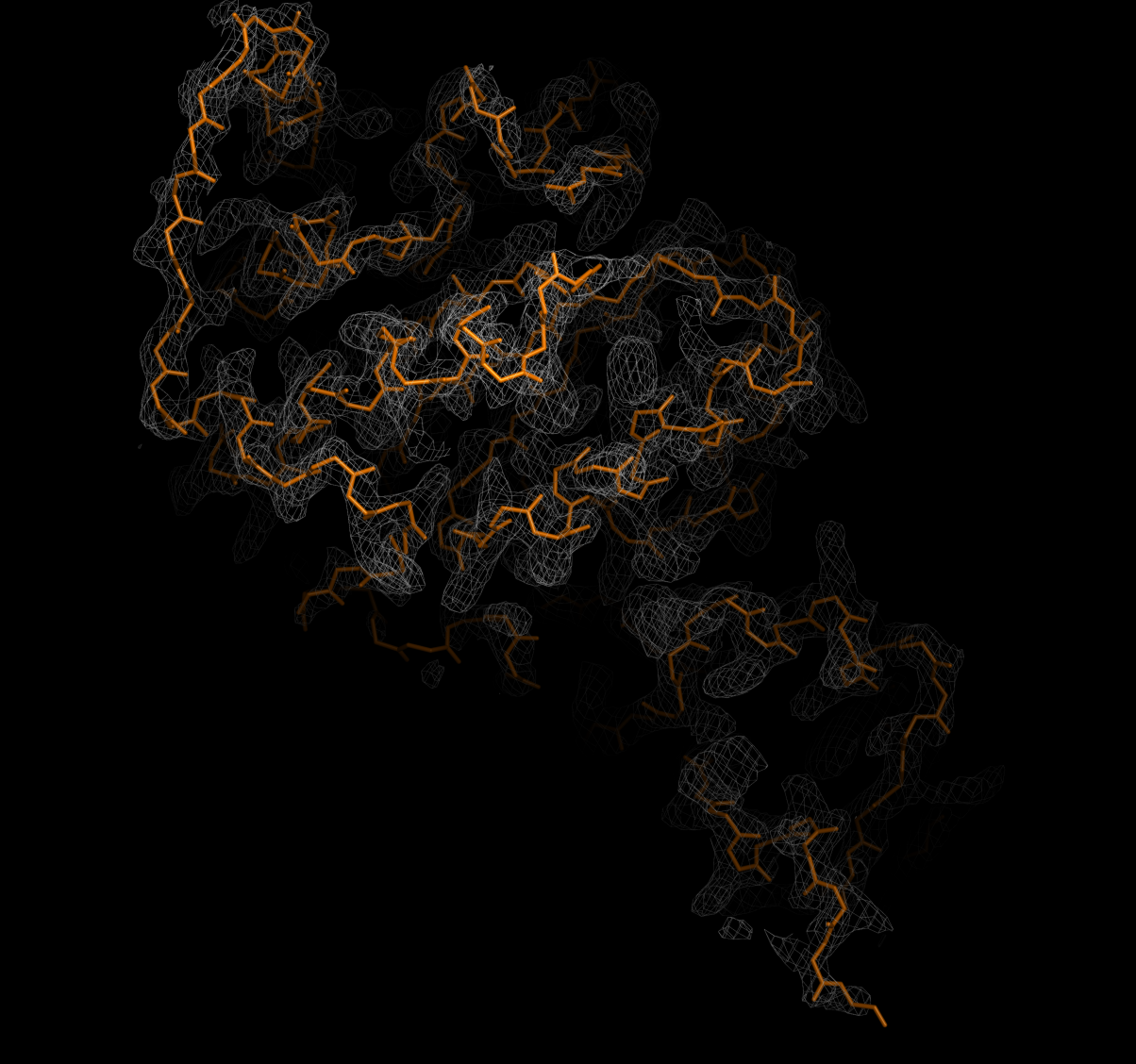
Для начала надо было оценить, насколько хорошо электронная плотность описывает полипептидный остов цепи.
 |
 |
 |
| |
|
|
Видно, что ЭП хорошо соотносится с полипептидным остовом на всех уровнях подрезки.
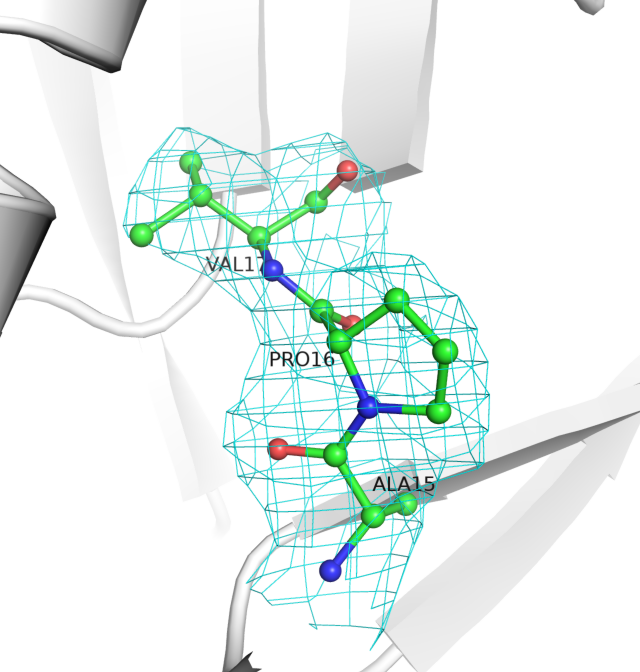
Далее нужно было оценить, насколько хорошо ЭП соотносится с отдельными остатками.
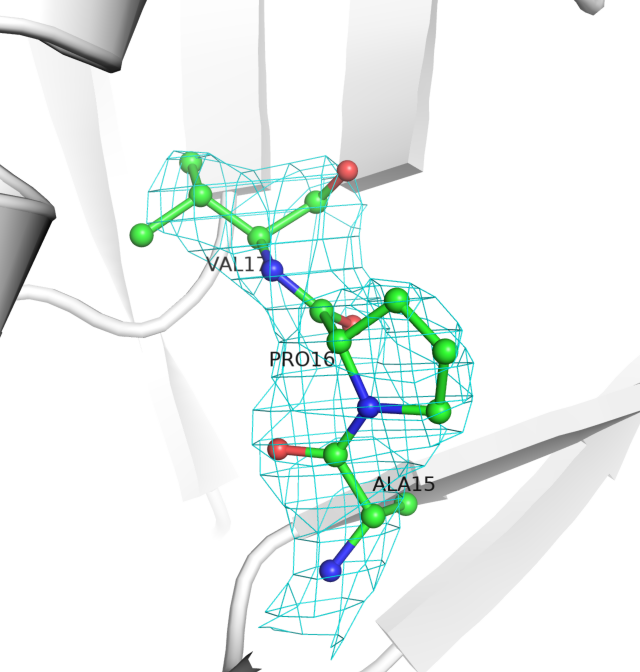
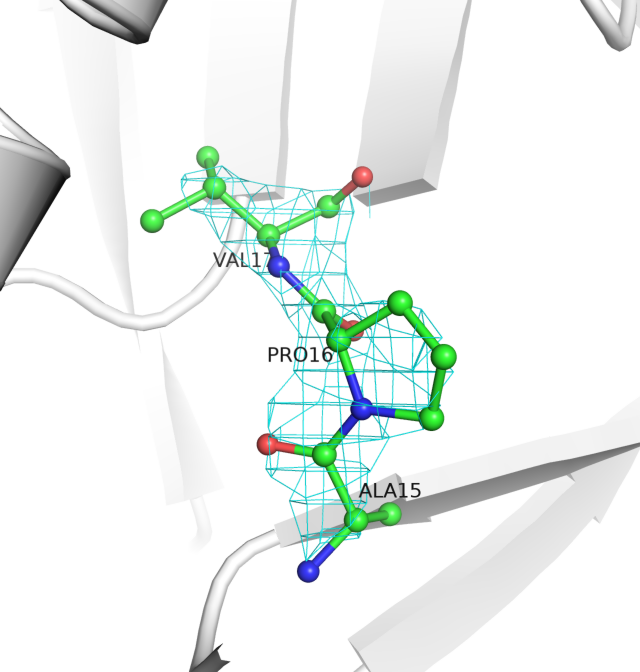
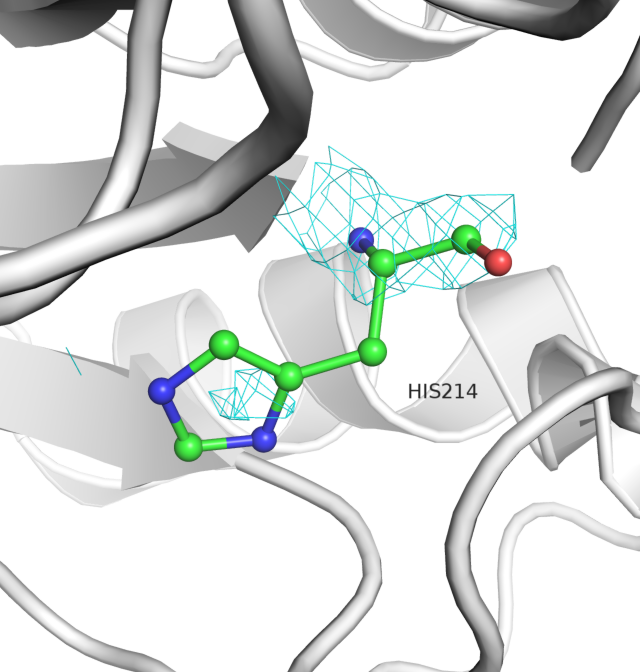
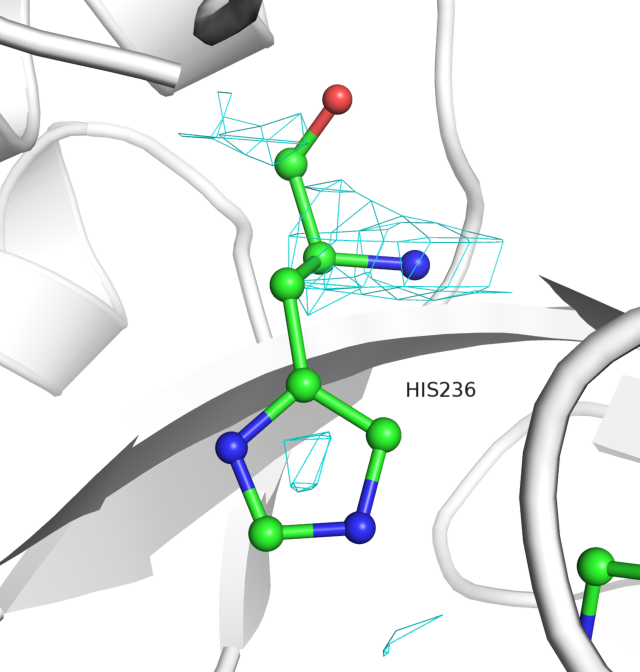
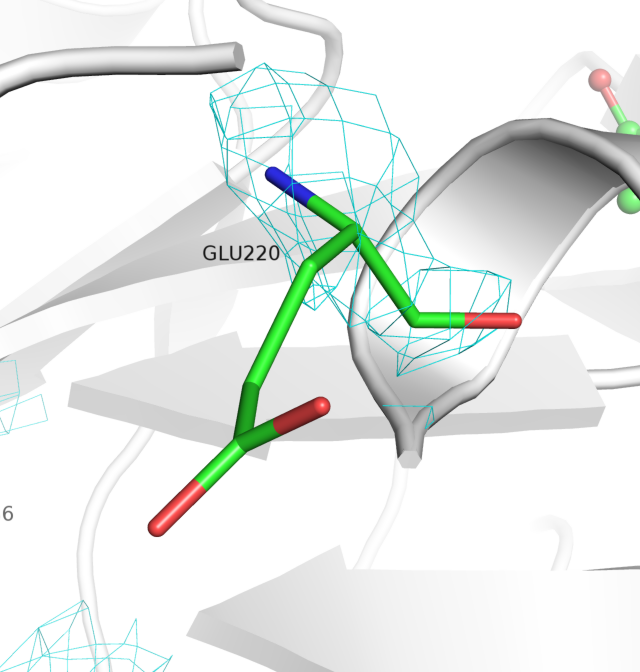
Для этого были построены изображения электронной плотности для 3 уровней электронной плотности вокруг трех аминокислот (15-17) на поверхности белка и аминокислот, которые принимают участие в связывании атома цинка, который необходим для катализа (214, 220, 236).
 |
 |
 |
| |
|
|
 |
 |
 |
| |
|
|
Вообще, разрешение у данного белка 2.38 ангстрем, что является приемлимым разрешением для белков. В примере с тремя аминокислотами 15-17 атомы достаточно хорошо вписываются в электронную плотность. При этом у аминокислотных остатков, которые взаимодействуют с ионом цинка в активном центре металла, с 3 уровнем подрезки электронной плотности дело обстоит хуже. Видно, что она практически полностью обходит стороной боковые радикалы, хотя в глутаминовой кислоте есть два электроотрицательных атома кислорода. А гистидинах плотность присуствует в середине кольца, что логично, поскольку заряд "размазан" по кольцу. Общий вывод - координаты атомов в принципе соответствуют "сгусткам" электронной плотности (особенно на небольших уровнях подрезки), и бОльшее скопление соответствует атому кислорода в основной цепи (видно по последним трем рисункам).