Работа с выравниваниями
1. Нахождение отличий между выравниваниями
Были проведены выравнивания белков арил-алколь дегидрогеназы из Pseudomonas putida и алкоголь дегидрогеназы из Homo sapiens при помощи needle со стандартными опциями и BLAST с ожидаемым порогом E-value не более 0.01 и другими настройками по умолчанию.
Ниже представлен отрезок выравнивания в BLAST, на котором аминокислотные остатки 290-300 белка Pseudomonas putida сопоставляются с отсатками 298-308 белка Homo sapiens.
GEEIATRPFQL G + L GATVPVDINFL
А тут - отрезок выравнивания в needle, на котором аминокислотные остатки 290-300 белка Pseudomonas putida сопоставляются с отсатками 298-308 белка Homo sapiens.
GEEIATRPFQ---L | ||.|.. | G---ATVPVDINFL
Видно, что в первом случае остаткам E299, E300, I301 Homo sapiens сопоставлены остатки A291, T292, V293 соотвнтственно, а во втором случае - только индель.
Также можно заметить, что в первом случае остаткам I297, N298, F299 белка бактерии сопоставлены остатки P305, F306, Q307 соотвнтственно, а во втором случае - снова индель.
Такое различие можно объяснить тем, что в процессе работы BLAST разделяет всю данную последовательность на слова длины 6 (в данном случае), и поэтому он предпочел выравнивание без инделей, в то время как needle предпочел полное соответствие A302 и A291, T303 и T292, P305 и P294 и схожесть R304 и V293, F306 и V295, Q307 и D296 ценой двух инделей.
2. Описание карты локального выравнивания белков
Для построения карты локального выравнивания были взяты белки, описанные в таблице 1:
| Организм | Poliovirus type 2 | Foot-and-mouth disease virus |
| ID | POLG_POL2W | POLG_FMDVO |
| AC | P23069 | P03305 |
| Рекомендуемое имя | Genome polyprotein | Genome polyprotein |
Аминокислотные последовательности этих белков были выровнены в BLAST, выравнивание с наибольшим счетом:
Query 1592 GVHDNVAILPTH--ASPGETIVIDGKEVEVLDAKALEDQ---AGTNL--EITIVTLKRNE 1644
GV ++P H A + I++DG+ + D + E + G ++ + ++ L R
Sbjct 1684 GVFGTAYLVPRHLFAEKYDKIMVDGRAMTDSDYRVFEFEIKVKGQDMLSDAALMVLHRGN 1743
Query 1645 KFRDIRPHI--PTQITETNDGVLIVNTSKYPNMYVPVGAVTEQG-YLNLGGRQTARTLMY 1701
+ RDI H ++ + V ++N + + A+T + + + G Y
Sbjct 1744 RVRDITKHFRDTARMKKGTPVVGVINNADVGRLIFSGEALTYKDIVVCMDGDTMPGLFAY 1803
Query 1702 NFPTRAGQCGGVITCTGK----VIGMHVGGNGSHGFAAALKRSYFTQSQGEIQWMRPSKE 1757
T+AG CGG + ++G H G G+ + + RS + + I P E
Sbjct 1804 RAATKAGYCGGAVLAKDGADTFIVGTHSAGGNGVGYCSCVSRSMLLKMKAHID-PEPHHE 1862
Query 1758 VGYPV--------INAPSKTKLEPSAFHYVFEGVKEPAVLTKSDPRLKTD--FEEAIFSK 1807
G V ++ KTKL P+ H VF PA L+ DPRL +E IFSK
Sbjct 1863 -GLIVDTRDVEERVHVMRKTKLAPTVAHGVFNPEFGPAALSNKDPRLNEGVVLDEVIFSK 1921
Query 1808 YVGN-KITEVDEYM-KEAVDHYAGQLMS-LDINTEQMCLEDAMYGTDGLEALDLSTSAGY 1864
+ G+ K++E D+ + + YA +L S L + + +A+ G DGL+A++ T+ G
Sbjct 1922 HKGDTKMSEEDKALFRRCAADYASRLHSVLGTANAPLSIYEAIKGVDGLDAMEPDTAPGL 1981
Query 1865 PYVAMGKKKRDILNKQTR----DTKEMQRLLDTYGINLPLVTYVKDELRSKTKVEQGKSR 1920
P+ GK++ +++ + + + +L++ T++KDE+R KV GK+R
Sbjct 1982 PWALQGKRRGALIDFENGTVGPEVEAALKLMEKREYKFVCQTFLKDEIRPLEKVRAGKTR 2041
Query 1921 LIEASSLNDSVAMRMAFGNLYAAFHKNPGVVTGSAVGCDPDLFWSKIPVLMEEKL----F 1976
+++ + + RM G A H N G GSAVGC+PD+ W + +
Sbjct 2042 IVDVLPVEHILYTRMMIGRFCAQMHSNNGPQIGSAVGCNPDVDWQRFGTHFAQYRNVWDV 2101
Query 1977 DYTGYDASLSPAWFEALKMVLEKI-----GFGDRVDYI-DYLNHSHHLYKNKTYCVKGGM 2030
DY+ +DA+ +A+ ++ E++ GF ++I L ++ H Y+NK V GGM
Sbjct 2102 DYSAFDANHCS---DAMNIMFEEVFRTEFGFHPNAEWILKTLVNTEHAYENKRITVGGGM 2158
Query 2031 PSGCSGTSIFNSMINNLIIRTLLLKTYKGIDLDHLKMIAYGDDVIASYPHEVDASLLAQS 2090
PSGCS TSI N+++NN+ + L + Y+G++LD MI+YGDD++ + +++D L
Sbjct 2159 PSGCSATSIINTILNNIYVLYALRRHYEGVELDTYTMISYGDDIVVASDYDLDFEALKPH 2218
Query 2091 GKDYGLTMTPADKSATFETV--TWENVTFLKRFFRADEKYPFLVHPVMPMKEIHESIRWT 2148
K G T+TPADKS + + +VTFLKR F D F PVM K + + +
Sbjct 2219 FKSLGQTITPADKSDKGFVLGHSITDVTFLKRHFHMDYGTGFY-KPVMASKTLEAILSFA 2277
Query 2149 KDPRNT-QDHVRSLCLLAWHSGEEEYNKFLAKIRSVPIGRALLLPEYSTLYRRWLDS 2204
+ R T Q+ + S+ LA HSG +EY + P +P Y +LY RW+++
Sbjct 2278 R--RGTIQEKLISVAGLAVHSGPDEYRRLFE-----PFQGLFEIPSYRSLYLRWVNA 2327
В обоих полипротеинах на выровненных участках находятся части полипротеина, затем модифицирующиеся до протеазы 3C и РНК-зависимой РНК-полимеразы, что может свидетельствовать о явной гомологичности этих белков.
При использовании же программы water для поиска лучшего локального выравнивания, был получен совсем другой результат, не совпадающий с лучшим выравниванием по BLAST даже по области. наиболее соответствующее выравнивание BLAST тут.
Эти выравнивания так же не совпадают как по покрытию, так и по общей области выравнивания - например, основаниям 400-415 из полипротеина полиовируса по результатам выравнивания water соответствует один длинный индель, а по версии BLAST - лишь один индель и множнство остатков. Это может быть связано с гораздо большей длиной сопоставленных участков в первом случае, чем во втором, что сделало бы выгодным использование длинного инделя - water провел почти глобальное выравнивание.
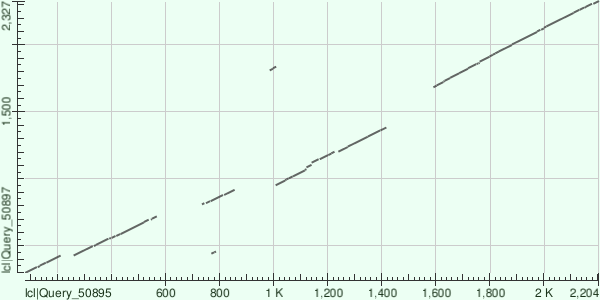
Карта локального сходства этих полипротеинов представлена на рисунке 1:

На карте можно увидеть множнство гомологичных участков, не все из которых расположены по диагонали. Так, участку 770-790 последовательности полипротеина полиовируса, судя по карте, соответствуют сразу два участка в последовательности вируса ящура. Скорее всего, это связано с дупликацией этого участка гена в процессе эволюции.
Участкам же 570-730 а. о. и 860-980 а. о. полипротеина полиовируса не соответствует никакой участок полипротеина ящура, при том что у выравнивания соответствующих участвков последовательности из вируса ящура в этих местах разрывов нет. Это можно объяснить либо делецией выделенных участков у ящура, либо дупликацией в эти места других частей генома полиовируса.