Эволюция доменной архитектуры
Информация о выбранном семействе доменов
Для выполнения данного задания было выбрано семейство доменов PF00129, Class I Histocompatibility antigen, domains alpha 1 and 2(MHC_I ). MHC класса I, также известные как молекулы основного комплекса гистосовместимости (major histocompatibility complex), представляют собой гликопротеиды, локализующиеся на клеточной мембране всех соматических клеток за исключением нейронов. Они играют важную роль в регуляции иммунного ответа на чужеродные антигены, а также сами являются сильными антигенами. МНС класса I обеспечивают представление (презентацию) фрагментов антигенов, включая самопептиды, цитотоксическим Т-лимфоцитам, которые обладают способностью уничтожать вирусно-инфицированные или злокачественные (избыток самопептидов) клетки.
Молекулы МНС класса I – это гетеродимеры, состоящие из двух цепей: α-цепи МНС (тяжелая цепь) и цепи β2-микроглобулина (легкая цепь), который также встречается в свободной форме в сыворотке крови; из них только α-цепь охватывает мембрану. α-цепь имеет три внеклеточных домена ( α-1-3, с α-1 на N-конце), трансмембранный участок и C-концевой цитоплазматический хвост. Растворимая внеклеточная цепь β2-микроглобулина связывается преимущественно с α-3-доменом, что необходимо для стабилизации димера. Домены α-цепи α-1 и α-2 называются областью распознавания, поскольку пептидный антиген связывается MHC в удлиненной глубокой полости, образуемой между этими двумя доменами при их взаимодействии друг с другом.
AC
: PF00129
ID
: MHC_I
34
architectures
104
species
2585
sequences
1390
structures
Выравнивание доменов выбранного семейства было скачено из Pfam и визуализировано в программе JalView, также к выравниванию была добавлена 3D структура белка A01_HUMAN, PDB ID: 4nqv. Все выравнивание раскрашено ClustalX, порог консервативности - 10. Ссылка на проект.
На рисунке 1 представлена 3D структура
1A01_HUMAN: HLA class I histocompatibility antigen, A-1 alpha chain (HLA -
human leukocyte antigens, антигены лейкоцитов человека), принадлежащий к
группе антигенов класса I молекул главного комплекса гистосовместимости
MHC.

Рисунок 1. 3D структура антигена лейкоцитов человека
Выбор архитектур
Для дальнейшей работы из 34 известных архитектур, содержащих домен MHC_I, мною были выбраны следующие:

Рисунок 2. Графическое представление выбранных архитектур
MHC_I – гетеродимеры, молекулы класса I основного комплекса гистосовместимости, представляющие собой гликопротеиды, локализованные на мембране клетки. Они играют роль в регуляции иммунного ответа, обеспечивая представление антигенов Т-лимфоцитам, уничтожающим инфицированные и злокачественные клетки.
C1-set – классический Ig-подобный домен. С1-set домен встречается почти исключительно у молекул, вовлеченных в иммунную систему, например, в легких и тяжелых цепях иммуноглобулинов, сложных молекулярных комплексах (гетеродимерах) основного комплекса гистосовместимости (МНС) I и II и в различных рецепторах Т-клеток.
С помощью скрипта swisspfam_to_xls.py (/srv/databases/pfam/swisspfam.gz) была получена excel таблица, содержащая информацию об архитектуре всех последовательностей, содержащих указанный домен (PF00129):
python swisspfam-to-xls.py -i /srv/databases/pfam/swisspfam.gz -z -p PF00129 -o PF00129.xls
Согласно указаниям по выполнению практикума, была составлена сводная таблица, где строки – AC последовательностей, а столбцы – домены Pfam. Таблица была добавлена в итоговый excel файл, где в дальнейшем проводилась сортировка по доменам и их количеству (лист "поиск архитектур"), таким образом, чтобы определить белки, соответствующие выбранным структурам (см. рисунок 2).
Выбор таксонов представителей архитектур
Затем я сохранила список AС из выдачи скрипта swisspfam_to_xls.py и провела поиск по базе данных Uniprot (AC → Retrieve), полученные данные были сохранены в файл uniprot-yourlist.txt. И с помощью скрипта python uniport-to-taxonomy.py были получены данные о таксономии организмов, содержащих белки с выбранными AC:
python uniport-to-taxonomy.py -i uniprot-yourlist.txt taxonomy_only.xls
Результаты работы были добавлены в итоговый excel файл (лист taxonomy result).
Для дальнейшей работы мною был выбран класс Mammalia и два его подтаксона: отряд Primates и Glires - клада ранга грандотряда (grandorder), в которой объединяют отряды грызунов (Rodentia) и зайцеобразных (Lagomorpha). Из подходящих по архитектуре белков мною были случайным образом отобраны по 30 представителей каждой из архитектур для каждого подтаксона (всего 120). AC отобранных белков были сохранены в текстовый файл my_ac.txt.
Используя скрипт filter_alignment.py, я отфильтровала выравнивание доменов выбранного семейства, скаченное из Pfam на начальном этапе работы, по списку АС my_ac.txt, оставив только выровненные последовательности доменов 120 отобранных представителей:
python filter-alignment.py -i PF00129_all_align.fasta -m my_ac.txt -o my_align.fasta -a "_"
Из 120 отобранных представителей скриптом были найдены 108. Полученное выравнивание было обработано вручную с помощью программы JalView:
- последовательности переименованы согласно доменной архитектуре и таксономическому положению
- выравнивание разбито по архитектурам на 2 группы, группы покрашены ClustalX by conservation (50)
- удалены пустые и неинформативные столбцы
- удалены N-концевой участок в обеих группах и С-концевой участок в группе I
- удалены последовательности, которые плохо выравнивались
Скачать проект можно по ссылке.
На рисунке 3 представлено изображение финального выравнивания последовательностей белковых доменов отобранных представителей, почищенного и разбитого на группы по архитектурам (проекта).

Рисунок 3. Блок выравнивания проекта .jvp
Построение филогенетического дерева
Ранее для наглядности и удобства интерпретации построенного
филогенетического дерева последовательности были переименованы в
соответствии с доменными архитектурами:
I – однодоменный (MHC_I (PF00129)),
II - двухдоменный (MHC_I (PF00129) + C1-set (PF007654))
и таксономическим положением (G - Glires, P - Primates).
Итоговое выравнивание в формате fasta было обработано программой MEGA, построение филогенетическое дерева было произведено методом Neighbor-Joining, для оценки достоверности ветвей был применен метод бутстрэп реплик с количеством реплик 100. На рисунке 4 представлено полученное дерево, ветви окрашены по архитектурам.
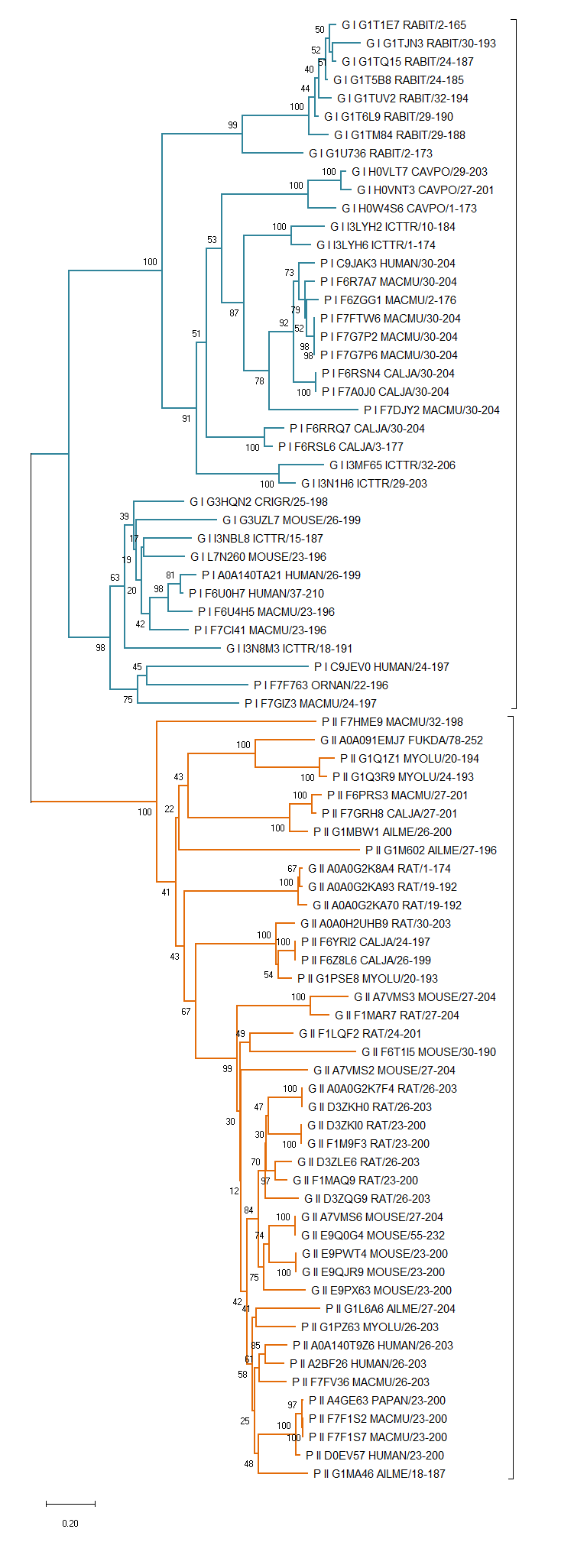
Рисунок 4. Филогенетическое дерево организмов, построенное по представляющим две различные доменные архитектуры последовательностям белковых доменов (по 20 представителей каждой из архитектур на таксон, итого 80), содержащим этот домен (анализируемые в данном практикуме архитектуры – 2 из 34 описанных архитектур, содержащих домен MHC_I)
В построенном дереве наблюдается четкое разделение в линии эволюции по доменным архитектурам, но не по таксонам. Так, видно, что последовательности различные по доменным архитектурам образуют две клады, внутри которых расположены последовательности из разных таксонов. Основываясь на организации древа, можно предположить, что разделение белков с доменом MHC_I на две группы произошло до разделения данных таксонов, о чем свидетельствует наличие в каждом таксоне белков - представителей обеих архитектур.
© Енькова Анна, 2017 |