Пакет 3DNA. A-, В- и Z- формы DNA. Структура RNA
Этот практикум посвящён анализу строения различных форм нуклеиновых кислот, теоретически предсказанных с помощью пакета 3DNA и полученных экспериментально.
Генерация структуры DNA
Команда fiber из пакета 3DNA позволяет моделировать вторичные структуры нуклеиновых кислот в формате pdb.
Однако, сгенерировать она может не какие угодно структуры. Поэтому воспользуемся
fiber -m, чтобы получить список структур, доступных для моделирования.
К сожалению, Z-ДНК с произвольным нуклеотидным составом получить нельзя (есть всего два варианта: поли-As4T и поли-GC -спираль).
Для генерации файлов были использованы следующие команды:
fiber -seq=GATCGATCGATCGATCGATC -a gatc-a.pdb
fiber -seq=GATCGATCGATCGATCGATC -b gatc-b.pdb
fiber -z gatc-z.pdb
Затем для поли-GC-Z-спирали я ввёл число 20 (число пар оснований).
Полученные модели можно скачать здесь:
Сравнение предсказанной модели с экспериментальными данными
В этом задании нужно было сравнить сгенерированную нами модель с экспериментальными образцами.
Для этого я решил взять pdb файл B-формы DNA 1BNA. В B-форме определить бороздки несложно: большая значительно шире малой.
На рис. 1 изображён цитозин, находящийся на положении 15 цепи B экспериментальной модели. Изображение получено с помощью использованного нами ранее MarvinSketch.
- В сторону большой бороздки обращены атомы: С15.C4, С15.N4, С15.C5, С15.C6.
- В сторону малой бороздки обращены атомы: С15.N1, С15.C2, С15.O2, С15.N3.
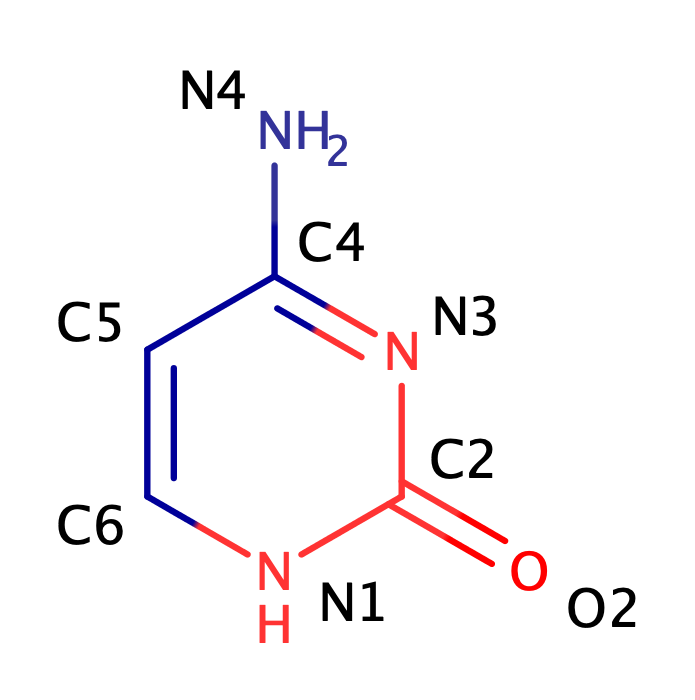
смотрят в сторону малой бороздки, а синие - в сторону большой.
Таблица 1. Параметры разных структур DNA
| A-форма | B-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали, Å | 28,03 | 33,75 | 42,35 |
| Число оснований на виток | 11 пар | 10 пар | 12 пар |
| Ширина большой (глубокой) бороздки, Å | 7,98 (T11:A - C24:B) | 17,21 (A12:A - A26:B) | 13,21 (C40:A - C44:B) |
| Ширина малой бороздки, Å | 16,81 (С20:A - T23:B) | 11,69 (C20:A - G25:B) | 7,2 (G39:A - G45:B) |
Структура RNA
В этом задании мы должны были определить параметры структуры РНК с помощью программ find_pair и analyze
уже знакомого нам пакета 3DNA. Я рассматривал структуру с PDB ID: 1F7U. Я конвертировал его в старый формат, чтобы можно было работать с пакетом:
remediator --old ''1f7u.pdb'' > ''1f7u_old.pdb''
Затем мы определили спаренные основания и положение спиралей в структуре:
find_pair -t 1f7u_old.pdb stdout | analyze
В результате получился 1f7u_old.out и stacking.pdb.
В некотором приближении можно считать, что тяжи RNA похожи на A-форму DNA.
Таблица 2. Сравнение средних значений торсионных углов модели с теоретическими.
| Углы | α | β | γ | δ | ε | ζ | χ |
| A-форма (теоретическая) | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| B-форма (теоретическая) | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
| Цепь 1 (среднее) | -40 | 56 | 57 | 90 | -144 | -65 | -144 |
| Цепь 2 (среднее) | -39 | 32 | 46 | 89 | -149 | -34 | -157 |
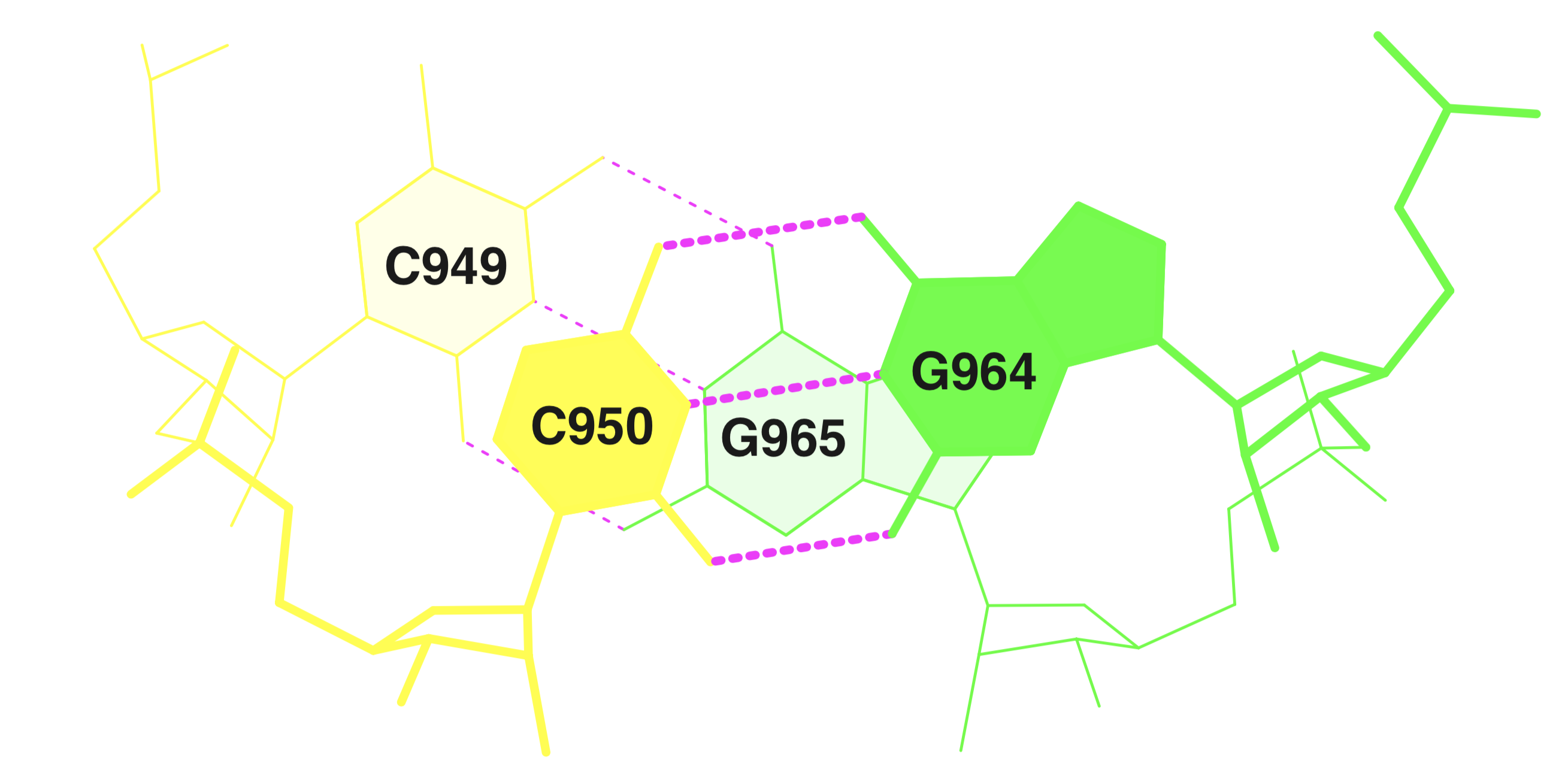
Теперь рассмотрим неканонические пары всё в том же файле. Видно, что в RNA присутствуют псевдоуридин, 5-метилурацил и N-метилгуанозин:
B:.901_:[PSU]P-**--A[..A]:.972_:B B:.905_:[..U]U-*---G[..G]:.968_:B B:.954_:[5MU]t-**--a[1MA]:.958_:B B:.955_:[PSU]P-**+-G[..G]:.917_:B B:.943_:[..A]A-**--P[PSU]:.927_:B B:.944_:[..A]A-**--g[M2G]:.926_:B B:.913_:[..C]C-**--C[..C]:.922_:B B:.914_:[..A]A-**--U[..U]:.908_:B B:.915_:[..A]A-**+-U[..U]:.948_:B
Координаты стеблей:
(901...907 - 966...972)
(949...955 - 958...965)
(939...944 - 926...931)
(910...918 - 923...925)