
Комплексы ДНК-Белок.
| GO TO: |
| Таблица контактов ДНК-БЕЛОК | ||||||||||||||||||||||||
|
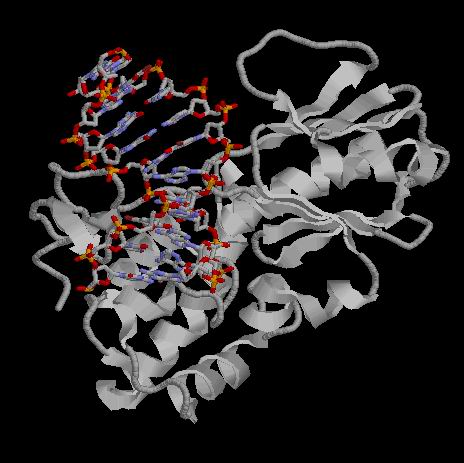
В данной работе нам было пердложенно исселдовать заданную структуру из PDB
(code: 1K3X), содержащую белок
DNA repair enzyme endonuclease viii класса
E.C.4.2.99.18,
на наличие в ней связей между ДНК и Н-терминальным доменом белка, выяснить их количество и характер.
Для этого из исходного PDB файла были удалены все остатки, начиная с 105-ого. Поиск связей осуществлялся программой RasMol с использованием следующего скрипта (note: скрипт не демострирует самих связей в виде пунктирных палочек и не пишет расстояний - ибо это можно делать только при работе с отдельными атомами, а не со множествами, но в процессе работы скрипта можно измерять расстояния), в результате получились следующие данные: Таблица. Контакты разного типа в комплексе 1K3X
Из таблицы хорошо видно, что белок контактирует в основном с атомами сахарных остатков и полярными атомами фосфатов, что видимо объяснеятся тем, что атомы оснований смотрят скорее "внутрь" спирали. |
||||||||||||||||||||||||
| Поиск специфических контактов, обеспечивающих узнавание сайта в молекуле ДНК | ||||||||||||||||||||||||
|
В качестве наиболее специфичного контакта между ДНК и Н-терминальным доменом белка были выбраны контакты трех последовательно лежащих
аминокислот GLN69, LEU70 и TYR71, а так же двух последовательных ак PRO1 и GLN2, с фрагментом моей днк. Особенность этого контакта состоит в том, что ДНК
в этом месте явно дефектна: Выделенный красным Тимин имеет разорванное сахарное кольцо, а основание вообще отсутсвует.
Особождённый из кольца, и выставленный тем самым вбок атом O4' связан полярной связью с одним из OE атомов GLN2, в брешь в бороздке между двумя
цепями входит триплет GLN69-LEU70-TYR71, взаимодействующий как полярным, так и гидрофобным способом с
окружающими его нуклеотидами. Очевидно, что такой контакт возможен только при наличии разорванного кольца и бреши между двумя цепями,
образованной отсутсвием основания у дефектного нуклеотида.
|
||||||||||||||||||||||||
| Описание белка и его основных доменов в базах Uniprot и Pfam. | ||||||||||||||||||||||||
|
Мой белок записан в базе Uniprot как END8_ECOLI
и является DNA repair enzyme endonuclease viii. Это белок E.Coli K12(как впрочем вероятно и других штаммов).
Он принимает участие в процессах ремонта повреждённых вследствие окисления или мутаций ДНК. Данный белок
узнает, прежде всего, повреждённые пиримидины, такие
как тимингликоль, 5,6-дегидроурацил, 5,6-дегидротимин. Будучи лиазой, он удаляет повреждённые основания,
оставляя разрез на повреждённой цепи ДНК-овой спирали, который, видимо, затем должен узнаваться другими
ремонтными белками, призванными заделать эту щель. При этом этот белок вместе с повреждённым основанием
и его сахарным кольцом удаляет и оба фосфата с 5' и 3' концов.
Исходя из данных Pfam в белке есть два домена:
Первый домен - тот, который мы и рассматриваем - это собственно ремонтный домен, который осуществляет удаление повреждённых оснований из ДНК. Второй домен является по сути ДНК-связывающим доменом. |
||||||||||||||||||||||||
| Поиск контактов ДНК-белок программой nucplot | ||||||||||||||||||||||||
|
Так же для поиска контактов ДНК-белок в моей структуре была использована программа nucplot.
К сожалению исходный файл не обрабатывался программой, поэтому он был изменён следующим образом: все группы атомов вида
DG, DC, DT и DA были заменены на G, C, T и А соответсвенно; группы BRU были заменены на T. В обозначениях всех атомов сахаров в днк " ' " был заменён на " * ", а все атомы OP1 и OP2 на O1P и O2P соответсвенно. Помимо этого из записи были удалены все атомы воды, дабы не захламлять рисунок. После этой замены nucplot обработал полученный файл pdb и выдал следующий результат:
Из результатов видно, что предложенные в качестве специфческих контактов мотивы (GLN69-LEU70-TYR71) и (PRO1-GLN2) дейтвительно специфически взаимодейтсвует 1-ый с 407-ым A, у которого нет парного основания, 2-ой - с дефектиыным 427-ым Т (DA407 и PED427 на рисунке выше - картинка создана с "новоформатного" pdb).
NUCPLOT v.1.0 - Bond file (1K3X.bond)
-----------------------------------------------
**** Hydrogen Bonds ***************************
Donor Acceptor Distance
LYS A 52 NZ G C 428 O1P 2.71
LYS A 52 NZ A C 429 O2P 2.72
HIS A 67 NE2 A C 429 O1P 2.74
GLN A 69 NE2 A B 407 N3 2.93
VAL A 88 N C B 409 O1P 2.86
ARG A 90 NH1 T B 408 O1P 2.90
ARG A 90 NH2 T B 408 O1P 2.92
SER A 104 OG C B 409 O1P 2.67
**** Non Bonded Contacts **********************
protein DNA Distance
PRO A 1 C T C 427 C1* 2.96
PRO A 1 CD T C 427 C2* 3.29
GLU A 2 OE2 T C 427 C4* 3.22
GLU A 2 OE2 T C 427 O4* 2.56
TYR A 71 OH A B 407 C1* 3.22
ARG A 87 CA C B 409 O1P 3.25
**** Covalent Bonds ***************************
protein DNA Distance
PRO A 1 N T C 427 C1* 2.96
В целом же сравнивать результаты полученные в ручную в первом задании и этой программы сложно: Во-первых, программа закономерно считает, что кислороды остатков фосфорной кислоты образуют скорее водородные связи с белком, нежели полярные, в то время как мы считаем их полярными, во-вторых, программа в принципе настроена искать прежде всего водородные связи, считая остальные ги дрофобные взаимодействия не столь важными. Ниже представлено выравнивание моего домена по базе Pfam (выравнивание было сделано по всем имеющимся белкам с этим доменом - их 665шт.): Как видно по выравниванию, пара PRO1-GLN2 действительно является довольно постоянным мотивом в моём домене. Выделенный же мной триплет оказался менее постоянным. Наиболее консервативным оказался Leu70, в то время как два остальных остатка варьировались довольно заметно. | ||||||||||||||||||||||||