Особенности мембранных белков
Структуры мембранных белков
Мембранные белки должны отвечать основному требованию: обращенные во внешную сторону (к собственно билипидному слою) атомы должны составлять гидрофобную структуру. В связи с этим есть два основных варианта вторичной структуры этих белков: альфа-спирали, практически полностью состоящие из гидрофобных аминокислотных остатков, и "бочонки" из бета-листов, или баррели, в которых все карбоксильные группы каждого листа задействованы в образовании водородных связей с атомами соседнего листа.
В данном разделе представлена информация о трех случайных спиральных и баррельных мембранных белках.
Данные приведены в таблице 1. Они извлечены из баз данных (БД) OPM (Orientations of Proteins in Membranes) и TCDB (Transporter Classification Database). Для белков, которые есть во второй БД, указан TCID и функции, соответственно, из этого источника.
Таблица 1. Данные о нескольких мембраннах белках: спирали и баррели во вторичной структуре. К PDB коду прикреплены ссылки на страницу OPM для данного белка. Сокращение ТМ означает слово "трансмембранный".
| PDB код | Тип | Мембрана и некоторые функциональные особенности белка | TCID | Толщина гидрофобной части мембраны, A | Медиана числа остатков в одном ТМ участке |
| 1R3J | Спираль | Цитоплазматическая мембрана грамположительных бактерий (ионный калиевый канал) | 1.A.1.1.1 | 34.8 ± 1.2 | 26 |
| 1E12 | Спираль | Мембрана Halobacterium salinarum (Archaebacteria) (связан с переносом хлора в клетку) | 3.E.1.2.1 | 31.8 ± 1.4 | 22 |
| 2UUH | Спираль | Мембрана эндоплазматического ретикулума Homo sapiens (метаболизм глутатиона) | - | 29.4 ± 1.1 | 18,5 |
| 3O44 | Баррель | Секретируется Vibrio cholerae | - | 24.6 ± 0.0 | 7 |
| 1PHO | Баррель | Наружная мембрана грамотрицательных бактерий (поглощение неорганического фосфата, фосфорилированных соединений и некоторых других отрицательно заряженных растворенных веществ) | 1.B.1.1.2 | 24.1 ± 1.2 | 9 |
| 2JK4 | Баррель | Внешняя мембрана митохондрий Homo sapiens (ионный канал) | - | 23.4 ± 2.3 | 7 |
Белок MetI
Белок MetI E.coli и его гомологи в других организмах
Белок MetI (PDB код: 3TUI, Uniprot ID: P31547) принадлежит бактерии E.coli филума Proteobacteria (класс гамма-протеобактерии). Данный раздел посвящен составлению репрезентативной выборки гомологов этого белка в различных организмах.
Для создания такой выборки можно использовать два метода: белковый BLAST и множество усилий или несколько итераций PSI-BLAST (который используется для составления семейств гомологов). В данном случае мы выбрали blastp (protein-protein BLAST). Ход работы и результат приведены ниже.
Сперва было подобрано время, когда в США еще не рассвело и биоинформатики не начали бластовать, потому что белок содержить очень частый домен ABC-транспортера, что означает необходимость в большом количестве хитов для хорошей выборки.
Для поиска использовали БД refseq. Была произведена попытка провести blast по всем организмам. Но даже при количестве показанных хитов 1000 худшие найденные выравнивания имели хороший e-value (порядка 10e-40), их длина была около 200 (что соответствует длине исходной последовательности), то есть этим выравниваниям, вероятно, можно было доверять. В связи с таким большим количеством гомологичных последовательностей было решено проводить blast по разным таксонам: отдельно по археям, по прокариотами (без архей и исключая протеобактерий) и эукариотам. Поиск проводили с порогом e-value: 1e-5, максимальным количеством хитов: 1000 для поиска по археям и эукариотам, 5000 для поиска по прокариотам. В последнем случае было найдено 4756 хитов, последние находки имели пороговй e-value. Соответственно, была возможность вычерпать разнообразие гомологов среди прокариот.
В каждом случае получали GenPept и выбирали по несколько последовательностей белков организмов, принадлежащих разным таксонам (в случае бактерий - к разным филумам, белок встречается в большом количестве филумов).
Итого вышло 3 гомолога из архей, 12 гомологов из разных групп бактерий. Все же для дальнейшей работы работы 1 архейная последовательность была удалена (длина белка была в два раза больше, чем у исходной последовательности). Среди эукариот было найдено около десятка последовательностей, но все они оказались "предсказанными", то есть для них нет доказательства существования белка на молекулярном уровне. Поэтому в выборку они не добавлены.
Были получены последовательности 14 гомологов в fasta-формате (в названиях указаны организмы и идентификаторы белков).
Анализ множественного выравнивания гомологов MetI
С помощью программы MUSCLE было построено множественное выравнивание отобранных гомологов. Результат был интегрирован в программу JalView.
К выравниванию были добавлены аннотации TM_REAL и TM_PREDICTED.
Для выравнивания была применения цветовая схема, позволяющая визуально различать гидрофобные и гидрофильные остатки (Hydrophobicity: гидрофобные остатки выделены красным цветом), вместе с зависимостью интенсивности цвета от консервативности позиции (By Conservation, было использовано значение 25% в качестве порога). Полученное изображение структуры приведено на рисунке 1.
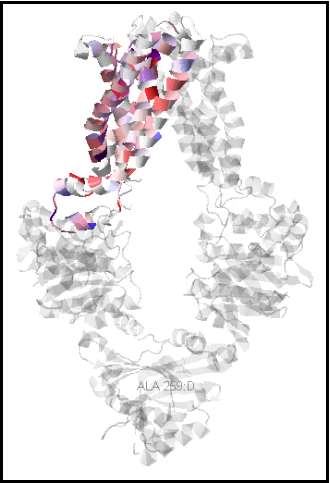
Рисунок 1. Изображение структуры MetI. Ярко выделена А-цепь. Пояснения в тексте.
Часть белка, ориентированная в n-сторону мембраны (в сторону цитоплазмы), расположена снизу (так же изображают этот белок в OPM). Участки, относящиеся к трансмембранным спиралям, консервативны (хотя встречаются фрагменты участков неконсервативных остатков), при этом наиболее часто в спиралях встречаются остатки лейцина, изолейцина, валина, аланина, фениалаланина, метионина, глицина и пролина - гидрофобных аминокислот. Среди участков между спиралями встречаются как консервативные участки, так и неконсервативные (для порога 25% для цветового отображения консервативности).
Отметим также, что в трансмембранных участках встречаются консервативные остатки полярных аминокислот. Вероятно, это как-то связано с функцией поддержания структуры.
Стоит отметить, что в трансмембранных спиралях также нередко встречаются остатки серина, аспарагина,
Проект выравнивания в JalView можно скачать по ссылке.
Аннотация TM_REAL содержит указания на участки выравнивания отвечающие трансмембранным спиралям в белке со структурой 3TUI:A, которая была прикреплена к последовательности белка в выравнивании в JalView.
Аннотация TM_PREDICTED обозначает предсказанные с помощью TMHMM трансмембранных спиралей в гомологе из Oscillibacter (WP_021748569.1). Предсказание представлено на рисунке 2.
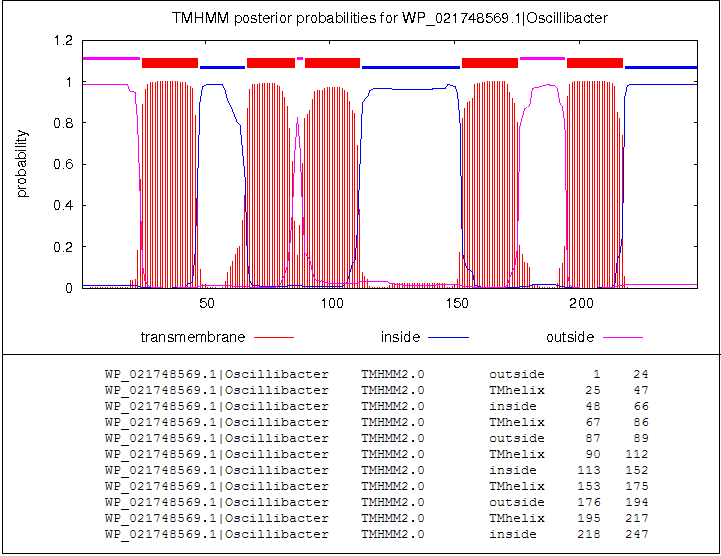
Рис. 2. Предсказание TM_PREDICTED для гомолога MetI из Oscillibacter.
На рисунке 3 приведено полученное выравнивание. Для получение полноразмерного изображения необходимо кликнуть по картинке.
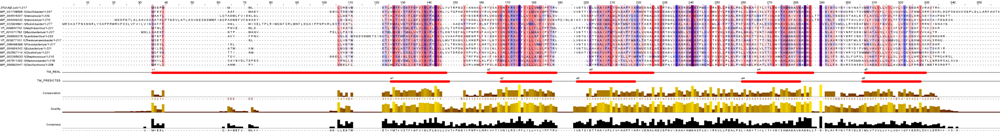
Рис. 3. Выравнивание гомологов MetI. Пояснения в тексте.
Всего в предсказании найдено 5 альфа-спиралей. Это соответствует количеству трансмембранных спиралей в белке MetI. Спираль 1 в TM_REAL длинная (она проходит 27 аминокислотных остатков MetI, или 3TUI), видно, что у некоторых гомологов первые 5-10 остатков не участвуют в образовании спирали. Вторая спираль более консервативная (в MetI длится с 51 по 80 а.о.). Третья предсказанная спираль перекрывается по консервативному блоку с точной (из 3TUI, 89-108 а.о.). Аналогичная ситуация наблюдается для 4 и 5 спиралей (в 3TUI - 146-167, 187-210 а.о.). Некоторые найденные элементы совпадают с частями вторичной структуры MetI. Однако, для гомолога из Oscillibacter нет вторичной структуры, поэтому мы не оцениваем правильность предсказания по вторичной структуры. Тем не менее, предсказание, вероятно, правдивое, так как трансмембранные спирали TM_REAL и TM_PREDICTED почти совпадают.
Анализ структуры MetI
MetI (D-methionine transport system permease protein) был найден в различных БД (OPM, TCDB). В этом разделе приведена некоторая информация об этом белке. Информация о структуре приведена в таблице 2.
Таблица 2. Описание структуры трансмембранного белка MetI (3TUI:A).
| PDB ID | Организм | Мембрана | TC-код | Угол наклона спиралей (бета-тяжей) к нормали | Количество трансмембранных спиралей |
| 3TUI | E.coli | Внутренняя | 3.A.1.24.1 | 1 ± 1° | 10 |
Расшифровка TC-кода для 3TUI:
- 3.* - первичные активные транспортеры;
- 3.A.* - транспортеры, осуществляющие гидролиз P-P-связей;
- 3.A.1.* - суперсемейство белков, содержащих АТФ-связывающую кассету (ATP-binding cassette, ABC);
- 3.A.1.24.* - поглощающие метионин;
- 3.A.1.24.1 - L- и D-метионин-транспортеры у протеобактерий
В TCDB есть 2 белка с таким же TC-кодом, как у MetI: MetN и MetQ. В OPM MetI относится к семействую белок-связывающих транспортных систем (1.1.10.02) с 32 белками. Белки суперсемейства АВС (3.А.1) катализируют две реакции в две стороны (поглощение и секреция):
- Solute (out) + ATP ---> Solute (in) + ADP + Pi.
- Substrate (in) + ATP ---> Substrate (out) + ADP + Pi.
10 трансмембранных спиралей белка MetI участвуют в переносе соединений (D-methionine Transport System Permease Protein MetI). В цитоплазматической части белка находятся сайты связывания АТФ (Methionine Import Atp-binding Protein Metn), который превращается в АДФ. На рисунке 4 приведены изображения из NCBI, схематично показывающие эти структуры белка.
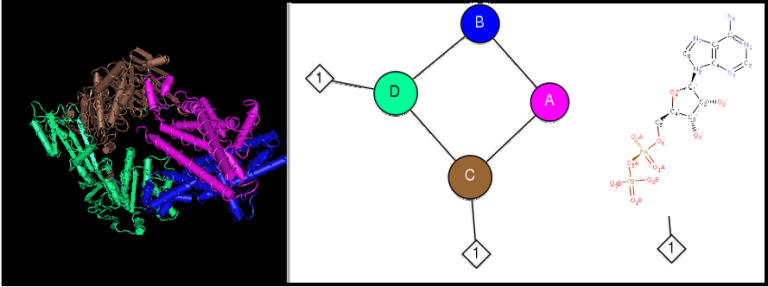
Рисунок 4. Справа - 3D-структура комплекса MetI и Metn: зеленым и коричневым выделены - АВС-АТФазные части, розовым и синим - часть пермеазы. Слева - схематичное представление и связывание с лигандом - АДФ, приведена также структурная формула последнего.
Также в аннотации PDB можно найти схему реальной вторичной структуры белка. С ним было бы удобнее переносить в множественное выравнивание элементы альфа-спиралей в JalView, чем с использованием 3D структуры, связанной с последовательностью. Две схемы приведены на рисунке 5.
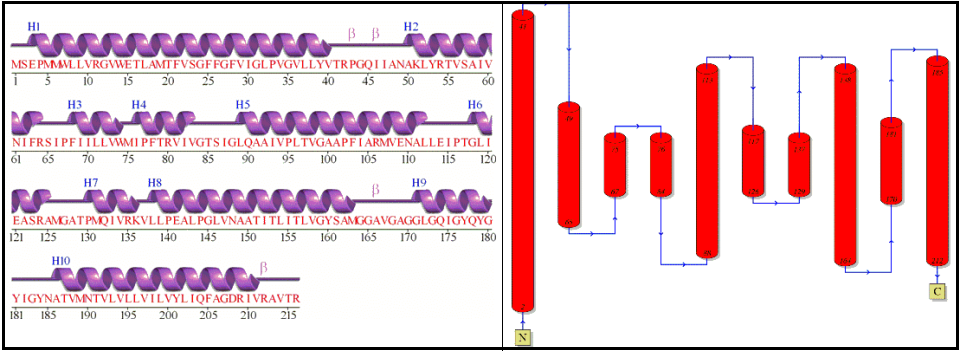
Рис. 5. Вторичная структура С-цепи белка: с указанием аминокислотных остатков (слева), с указанием только номеров остатков (справа).
Клетка размером с дыню
В качестве отсутпления приведем оценку толщины мембраны в клетке размером с плод Cucumis melo L., геном которой был отсеквенирован в 2012 году. Клетка бактерии E.Coli, которой принадлежит белок MetI, имеет диаметр около 1 мкм. Средняя дыня, предположим, имеет диаметр (среднее арифметическое между длиной и шириной) 20 см. Тогда линейный размер дыни превышает размер клетки в 20000 раз. Толщина мембраны клетки примерно равна 40 Ангстремам. Значит толщина мембраны клетки размером с дыню будет равна примерно 0,08 см.
Правило positive inside
Для белка MetI (цепь A) можно выяснить, выполняется ли правило positive inside для трансмембранных белков, состоящее в том, что часть белка, расположенная с n-стороны от мембраны, содержит больше положительно заряженных остатков. Если рассмотреть остатки положительно заряженных аминокислот (лизина, аргинина и гистидина) в трансмембранных участках данного белка и определить с какой стороны мембраны число положительно заряженных остатков больше, то можно наблюдать чёткое следование расположения остатков этому правилу. Оно очевидно в данном случае, потому что основная часть белка и так находится в цитоплазме клетки (inside), поэтому и аминокислот с любыми свойствами должно быть больше. Тем не менее, в части белка за пределами клетки положительных аминокислот нет совсем. И всего несколько встречается в трансмембранном участке. На рисунке 6 приведена структура белка с выделенными остатками положительно заряженных аминокислот.
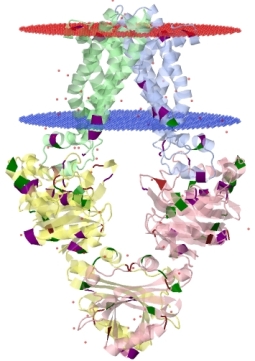
Рис 6. MetI и положительно заряженные аминокислоты: аргинин - фиолетовый, лизин - зеленый, гистидин - красно-коричневый (всего несколько остатков). Снизу - внутренняя часть клетки с цитоплазматической частью белка.
Предсказание бета-баррелей по последовательности
Данный раздел работы посвящен проверке сервиса предсказания бета-барреля по последовательности - PRED-TMBB. Для этого был взят белок с известной вторичной структурой - 2jmm, принадлежащего E.coli. Был найден его гомолог без известной структуры - (представлена последовательность в fasta-формате) из клещевины Ricinus communis. Было получено выравнивание с помощью muscle.
В PRED-TMBB было проверено, что для 2jmm получается правильно предсказание. Было получено предсказание для белка клещевины. На рисунке 7 представлены эти два предсказания.
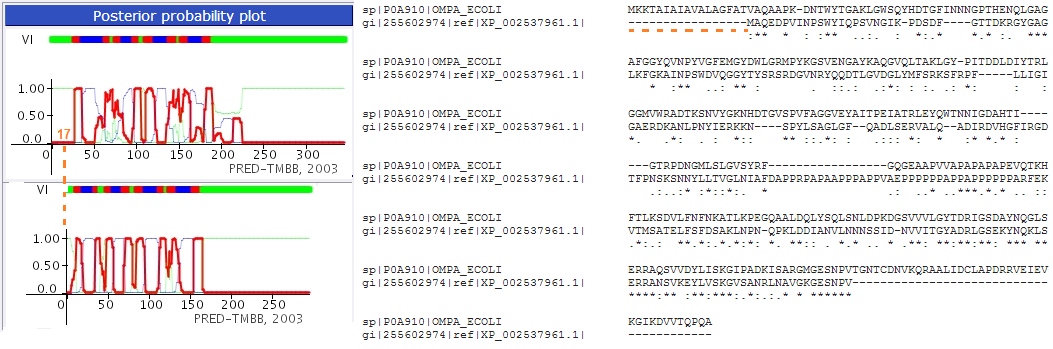
Рис. 7. Пресказания PRED-TMBB для последовательностей 2jmm (сверхну) и гомолога из клещевины (снизу). Справа - выравнивание muscle.
Мы видим, что предсказание для гомолога белка 2jmm очень правдоподобное. На рисунке предсказание для гомолога смещено вправо в связи с тем, что выравнивание начинается с его 17-го аминокислотного остатка. Похоже, не пропущен ни один трансмембранный элемент. В середине есть смещение, которое тоже связано с дополнительным куском последовательности в клещевине.
Для предсказания бета-баррелей можно также использовать, например, сервисы TBBpred или BOCTOPUS. Но их минус в том, что там не очень удобное представление выдачи. Оно понятное, но с помощью этих сервисов не удалось бы представить результат так, как показано выше.
Остатки бета-барреля, обращенные в мембрану, гидрофобны
В данном разделе работы был проведен анализ структуры белка 2jmm. Для этого был использован сервис SheeP - инструмент для описания бета-листов в 3D структурах белка, разработанный преподавателями биоифнорматики Аксяновым Е.А. и Алексеевским А.В. На составленной карте, приведенной на рисунке 8, красным выделены остатки, боковые цепи которых "смотрят" наружу баррели, жёлтым – внутрь.
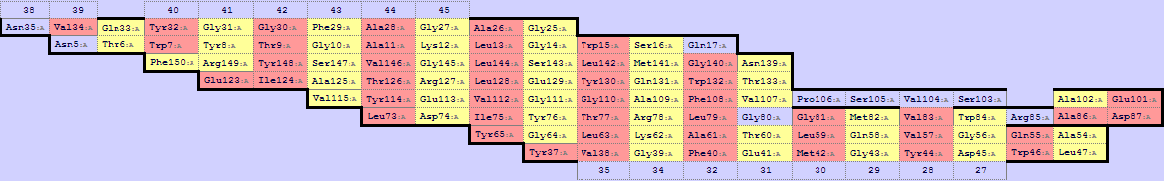
Рис. 8. Карта остатков цепи белка 2jmm, полученная в SheeP (SheeP sheet map), пояснения в тексте.
На риснуке 9 представлены: локализация белка в мембране (из OPM); изображение 3D структуры, на котором смотрящие наружу боковые цепи указаны красным цветом, а также приведена структура, в которой коричневым отмечены полярные остатки. Видно, что они в не входят в трансмембранную часть белка. Они либо близко расположены к поворотам, либо находятся в цитоплазматической части белка, либо не являются обращенными в мембрану. В это время обращенные в мембрану аминокислотные остатки гидрофобны.
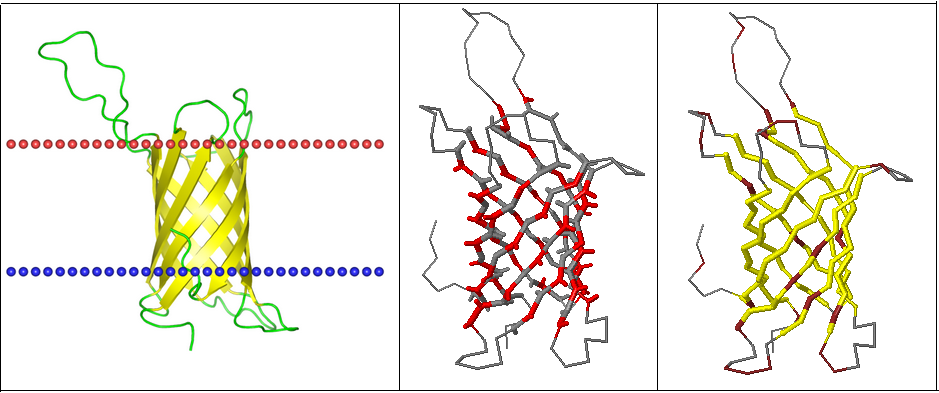
Рис. 9. 3D структуры белка 2jmm. Пояснения в тексте.