Расссмотрение структуры белка неорганической пирофосфатазы (inorganic pyrophosphatase) из генома бактерии Helicobacter Pylori штамм J99 в программе Jmol
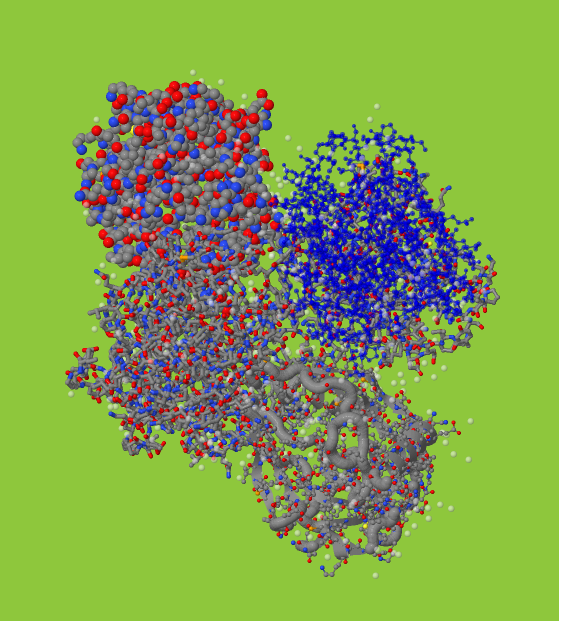
Рисунок 1. Структура белка неорганической пирофосфатаза в программе Jmol.
Белок состоит из 6 кластеров одинаковой структуры. Каждому присвоена буква английского алфавита: A,B,C,F,E,D.А кластер изображена в виде шариков, где каждый шарик представляет собой атом какого-либо химического элемента (данная структура в программе называется cpk). Так же атомы покрашены в стандартные цвета в зависимости от атома: углерод - серый, азот - синий, кислород - красный, водород - бесцветный, сера - желтая (такая раскраска также называется cpk).
В и Е кластеры изображены в виде связей (левая нижняя и права по центру). Такой тип называется wireframе, что дословно означает "проволока". Данный тип является наиболее наглядным, так как отражает все возможные связи между атомами в структуре белка. Раскраска соответственно тоже cpk.
C и F кластеры изображены в виде связей. В нём (backbone) все структуры представлены в связей между альфа-атомами аминокислот, без особого углубления в атомную структуру и связи в данном элементе белка. Раскраска у кластера С - синий цвет (команда color цвет), у F - chain (разные элементы имеют разный цвет).
Ниже представлен скрипт для воссоздание данного изображения в Jmol. Достаточно сохранить этот текст в текстовый документ и изменить формат на spt. Затем просто запустить его при помощи bat-файла в дистрибутиве Jmol:
load 1YGZ.pdb
background [140,198,62]
select *A
cpk 250
color cpk
select *B
wireframe 100
color cpk
select *C
backbone
color blue
color translucent 0.4
select *D
wireframe 100
color cpk
select *E
wireframe 100
color cpk
select *F
backbone 250
rotate z -87.58
rotate y 23.55
rotate z -62.28
zoom 50v
select HOH
cpk 150v
color white
color translucent 0.5
echo "Image is ready!"
В качестве дополнительного задания было предложено скачать файл, но другого вида, Biological Assembly (BA), просмотреть его в Jmol и найти, если есть, отличия от исходного белка. Первое отличие - формат; формат первого файла - pdb, а BA - pdb1. Запустив тот же скрипт, но заменив в load исходный файл, я получил другое изображение, которое является идентичным изображением белка. Доказательство этого приведено на рисунке 2.
Рисунок 2. Сравнение структур белка в формате Text file и Biological Assembly.
Ниже приведен список основной информации, которая может получена из .pdb файла данного белка:
- TITLE CRYSTAL STRUCTURE OF INORGANIC PYROPHOSPHATASE FROM HELICOBACTER - Название: кристаллическая структура неорганической пирофосфатазаы из Helicobacter Pylori.
- HEADER HYDROLASE 06-JAN-05 1YGZ - Заголовок: гидралаза (тип фермента), дата создания - 6 января 2005, код структуры в Protein Data Bank.
- COMPND 3 CHAIN: A, B, C, D, E, F; - Компоненты в форме цепей : 6 штук - A, B, C, D, E, F.
- COMPND 2 MOLECULE: INORGANIC PYROPHOSPHATASE; - Компненты в форме молекулы:неорганическая фосфатаза (сам белок).
- SOURCE 2 ORGANISM_SCIENTIFIC: HELICOBACTER PYLORI; - Источник: ислледованный организм Helicobacter Pylori (Геликобактер пилори).
- EXPDTA X-RAY DIFFRACTION - Способ получения: дифракция рентегновских лучей кристаллической структурой вещества.
- REMARK 2 RESOLUTION. 2.60 ANGSTROMS. - Замечания: разрешение - 2.6 ангстрема.
Данная информация получена из файла 1YGZ.pdb с помощью bash и команды egrep '^SOURCE|EXPDTA|TITLE|COMPND|REMARK' 1YGZ.pdb > Prosvirov_pr1.txt. Полученная информация отредвактирована, в виду наличия пустых ремарок и непредставляющих интереса строчек. Полную информацию можно получить в данном файле: Полная информация