Сравнение структуры белка inorganic pyrophosphatase из генома бактерии Helicobacter pylori штамм J99 и белка inorganic pyrophosphatase из Coxiella burnetii в программе Jmol
- RMDS - 0,82 Å
- Процент совпадения - 48%
- Число гэпов - 1
- Число сопоставлений Сα-атомов - 90%
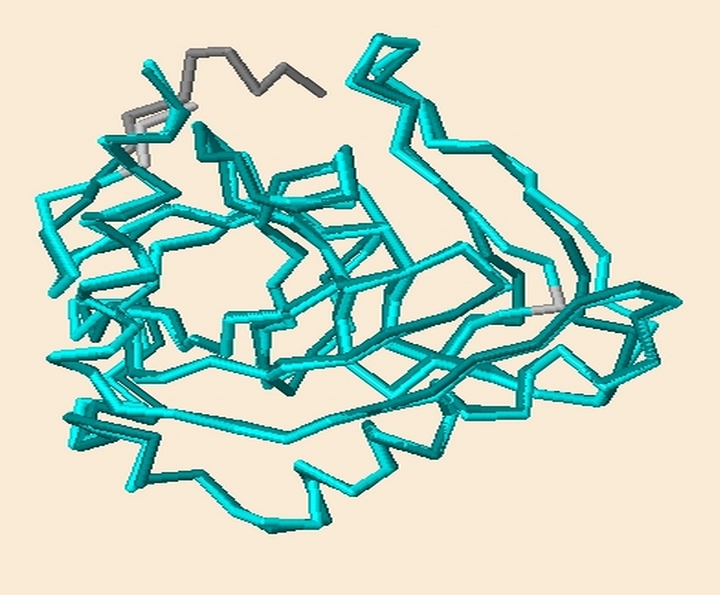
Рисунок 1. Пространственное сопастовление структур. Светлее - A цепь моего белка, темнее - D цепь белка- сравнения.
Как видно рисунка 1 совпадение ультрахорошее. Есть лишь несколько несовпадений: лишний остаток глицина и хвосты. На рисунке 2 приведено частичное сравнение вторичных структур. Как видно из рисунка цепи практически совпадают. Цепь 1YGZ:A - синяя, 3TR4:D - желтая. Красным показана область отклонения хода цепи, в данном случае это наличие лишнего глицина в цепи 3TR4:D с порядковым номером 26. Шариковой моделью показаны Сα-атомы на разных цепях, находящиеся на расстоянии меньшем 1.5Å от Сα-атома соседней цепи. Это среднее квадратичное расстояние между Сαатомами разных цепей.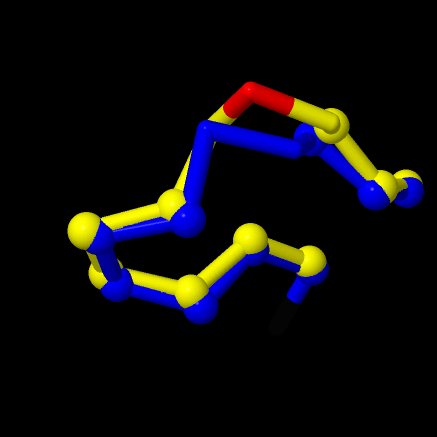
Рисунок 2. Сравнение вторичных втруктур. Описание в тексте.
Также в качестве другого задания было предложены изучить возможности Advanced Search в PDB. В качестве результаты работы привожу электронную таблицу с результатами поиска похожих белков на мой. Моими критериями были:- получение структуры с помощью дифракции ренгеновских лучей( X-ray диффракция на Вики )
- полученная структура - белок
- с 6 цепями в Biological Assembly
- организм, синтезирующий данный белок принадлежит к надклассу Proteobacteria, классу Gammaproteobacteria, подклассу Oceanospirillales
- фермент из класса гидролаз
- наличие лигандов