Работа в BLAST.
В качестве первого задания было предложено найти гомологов своего белка в базе Uniprot/SwissProt. Как видно из рис. 1, первым белок - исходный белок.
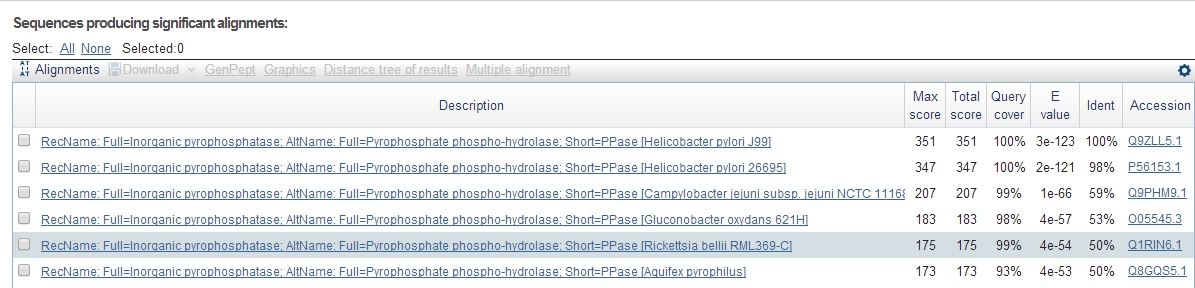
Рисунок 1. Результаты поиска гомологов белка неорганической пирофосфатазы.
Теперь я поясню, что значат различные столбцы.- Max Score - максимальный вес выравнивания одного фрагмента
- Total Score - полный вес выравнивания всех фрагментов (совпадает с Max Score, если выравнивается 1 фрагмент)
- Query Cover - процент покрытия данной последовательности и белка-гомолога
- E-value - количество ожидаемых случайных находок с таким же или, может быть, лучшим счетом
- Ident - процент совпадения (идентичности)
- Accession - AC number базы Uniprot для данного белка

Рисунок 2. Выбранный мною гомолог.
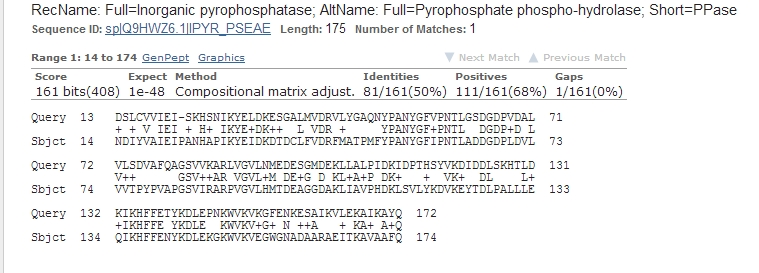
Рисунок 3. Выравнивание моего белка и белка-гомолога.
Далее было предложено построить карту локального сходства двух белков. Я скачал fasta файл гомолога. Дальше запустил все тот же pBLAST, но указав на вход Align two sequences, добавил две последовательности и в окне результата открыл вкладу Dot Matrix. Как видно из рис. 4 (карты) последовательности очень хорошо выравниваются. Из параметров выравнивания (E-value, Score и т.д.) и карты локального сходства можно с уверенностью говорить о гомологии двух белков. Это подтверждает тот факт, что это белки оба являются пирофосфатазами и назодятся у разных штаммов одного вида бактерий Helicobacter Pylori .
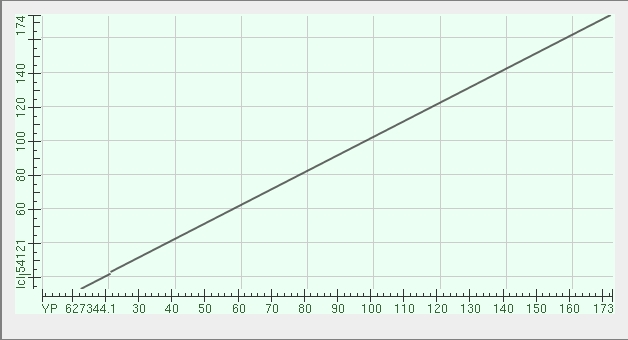
Рисунок 4. Карта сходства выравнивания.
Далее необходимо было найти гомологов своего белка среди эукариот. Для этого используя pBLAST, надо было указать в окне Organism - eurakyotes (taxid:2759). В итоге нашлось 100 эукариотных гомологов, многие из которых были либо гипотетическими и предсказанными белками. Но все имели достаточно высокий процент сходства и крайне маленький E-value. Для следующего задания надо было выбрать 5-15 гомологов для построения множественного выравнивания. Я старался выбирать белка не имеющие в название Hypothetical и Predicted .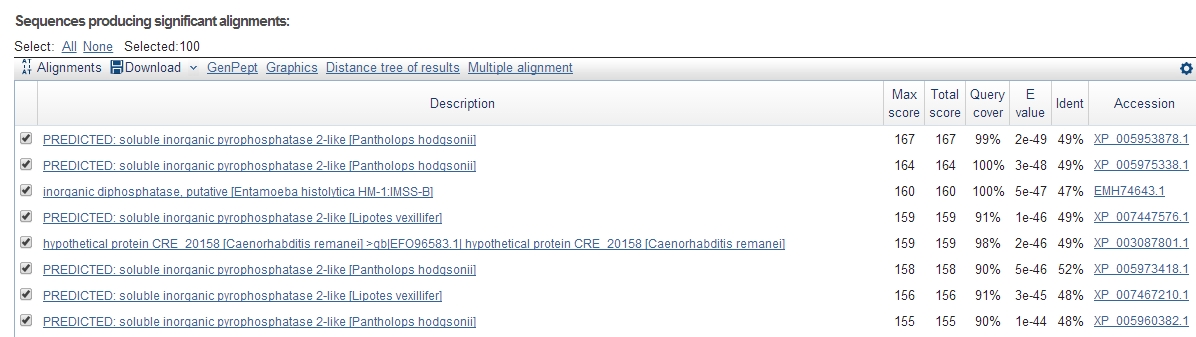
Рисунок 5. Гомологи моего белка среди эукариот.
Результат множественного выравнивания представлен на рис. 6.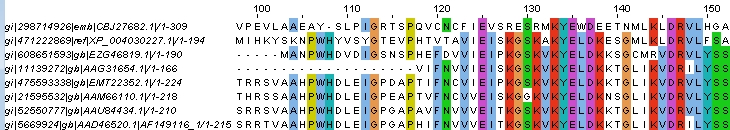
Рисунок 6. Множественное выравнивание, полученное с помощью JalView
Для выравнивания использована раскраска ClustalX, процент идентичность >70%. Как видно из выравнивания, последовательности действительно можно назвать гомологичными, так как высок процент колонок, где полностью совпадают аминокислоты всех последовательностей. Также в пользу гомологии говорят и функции данных белков все они, как минимум относятся к классу гидролаз. Скачать множественное выравнивание можно по ссылкам: jar, fasta.