Освоение навыков работы в Jalview и выравнивание последовательностей.
Для начала работы надо открыть программу, для этого нужно иметь установленный Java SE (скачать его можно на оффициальном сайте: Оффициальный сайт Oracle. Открываем программу, вставляем нужный файл для множественного выравнивания. Параметры раскраски: color - ClustalX, Above identity Threshold : 70%.
В качестве первого задания было предложено найти два блока: а) два участка, на которых для каждой колонки можно ожидать гомологию между остатками из ВСЕХ последовательностей, б)участок, на котором остатки двух или более последовательностей, предположительно, гомологичны, а остатки остальных - скорее не гомологичны им (точнее, нет данных за их гомологичность). Результаты отображены на Рисунках 1,2 и 3 соответственно. Результаты работы в формате .jar можно будет скачать в конце страницы.

Рисунок 1. Блок 1, полученный с помощью JalView, несколько вертикальных блоков (колонок), на которых можно ожидать гомологию между остатками всех последовательностей. Позиции №№306 - 341.
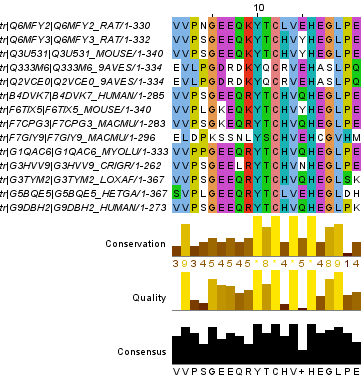
Рисунок 2. Блок 2, аналогичный блоку 1. Позиции №№380 - 400.

Рисунок 3. Блок 3. Две серые последовательности гомологичны между собой на участке 6-15,а данных относительно гомологии других нет.
Как видно из Рисунка 3, последовательности, к примеру,tr|Q333M6|Q333M6_9AVES, tr|Q2VCE0|Q2VCE0_9AVES и tr|F7GIY9|F7GIY9_MACMU имеют вставки не гомологичные таким у других последовательностей, но между собой вставки tr|Q333M6|Q333M6_9AVES и tr|Q2VCE0|Q2VCE0_9AVES похожи, что даёт нам возможность говорить об их гомлогии, но tr|F7GIY9|F7GIY9_MACMU им не гомологична.Остановимся поподробнее на блоке 1 с Рисунка 1. Можем ли мы считать позиции данного блока, имеющими общее происхождение?
Таблица 1. Таблица консервативности блока с позициями №№306 - 341 из множественного выравнивания.
| Количество | Номера позиции | Процент | |
| Общее количество позиций | 36 | 306 - 341 | 100% |
| Абсолютно консервативных позиций | 8 | 9, 24, 25, 27, 29, 33-35 | 22,2% |
| Абсолютно функционально консервативных позиций | 8 | 9, 24, 25, 27, 29, 33-35 | 22,2% |
| Консервативных на 70% позиций | 15 | 1, 2, 3, 7, 10, 11, 12, 13,17-19, 21, 23, 31, 36 | 41,6% |
| Функционально консервативных на 70% позиций | 2 | 6, 22 | 6 |

Рисунок 4. Блок 4, полученный слияние блоков и позиций между ними.
Позиций всего: 95.Позиций с гэпами: 5.
Процент: 5,2%.
В качестве следующего задания было предложено выравнять еще одну последовательность относительно полученного блока. Я использовал следующую последовательность: Последовательность . Результат отображен на Рисунке 5.

Рисунок 5. Выравнивание последовательности относительно блока вручную.
Как видно из рисунка, совпадение не случайно, и ,вероятнее всего, данный участок гомолог остальных. Консенсусная последовательность данного множественного выравнивания: ETLQRxDPPKTHVTHHPxxDGxVTLRCWALGFYPAE(получено с помощью EMBOSS.Сайт: EMBOSS). Созданное на сайте WebLogo ( WebLogo) можно посмотреть на рисунке 6.
Рисунок 6. Лого консенсусной последовательности.
В качестве следующего задания было предложено выравнивание заведомо негомологичных последовательностей. Было предложено выбрать 5-7 белков, с которыми работают другие учащиеся 1 курса ФББ. На всех рисунках в качество названия примеден AC из базы Uniprot. Задание аналогичное первому. В качестве результата приведу несколько изображений различных блоков гомологичности.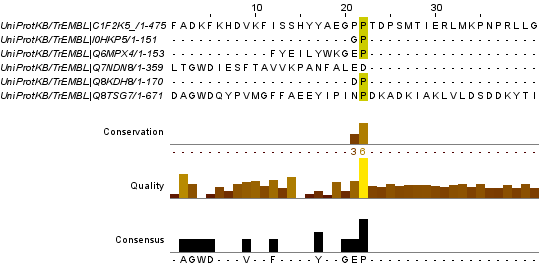
Рисунок 7.Вертикальный блок гомологичности.
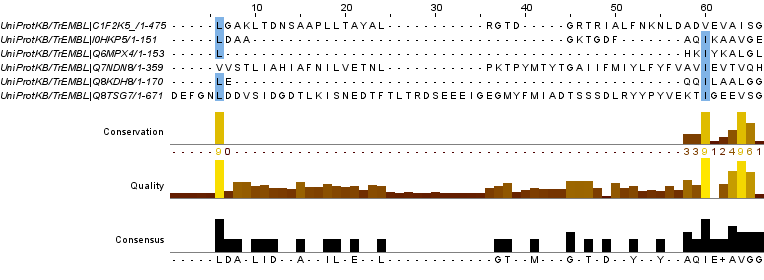
Рисунок 8.Вертикальные блоки гомологичности.
Как видно из рисунков при таких же параметрах, как в первой задаче, результат отличается. Есть всего три единичных вертикальных блока, в которых 85%+ аминокислот гомологичны. Результаты в формате .jar