Работа с Muscle.
- 90%> coverage > 70%
- ID= 50 - 60 %
- E-value < 0,00001
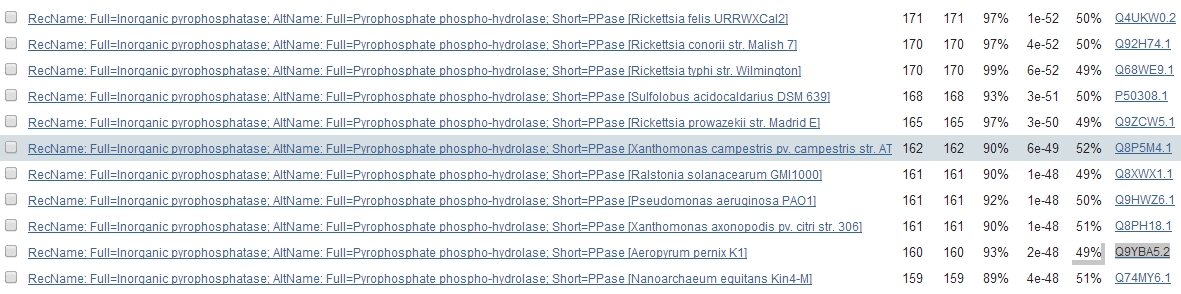
Рисунок 1. Фрагмент выходного списка последовательностей, гомологичных моему белка. Получено с помощью BLAST и базы Swiss-Prot.
Далее необходимо было, используя программу muscle на сервере Kodomo, построить выравнивание последовательностей. Я использовал следующую команду:muscle -in Homologs.fasta -out MuscleHomologs.fasta
Полученный файл можно посмотреть по ссылке: Выходной файл Затем это выравнивание было просмотрено в JalView, окраска ClustalX, Above identity treshold: 70%.

Рисунок 2. Выравнивание, полученное с помощью Muscle, в программе JalView.
В следующем задании был предложено выравнять исходные последовательности с помощью mafft. Программа также находится на сервере Kodomo, для неё команда следующая:mafft Homologds.fasta > MafftHomologs.fasta
Полученный файл можно посмотреть по ссылке: Выходной файл Затем это выравнивание было просмотрено в JalView, окраска ClustalX, Above identity treshold: 70%.

Рисунок 3. Выравнивание, полученное с помощью Mafft, в программе JalView.
Далее надо было сравнить выравнивания двумя способами.Первый способ
К одному выравнивания в JalView добавить второе и выравнять блоки друг относительно друга. Результат представлен на рис. 4, комментарии под ним.
Рисунок 4.Сравнение выравниваний друг с другом, способ первый.
Комментарий: на первый взгляд выравнивания одинаковы. Если всматриваться, можно заметить, что иногда возникают различия, но это свзяано с тем, что во втором выравнивании последовательности между собой чуть-чуть перемешаны. Из-за этого возникает иллюзия того, что оба выравнивания отличаются, хотя они идентичны.Второй способ
Второй способ заключается в объединении выравниваний с помощью Muscle. Для этого я использовал следующую команду:muscle -profile -in1 MuscleHomologs.fasta -in2 MafftHomologs.fasta -out MafftMuscleHomologs.fasta
Полученный файл можно посмотреть по ссылке: Выходной файл
Затем это выравнивание было просмотрено в JalView, окраска ClustalX, Above identity treshold: 70%.

Рисунок 5. Выравнивание,полученное в Muscle, способ второй.
Комментарий: здесь ситуация аналогичная первом способу выравнивания. По моему мнению это возникает из-за хорошей гомологичности всех последовательностей, из-за этого получается одно выравнивание, имеющее наибольший вес, и мы приходим к нему, в принципе, не зависимо от алгоритма, будь то Muscle или Mafft.Следующим задание был поиск доменов Pfam-семейств в исходной последовательности. Для этого надо было использовать сервис Pfam (Офф.сайт). В результате поиска по исходной последовательности моего белка было найдено 1 Pfam-A совпадение, приведенное на рис. 6.

Рисунок 6. Выходные данные для поиска по последовательности в Pfam.
В качестве дополнительного задания было предложено построить выравнивание с помощью других программ на выбор. Я выбрал ClustalOmega. Оффициальный сайт : ClustalO . Выходной файл выравнивания в формате clustal, можно просмотреть по ссылке: Выходной файл . На рис. 7 представлено выравнивание, полученное с помощью ClustalOmega. Параметры Jalview: раскраска ClustalX, Above Identity Treshold: 70%.

Рисунок 7. Выравнивание, полученное с помощью ClustalOmega, в программе JalView.
Для сравнения выравниваний был использован способ I.
Рисунок 8.Сравнение выравниваний Muscle и ClustalOmega, способ I. ClustalOmega - бесцветные, Muscle - серые.
Комментарий: выравнивания немного различаются. Например, 10 и 14 столбцы, - и А из выравнивания ClustalOmega, не соответствуют S и - из выравнивания Muscle. Аналогично A, N и K - из этих же столбцов. При просмотре остальных столбцом различий не было замечено.В качестве второго допонительного задания необходимо было проверить изменяет ли использованная мною ClustalOmega алгоритм, реализованный в Muscle. Так как Muscle требует на вход файлы формата fasta, необходимо перевести файла формата clustalo в fasta. Для этого использовался интернет-сервис Sequence Convertor . В checkBox выбираем protein, выбираем файл формата clustalo=> Convert=> получаем файл формата fasta. Для этого на сервере Kodomo необходимо было прописать следующую команду:
muscle -in Clustalo.fasta -out Refind_Clustalo.fasta -refine
Полученный файл можно посмотреть по ссылке: Выходной файл

Рисунок 9.Сравнение выравниваний ClustalOmega и Refined_ClustalOmega, способ I. ClustalOmega - бесцветные, Muscle - серые.
Комментарий: здесь также присутствуют небольшие различия в 10 и 14 столбцах, но в целом оба выравнивания сходны. Это говорит о том, что ClustalOmega модифицирует алгоритм Muscle.Список файлов .jar:
Для основного задания ; Для дополнительного задания