Парные выравнивания аминокислотных последовательностей.
Работа в командной строке Linux
Попытаемся создать файл, содержащий аминокислотную последовательность в fasta формате белка clpb_ecoli
и clpl_lacla. При этом последовательности "добудутся" из банка данных SwissProt
Выполняю команду:
seqret sw:p63284 –autо
seqret sw:Q06716 –autо
Появляются нужные мне файлы, содержащие а/п в fasta формате, пригодном для использования в программах из пакета
EMBOSS.
Более подробное описание и эмоции, вызванные работой с командной строкой Linux, можно найти здесь.
Строим и сравниваем оптимальные глобальное и оптимальное локальное выравнивание 2-х последовательностей
Строим полное (глобальное) оптимальное выравнивание с помощью программы needle пакета EMBOSS.
Пользуясь программой seqret, я создал файл с последовательностью белка, родственного моему (clpl_lacla – функция,
по UniProt, “ Could be the ATP-dependent specificity component of an ATP-dependent protease. - Мог быть АТФ-зависимым
специфическим компонентом АТФ-зависимой протеазы.” Белок из организма Lactococcus lactis subsp. lactis).
Далее выполняю команду needle clpb_ecoli.fasta clpl_lacla.fasta cola.needle -auto
в командной строке Linux. Полученный файл – это своего рода отчет парного выравнивания.
Потом я изменил значения по умолчанию. При повторе предыдущей команды, но опустив опцию "-auto" и дав параметр
вдвое большие значения, чем заданные по умолчанию (сообщаются как раз-таки не в квадратных скобках :-), получается
немного другое выравнивание: количество очков уменьшилось на 192, идентичность не изменилась, сходство уменьшилось на 0.8%,
гэпы(!) уменьшились на 5.2%(!).
cola.needle cola_schtr.needle
#================================ #================================
# Aligned_sequences: 2 # Aligned_sequences: 2
# 1: CLPB_ECOLI # 1: CLPB_ECOLI
# 2: CLPL_LACLA # 2: CLPL_LACLA
# Matrix: EBLOSUM62 # Matrix: EBLOSUM62
# Gap_penalty: 10.0 # Gap_penalty: 20.0
# Extend_penalty: 0.5 # Extend_penalty: 1.0
# #
# Length: 897 # Length: 872
# Identity: 310/897 (34.6%) # Identity: 302/872 (34.6%)
# Similarity: 458/897 (51.1%) # Similarity: 439/872 (50.3%)
# Gaps: 174/897 (19.4%) # Gaps: 124/872 (14.2%)
# Score: 1248.5 # Score: 1056.0
#================================ #================================
Для сравнения выравниваний мне понадобятся файлы, пригодные для импорта в GeneDoc.
Для этого выполняю команды:
needle clpb_ecoli.fasta clpl_lacla.fasta cola.msf -auto -aformat msf
needle clpb_ecoli.fasta clpl_lacla.fasta cola_schtr.msf -aformat msf
в командной строке Linux.
Строим локальное (частичное) оптимальное выравнивание с помощью программы water пакета EMBOSS
Аналогично, по той же схеме, выполняем команды:-
water clpb_ecoli.fasta clpl_lacla.fasta cola.water –auto Файл cola.water
water clpb_ecoli.fasta clpl_lacla.fasta cola_sctr.water (параметры в двое <) файл cola_sctr.water
для импорта в GeneDoc:-
water clpb_ecoli.fasta clpl_lacla.fasta cola.msf –auto -aformat msf Файл cola.water
water clpb_ecoli.fasta clpl_lacla.fasta cola_sctr.msf -aformat msf файл cola_sctr.water (параметры в двое <)
в командной строке Linux.
Сравнивание полученных выравниваний
• есть ли хотя бы один пример того, что одной и той же позиции первой последовательности в разных глобальных выравниваниях сопоставлены разные позиции второй последовательности?
Да, есть такие. При глобальном выравнивании (# Gap_penalty: 10.0 # Extend_penalty: 0.5 & # Gap_penalty: 20.0 # Extend_penalty: 1.0)
одной и той же позиции первой последовательности сопоставлены разные позиции второй последовательности. В таблице представлены некоторые из них.
| Глобальное выравнивание |
№№ позиций при # Gap_penalty: 10.0 # Extend_penalty: 0.5
“* - **” *-clpb_ecoli, **-clpl_lacla | №№ позиций при # Gap_penalty: 20.0 # Extend_penalty: 1.0
“* - **” *-clpb_ecoli, **-clpl_lacla |
| 44-18 | 44-24 |
| Gap-27 | 47-27 |
| 80-64 | 80-60 |
• есть ли хотя бы один пример того, что одной и той же позиции первой последовательности в разных локальных выравниваниях сопоставлены разные позиции второй последовательности?
Программа water, когда выводит файл в формат msf, путает номера последовательностей.
Можно только косвенно догадаться об номерах замен. Постараюсь это сделать с помощью исходных файлов .water,
представив данные в таблице.
| Локальное выравнивание |
№№ позиций при # Gap_penalty: 10.0 # Extend_penalty: 0.5
“* - **” *-clpb_ecoli, **-clpl_lacla | №№ позиций при # Gap_penalty: 5.0 # Extend_penalty: 0.25
“* - **” *-clpb_ecoli, **-clpl_lacla |
| 101-81 | 101-Gap |
| 103-83 | 103-Gap |
| 104-84 | 104-103 |
• есть ли хотя бы один пример того, что в одном глобальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск?
Да, есть - смотрите таблицу для 1 вопроса.
• есть ли хотя бы один пример того, что в одном локальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск?
Да, есть - смотрите таблицу для 2 вопроса.
• соответствуют ли оптимальные локальные выравнивания, построенными с использованием разных параметров, одним и тем же фрагментам последовательностей?
Соответствуют в некоторых местах.. получается, что особенно хорошо в конце и в середине…

• совпадают ли локальные выравнивания с соответствующими частями глобальных выравниваний?
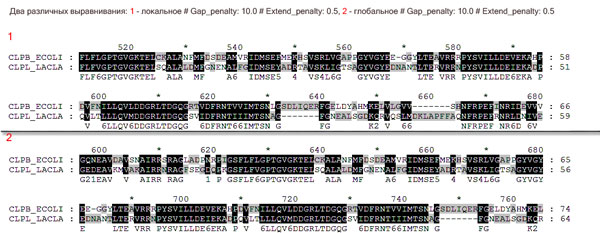
Опять-таки совпадают в некоторых местах, особенно в конце. Например кусочек из локального(1) выравнивания
clpb_ecoli: 571-583 соответствует глобальному(2) выравниванию с позицией (на рис внизу) - clpb_ecoli: 670-682.
----------------------------------------
Попытаемся построить карту локального сходства заданных последовательностей с помощью программы dotmatcher пакета EMBOSS
Для этого выполняем команду dotmatcher clpb_ecoli.fasta clpl_lacla.fasta -graph ps
в командной строке Linux.
Полученный файл dotmatcher.ps*
Конвертировал файл postscript в pdf. В конце получил картинку локального сходства: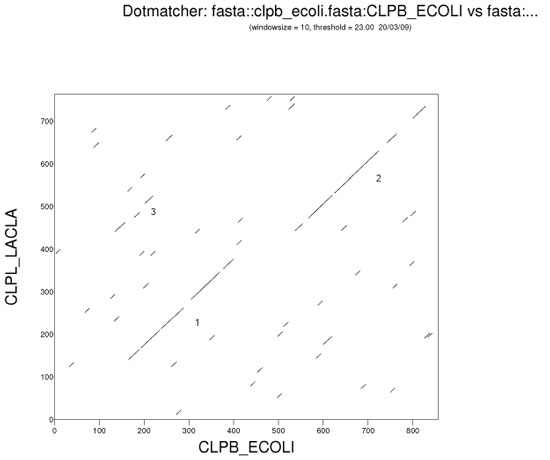
На картинке очень хорошо заметны две протяженные линии, обозначенные как 1 и 2. Возможно, это функциональные участки,
имеющие биологический смысл.
Попытаемся получить несколько субоптимальных локальных выравниваний заданных последовательностей с помощью программы matcher пакета EMBOSS.
Для этого выполняем команду matcher clpb_ecoli.fasta clpl_lacla.fasta -alt 6
в командной строке Linux.
Полученный файл cola.matcher*
Выберим из полученного файла несколько субоптимальных локальных выравниваний:
#--------
#
# Aligned_sequences: 2
# 1: CLPB_ECOLI
# 2: CLPL_LACLA
# Matrix: EBLOSUM62
# Gap_penalty: 14
# Extend_penalty: 4
#
#--------
#=======================================
#
# Length: 18
# Identity: 9/18 (50.0%)
# Similarity: 13/18 (72.2%)
# Gaps: 0/18 ( 0.0%)
# Score: 45
#
#
#=======================================
610
CLPB_E LFLGPTGVGKTELCKALA
: .: .::::: . . ::
CLPL_L LLVGESGVGKTAVVEGLA
180 190
#=======================================
#
# Length: 40
# Identity: 14/40 (35.0%)
# Similarity: 18/40 (45.0%)
# Gaps: 0/40 ( 0.0%)
# Score: 43
#
#
#=======================================
650 660 670 680
CLPB_E SVSRLVGAPPGYVGYEEGGYLTEAVRRRPYSVILLDEVEK
:: :: : : .: . .: .: :: :: :
CLPL_L SVERLTGIPVSDMGANDIEHLKNLDKRLKVMVIGEDEAVK
450 460 470 480
#=======================================
#
# Length: 26
# Identity: 10/26 (38.5%)
# Similarity: 17/26 (65.4%)
# Gaps: 1/26 ( 3.8%)
# Score: 41
#
#
#=======================================
140 150
CLPB_E ATTANITQAIEQMRGGESVNDQGAED
:: .. :..:.. : :.: :: :
CLPL_L ATIDDVAQSVERLTG-IPVSDMGAND
440 450 460
#=======================================

|