"Исследование структуры тРНК"
Краткое описание структуры в файле 1F7U.pdb
В файле приведены координаты атомов следующих молекул(молекула/кол-во):
-
ARGINYL-TRNA / 1
-
ARGINYL-TRNA SYNTHETASE / 1
Организм: SACCHAROMYCES CEREVISIAE.
Для исследования была выбрана
цепь B, представляющая аргинил-тРНК со следующей последовательностью:
[901] 5' -
UUCCUCGUGG CCCAAUGGUC ACGGCGUCUG GCUICGAACC AGAAGAUUCC
AGGUUCAAGU CCUGGCGGGG AAGCCA - 3' [976]
В последовательности на 3'-конце имеется триплет CCA, к которому присоединяется аминокислота аргинин. В документе PDB приведены
координаты ее атомов.
Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи
между азотистыми основаниями (1f7u_old.out). В соответствии с полученными данными:
- акцепторный стебель состоит из участка 901-907 и комплементарного ему участка 966-972.
- Т-стебель состоит из участка: 949-953 и компл. - 961-965
- D-стебель состоит из участка: 910-913 и компл. - 922-925
- антикодоновый стебель состоит из участка: 939-943 и компл. - 927-931
На Рис.1: Вторичная структура аргинин тРНК из SACCHAROMYCES CEREVISIAE
акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжево-желтый.
Структуру стеблевых дуплексов поддерживают 21 канонических и 4 неканонических пар оснований.
На Рис.2: Неканоническая пара гуанин-урацил(u905..g966:B)
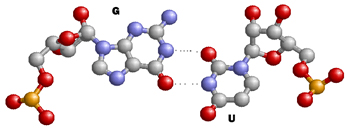
Также в структуре можно отметить отсутствие остатка тимидина в Т-петле, дигидроуридинов в D-петле и
наличие вариабельной петли(A915-A921)
На Рис.3: антикодон (UIC, где I - модфц. гуанин) в антикодоновой петле показан в шарнирной модели.
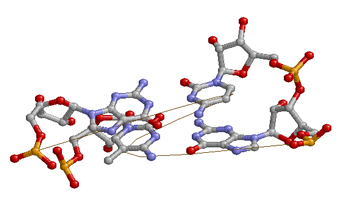
Исследование третичной структуры
1. Между основаниями конца акцепторно стебля и начала Т-стебля имеется стекинг-взаимодействия
(g965-c966/g907-5mc949 сум. интегрл перекрывания 13.35(5.58)).
На Рис.4: Cтекинг-взаимодействия Gc/GC.
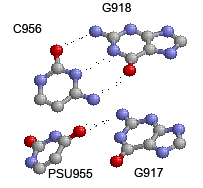
2. Имеются дополнительные водородные связи между основаниями D- и Т-петель. G918..C956 - каноническая пара и
PSU955..G917 - некаканоничеккская пара(псевдоуридин, гуанин).
На Рис.5: дополнительные водородные связи между основаниями D- и Т-петель.
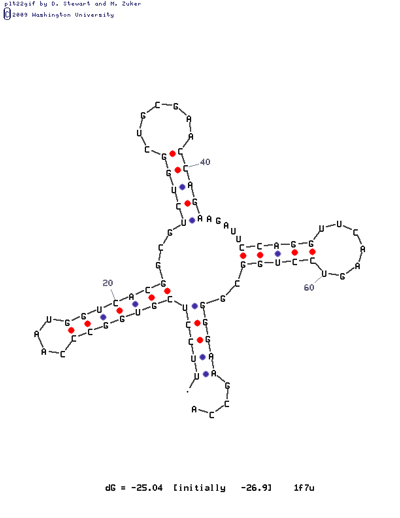
Предсказание вторичной структуры тРНК
При помощи программы mfold было полученно ряд предсказаний вторичной струтуры тРНК. Меняя значение P(Параметр, который указывает, на сколько процентов выдаваемое предсказание
структуры может отличаться по своей вычисленной энергии от оптимального.)
При P=0 и P=5, программа предлагала только одну структуру
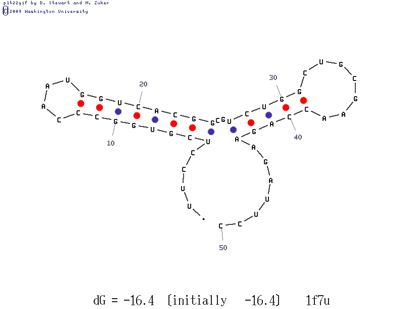
P=10 - 2 структуры, причем вторая схожа с наблюдаемой формой тРНК в Rasmol'e(дальнейшее увеличение(15%) -
увеличивает варианты резко до 7 и далее..)
Таб.1: сравнение результатов работы нескольких программ
|
Участок структуры
|
Позиции в структуре
по результатам find_pair
|
Результаты предсказания
с помощью einverted
|
Результаты предсказания
по алгоритму Зукера
|
|
Акцепторный стебель
|
901-907, 966-972
|
2-7, 66-71 (усл)
|
1-5, 68-72
|
|
D-стебель
|
910-913, 922, 925
|
0
|
6-12, 17-23
|
|
T-стебель
|
949-953, 961-965
|
49-53, 61-65 (усл)
|
49-53, 61-65
|
|
Антикодоновый стебель
|
939-943, 927-931
|
39-43, 27-31 (усл)
|
27-31, 39-43
|
Общее число
канонических пар нуклеотидов
|
21
|
16
|
22
|
Программа einverted находила стебли только при разных условиях. Для D стебля я так и не смог подобрать. Лучше всего, на
мой взгляд, справилась, конечно же, программа find_pair, потом программа mfold, которая более менее помогла определить
вторичную структуру тРНК.

|