Заданный фрагмент соответствует позициям 158-172 в полной последовательности белка GLUQ_Ecoli:
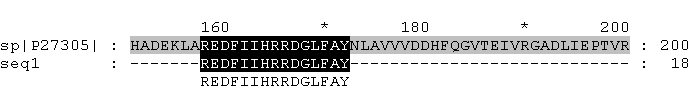
2. Построение "наилучшего" выравнивания вручную.
- исходные длины 2-х заданных фрагментов - 18 и 20
- длину выравнивания (число колонок) - 20
- вес выравнивания: W = M - nG = 13 - 1*2 = 11
- процент идентичности двух выровненных последовательностей (отношение числа колонок, в которых стоят одинаковые буквы, к общему числу колонок, включая "гэповые", умноженное на 100): 13/20*100 = 65%
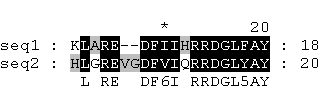
3. Поиск первой с N-конца выравнивания "близкородственной" замены а.о. Будем считать близкородственными заменами те, для которых значение элемента матрицы BLOSUM62 положительно.
- № первой позиции выравнивания, в которой наблюдается близкородственная замена а.о - 10
- полные названия и однобуквенные обозначения поменявшихся а.о: I-V (изолейцин (Ile) - Валин (Val))
- вес такой замены в соответствии с использованной матрицей: 3
Ссылки на файлы с выравниванием: alignment1.msf, alignment2.msf.
Дополнительные задания.
1. Определение % сходства выровненных фрагментов.
Процентом сходства будем считать отношение числа колонок со сходными буквами к общему числу, умноженное на 100%. Сходными буквами будем считать такие, для которых значение элемента матрицы BLOSUM62 положительно. Сходные буквы (кроме автоматически выделенных программой GeneDoc): нет. Значит, % сходства: 15/20*100 = 75%
2. Иное выравнивание 2-х заданных фрагментов с весом не хуже, чем получилось в обязательном упр.2.
Все близкородственные замены, кроме уже составленных, расположены далеко друг от друга, и сдвиг в их сторону нарушает множество других замен и совпадений, поэтому вес получается гораздо ниже. Сдвиг гэпов так же приводит к снижению веса выравнивания. Пример другого, "плохого" выравнивания:

Здесь гораздо меньше совпадений, зато остались только концевые гэпы, которые не штрафуются. Тогда вес: W = M - nG = 10 - 0*2 = 10.
3. Получение с помощью функций Excel из матрицы BLOSUM списка замен.
В приложенном файле показана работа формул на примере выравнивания, выполненного в обязательном задании №2.
Файл blosum62.xls