Объяснение кода заданного фермента (белок AMN_ECOLI).
Описание фермента AMN_ECOLI по данным International Union of Biochemistry and Molecular Biology- EC=3.2.2.4
- EC 3 Hydrolases (Гидролазы - катализируют гидролиз связи)
- EC 3.2 Glycosylases (Гликозилазы - катализируют гидролиз связи с сахаром)
- EC 3.2.2 Hydrolysing N-glycosyl compounds (Гидролизующие N-гликозидную связь)
- EC 3.2.2.4 AMP nucleosidase (АМФ нуклеозидаза)
- AMP + H2O = D-ribose 5-phosphate + adenine
- графическое изображение катализируемой реакции отсутствует
- EC=3.2.2.4
Определение метаболических путей, в которых участвует изучаемый фермент (белок AMN_ECOLI).
В документе UNIPROT с описанием заданного белка найдены имя локуса его гена.
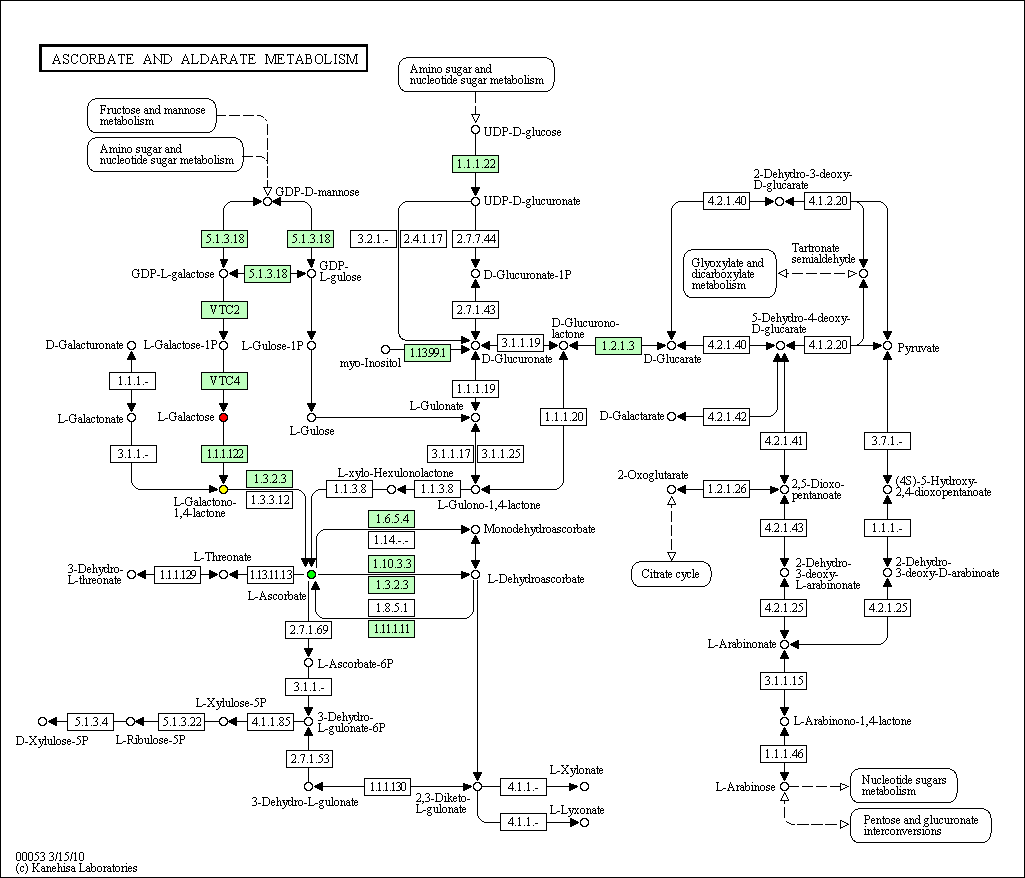
Использовались данные о моем белке на его страничке в KEGG (найден по локусу eco:b1982). Мой белок участвует в одном пути - Purine metabolism (Метаболизм пуринов). Изображение пути:
Поиск в KEGG структурных формул для L-галактозы L-аскорбата.
Поиск проводился по БД химических соединений (KEGG LIGAND).- L-галактоза (L-galactose)
- ID C01825
- структурная формула:

- L-аскорбат (L-Ascorbate; Vitamin C)
- ID C00072
- структурная формула:

- L-галактоза (L-galactose)
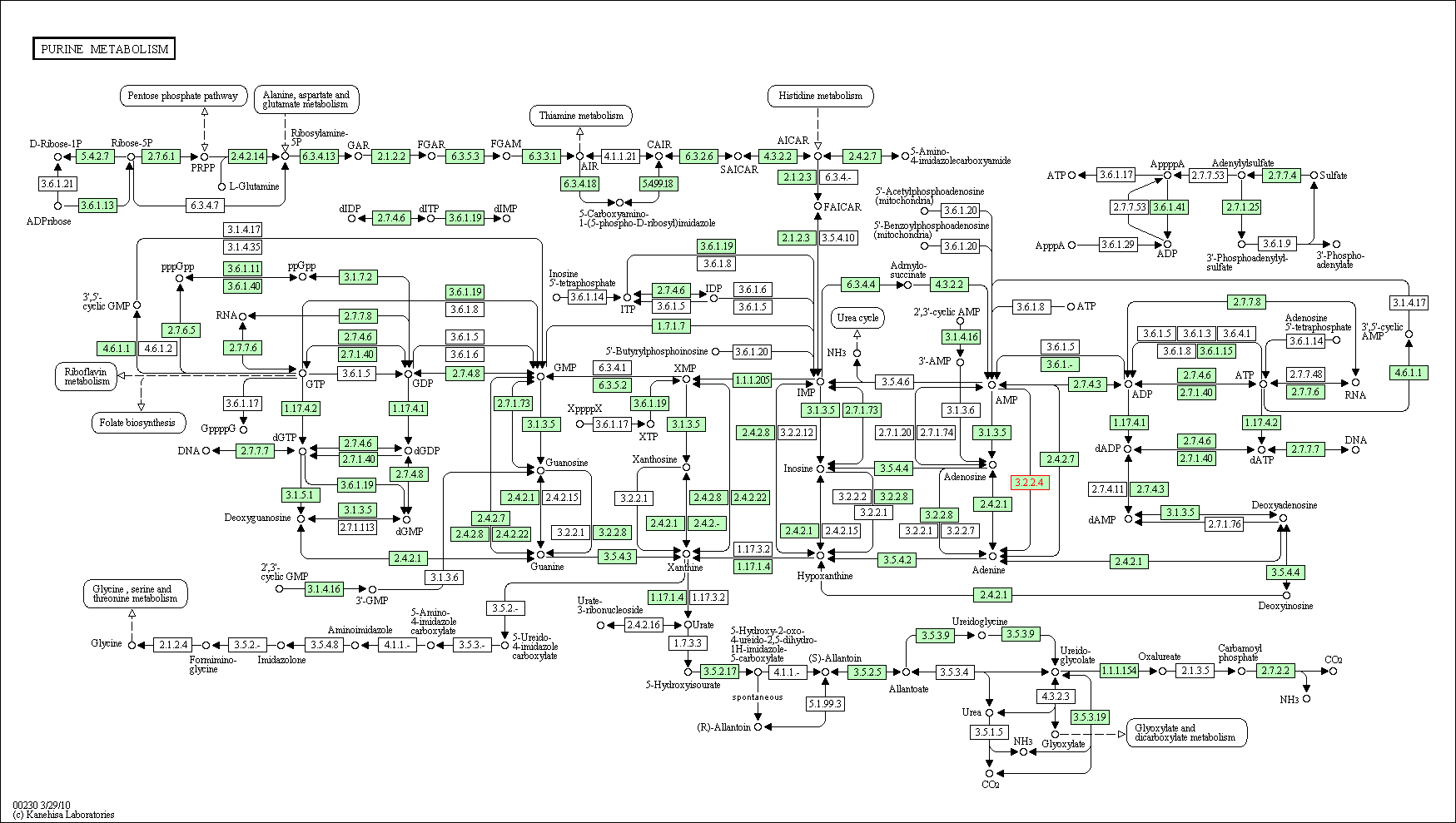
Поиск метаболического пути от одного заданного вещества к другому
Был найден один путь, задействующий оба вещества - Метаболизм аскорбата и альдарата(Ascorbate and aldarate metabolism)
Изображение пути (зеленым отмечен L-аскорбат, красным - L-галактоза, желтым - промежуточные вещества): Сравнение метаболических путей у разных организмов
Сравнение ферментов из далеких организмов
- С помощью одного запроса к SRS (маски были сняты, что особенно важно для EC - иначе могут найтись белки, у которых
номер EC отличается на лишние цифры в конце):
(([uniprot-ID:*_ARCFU] | [uniprot-ID:*_HUMAN]) & [uniprot-ECNumber:2.4.2.10])
были найдены ферменты с ЕС кодом 2.4.2.10 у человека и археи Archaeoglobus fulgidus. У этих организмов было найдено по одному такому белку (PYR5_HUMAN и PYRE_ARCFU), поэтому ссылка на UNIREF100 для отсева идентичных последовательностей не потребовалась. - По PFAM я сравнила доменную организацию найденных белков.
У человеческого белка PYR5_HUMAN - 2 домена: фосфорибозилтрансфераза и Orotidine 5'-фосфат декарбоксилаза (ядро). У белка археи PYRE_ARCFU - только один домен с функцией фосфорибозилтрансферазы. - С помощью инструментов KEGG были найдены для выбранного человеческого белка лучший ортолог
из архей, а для архейного – лучший ортолог у эукариот.
Ортолог для hsa:7372 (PYR5_HUMAN):- имя гена PAB2430
- идентичность 0.351
- длина выравнивания 171
- организм Pyrococcus abyssi (pab:)
Ортолог для afu:AF1741 (PYRE_ARCFU):- имя гена LbrM16_V2.0570
- идентичность 0.364
- длина выравнивания 154
- организм Leishmania braziliensis (lbz:)
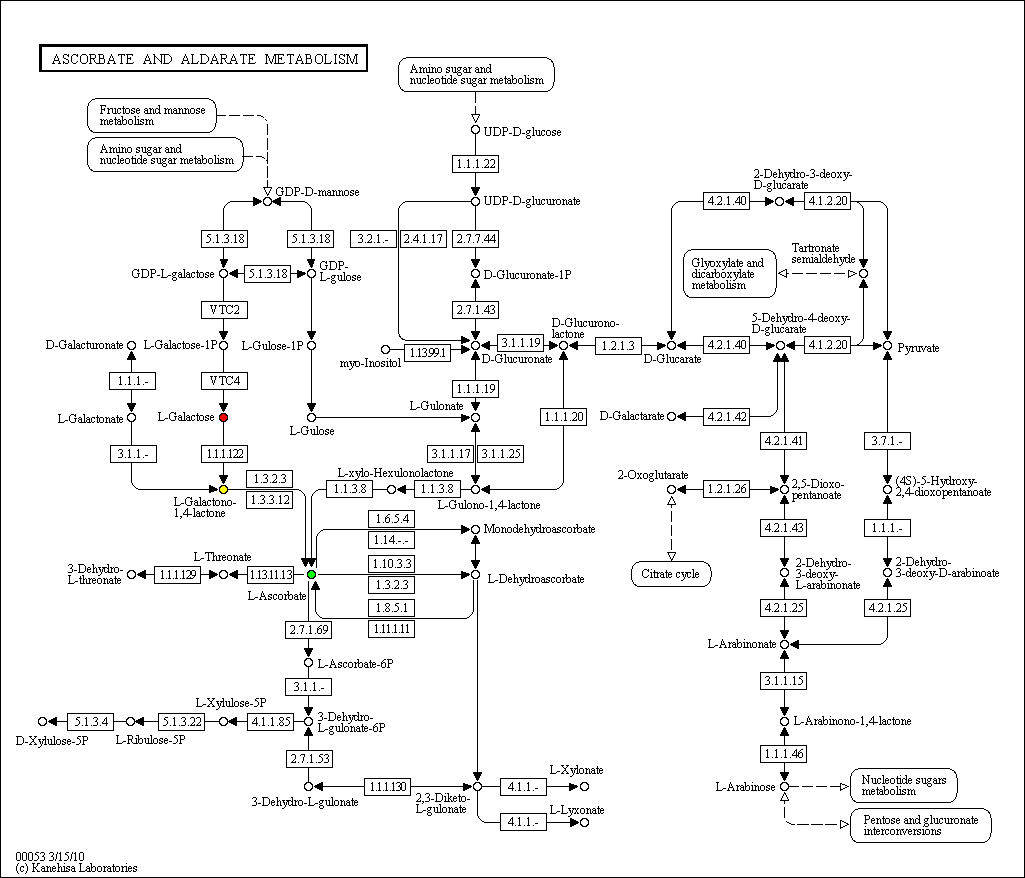
Кратчайший путь (односторонний):
L-галактоза --> L-Галактоно-1,4-лактон --> L-аскорбат (L-galactose) (L-Galactono-1,4-lactone) (L-Ascorbate)
| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | Нет | Нет генов ни для одного из возможных ферментов (1.1.1.122, 1.3.2.3, 1.3.3.12) |
| Archaeoglobus fulgidus | Нет | Не имеет такого пути вообще |
| Arabidopsis thaliana | Да | Есть гены двух возможных ферментов (1.1.1.122, 1.3.2.3) |
| Homo sapiens | Нет | Нет генов ни для одного из нужных ферментов (1.1.1.122, 1.3.2.3, 1.3.3.12) |
Карта для Arabidopsis thaliana: