Знакомство с терминологией GO
Описание функции белка с помощью аннотации Gene Ontology
Через UniProt я попала на страницу моего белка (AMN_ECOLI) в браузере QuickGO EBI.
Описание функции белка AMN_ECOLI в соответствии с GO-аннотацией
Онтология GO (название словаря) Количество разных ассоциированных терминов GO Функция белка
(краткое описание, близкое к тексту определения термина(ов) GOГде? Cellular Component - - Зачем, для чего? Biological Process 1 метаболизм нуклеозидов Молекулярный механизм? Molecular Function 3 гидролаза Специфичность? Molecular Function 3 нуклеозидаза АМФ (катализирует гидролиз АМФ до аденина) - Описание термина GO
Я выбрала термины nucleoside metabolic process, AMP nucleosidase activity и hydrolase activity, т.к. для моего белка всего 4 термина,
один из которых совсем неспецифичен (catalytic activity)
Поиск описаний выбранных терминов был проведен на главном сайте
консорциума Gene Ontology.
Изображения графов: GO:0009116, GO:0008714, GO:0016787.Описание терминов GO
GO ID выбранного термина Список синонимов Список ближайших родительских терминов GO с указанием типа связи Список ближайших дочерних терминов GO с указанием типа связи GO:0009116 nucleoside metabolism GO:0006139 nucleobase, nucleoside, nucleotide and nucleic acid metabolic process(is a) GO:0009118 regulation of nucleoside metabolic process (regulates), GO:0042278 purine nucleoside metabolic process (is a), GO:0009119 ribonucleoside metabolic process (is a), GO:0009120 deoxyribonucleoside metabolic process (is a), GO:0009163 nucleoside biosynthetic process (is a), GO:0009164 nucleoside catabolic process (is a), GO:0006213 pyrimidine nucleoside metabolic process (is a), GO:0070637 pyridine nucleoside metabolic process (is a), GO:0045978 negative regulation of nucleoside metabolic process (-ve regulates), GO:0045979 positive regulation of nucleoside metabolic process (+ve regulates) GO:0008714 adenosine monophosphate nucleosidase activity, adenylate nucleosidase activity, AMP phosphoribohydrolase activity GO:0016799 hydrolase activity, hydrolyzing N-glycosyl compounds (is a) - GO:0016787 - GO:0003824 catalytic activity (is a) GO:0051747 DNA demethylase activity (is a), GO:0003923 GPI-anchor transamidase activity (is a), GO:0008233 peptidase activity (is a), GO:0031964 beta-alanyl-histamine hydrolase activity (is a), GO:0003832 beta-alanyl-dopamine hydrolase activity (is a), GO:0019213 deacetylase activity (is a), GO:0017171 serine hydrolase activity (is a), GO:0046508 hydrolase activity, acting on carbon-sulfur bonds (is a), GO:0016788 hydrolase activity, acting on ester bonds (is a), GO:0016798 hydrolase activity, acting on glycosyl bonds (is a), GO:0016801 hydrolase activity, acting on ether bonds (is a), GO:0016810 hydrolase activity, acting on carbon-nitrogen (but not peptide) bonds (is a), GO:0016817 hydrolase activity, acting on acid anhydrides (is a), GO:0016822 hydrolase activity, acting on acid carbon-carbon bonds (is a), GO:0016824 hydrolase activity, acting on acid halide bonds (is a), GO:0016825 hydrolase activity, acting on acid phosphorus-nitrogen bonds (is a), GO:0016826 hydrolase activity, acting on acid sulfur-nitrogen bonds (is a), GO:0016827 hydrolase activity, acting on acid carbon-phosphorus bonds (is a), GO:0016828 hydrolase activity, acting on acid sulfur-sulfur bonds (is a)
Оценка качества функциональной аннотации белков в UniProt
Определение числа реальных и гипотетических белков из Cavia porcellus (Морская свинка)
Мой таксон: Домашняя морская свинка.
По Данным NCBI:Cavia porcellus Taxonomy ID: 10141 Genbank common name: Domestic guinea pig Rank: species
Соотношение между реальными и гипотетическими белками из Cavia porcellus (по данным UniProt)
Количество в UniProt Количество в SWISS-Prot Существование белка доказано экспериментально 94 90 Известны только соответствующие транскрипты 537 166 Гипотетический белок, предсказан по гомологии 103 40 Иные предсказанные гипотетические белки 69 0
Диаграмма данных таблицы:
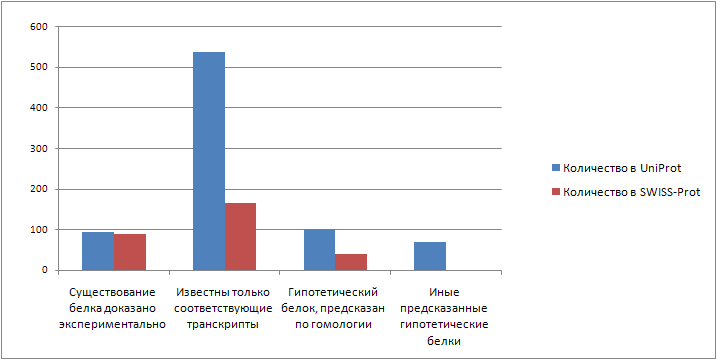
Хорошо видно, что больше всего данных - по транскриптам, меньше всего - по "иначе предсказанным" и подтвержденным экспериментально белкам. В Swiss Prot, как в наиболее проверяемой базе, находится меньше всего белков.Определение качества функциональной аннотации в UniProt
Запрос к SRS, определяющий, сколько из реальных белков Cavia porcellus аннотированы по всем трем словарям GO и у которых встречается хотя бы один раз хотя бы один из кодов экспериментального доказательства функции (EXP, IDA, IPI, IMP, IGI, IEP по данным Guide to GO Evidence Codes):
(([swissprot-NCBI_TaxId:10141*] & [swissprot-ProteinExistence:1:*]) & ([swissprot-DBxref_:GO:*] & (((((([swissprot-DBxref_:EXP*]) | [swissprot-DBxref_:IDA*]) | [swissprot-DBxref_:IPI*]) | [swissprot-DBxref_:IMP*]) | [swissprot-DBxref_:IGI*]) | [swissprot-DBxref_:IEP*]) & (((([swissprot-DBxref_:F:*]) & [swissprot-DBxref_:C:*]) & [swissprot-DBxref_:P:*]))))
По запросу найдено 4 белка.
Если из запроса убрать требование аннотированности как реального по SwissProt, будет 5 результатов.
Если, наоборот, убрать требование аннотированности как реального по GO, поиск дает 46 результатов - т. е. белки, аннотированные как реальные в SwissProt, чаще всего не аннотированы таким образом в GO.
- Использование GO для работы с массовыми данными
Получение выборки последовательностей белков с заданной функцией
среди всех терминов GO на сайте консорциума Gene Ontology был проведен поиск термина "carboxylation". В точности такого термина не было найдено, были найдены такие термины:GO:0018235 : peptidyl-lysine carboxylation (пептидил-лизин карбоксилирование) GO:0018214 : protein amino acid carboxylation (карбоксилирование белковых аминокислот) GO:0019803 : peptidyl-aspartic acid carboxylation (кабоксилирование пептидил-аспарагиновой кислоты) GO:0017187 : peptidyl-glutamic acid carboxylation (кабоксилирование пептидил-глутаминовой кислоты) GO:0050989 : N-terminal protein amino acid carboxylation (N-концевое карбоксилирование белковых аминокислот) GO:0050988 : N-terminal peptidyl-methionine carboxylation (N-концевое карбоксилирование пептидил-метионина)
(Все термины относятся к словарю biological process)
Я выбрала наиболее общий термин GO:0018214 (protein amino acid carboxylation) и провела поиск всех белков из Cavia porcellus с таким идентификатором GO.
Запросы к SRS:(([uniprot-DBxref:GO:0018214*]) & (([uniprot-Organism:Cavia*] & [uniprot-Organism:porcellus*]) | [uniprot-Organism:Cavia porcellus*]))
Поиск по обоим запросам не дал результатов (как и такой же поиск по всем найденным терминам).
Определение главной функции в большом списке белков
В файле - список белков, полученный в результате массового эксперимента. Список включает и заданный белок P0AE12. Чтобы определить – белки с какими функциями доминируют в этом списке, использовалась программа GOstat.Самые перепредставленные в выборке термины GO и их онтологии:
GO:0055086 nucleobase, nucleoside and nucleotide metabolic process (biological process) count=41; P-Value=7.46e-44; GO:0005515 protein binding (molecular function) count=52; P-Value=6.93e-38; GO:0005886 plasma membrane (cellular component) count=51; P-Value=2.86e-35; GO:0009116 nucleoside metabolic process (biological process) + count=51; P-Value=5.81e-30;
Среди перепредставленных функций нет четкой группы по P-Value, с каждой находкой рост P-Value составляет несколько порядков, поэтому порогом можно считать либо первое P-Value=7.46e-44, либо четвертое , т. к. дальше P-Value растет в основном не больше, чем на 1 порядок с каждой находкой. Между собой первые три функции не связаны, даже относятся к разным словарям: метаболизм азотистых оснований, нуклеотидов и нуклеозидов (процесс), связывание белка (функция) и плазматическая мембрана (компонент). Четвертая связана с первой, являясь более узким термином - метаболизм нуклеозидов (процесс). Из перепредставленных функций только термин GO:0009116 (метаболизм нуклеозидов) ассоциирован с моим белком.