Совмещения цепей A структур 1HY0 и 1I0A по доменам.
Домены по данным CATCH для обеих структур совпадают по номерам остатков:
Method Domain Id Fragment CATH 1HY0A1 27-123 , 195-234 1HY0A2 124-194 , 235-364 , 437-463 1HY0A3 365-436
Method Domain Id Fragment CATH 1I0AA1 27-123 , 195-234 1I0AA2 124-194 , 235-364 , 437-463 1I0AA3 365-436
Результаты совмещения в Pymol:Домен Изображение RMSD 1 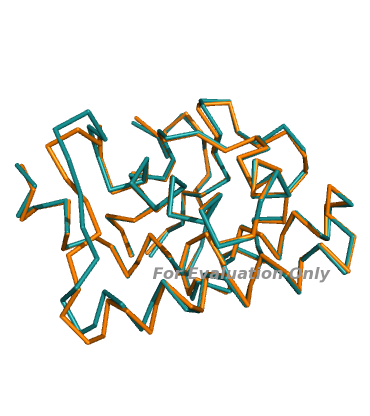
0.371 2 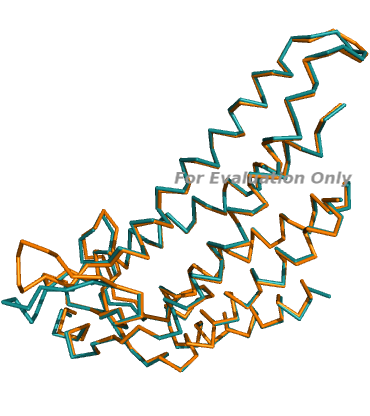
0.409 3 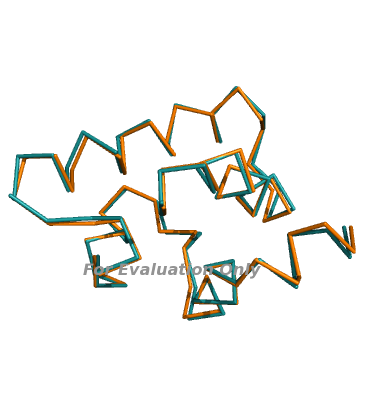
0.527
Видно, что основные структурные элементы доменов - альфа-спирали - совмещаются очень хорошо за счет жесткой структуры, а неструктурированные участки (петли) чаще всего немного не совпадают.PDBeFOLD .
Структурное выравнивание цепи A из 1AKH и цепи A из 1W0T.
По данному выравниванию с помощью сервиса Geometrical core найдено геометрическое ядро с порогом 2 Å:
Pos. 1AKH_A 1W0T_A 11 ALA83 LYS389 12 PHE84 ASN390 13 LEU85 LEU391 15 GLU87 SER393 28 LYS100 TRP403 29 GLU101 SER404 30 GLU102 LYS405 31 VAL103 ILE406 41 THR110 THR416 42 PRO111 SER417 43 LEU112 VAL418 44 GLN113 MET419 45 VAL114 LEU420 46 ARG115 LYS421 47 VAL116 ASP422 48 TRP117 ARG423 49 PHE118 TRP424 50 ILE119 ARG425 51 ASN120 THR426 52 LYS121 MET427 53 ARG122 LYS428 54 MET123 LYS429 55 ARG124 LEU430
Совмещение в PyMOL командой pair_fit структур по CA-атомам, входящим в геометрическое ядро (RMSD = 0.865):

Здесь видно, что как раз ядро взаимодействует с бороздкой ДНК, поэтому оно консервативно для двух структур:
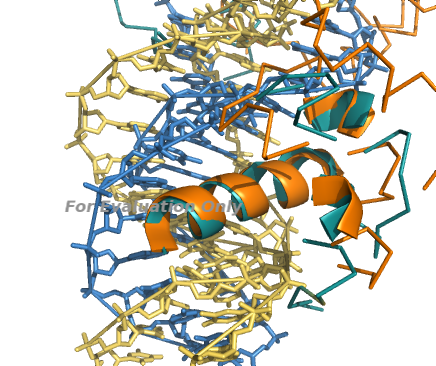
Результат команды align на полных цепях (RMSD = 1.340):
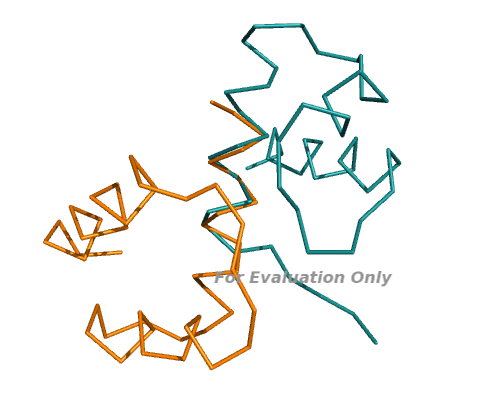
Совмещенными оказались примерно те остатки, которые входят в геометрическое ядро. Значение RMSD, однако, больше, т. е. совмещение хуже.