Классификация доменов согласно SCOP
В моем белке 1NZJ 1 домен по данным SCOP (вся цепь A):
Protein: Glutamyl-Q tRNA-Asp synthetase YadB from Escherichia coli
- Class: Alpha and beta proteins (a/b) [51349]
Mainly parallel beta sheets (beta-alpha-beta units)
Альфа и бета белки, в основном параллельные бета-листы - Fold: Adenine nucleotide alpha hydrolase-like [52373]
core: 3 layers, a/b/a ; parallel beta-sheet of 5 strands, order 32145
Укладка "Аденин нуклеотид альфа-гидролаза подобная", такую укладку имеют 3 суперсемейства - Superfamily: Nucleotidylyl transferase [52374]
Суперсемейство: Нуклеотидил трансферазы (включает 5 семейств) - Family: Class I aminoacyl-tRNA synthetases (RS), catalytic domain [52375]
contains a conserved all-alpha subdomain at the C-terminal extension
Семейство: I аминоацил тРНК-синтетазы, каталитический домен
В моем белке больше нет доменов, поэтому для описания я взяла белок с PDB ID 1Z3E. По данным SCOP он содержит два домена:
Protein: C-terminal domain of RNA polymerase alpha subunit from Bacillus subtilis (участок цепи b:245-311)
- Class: All alpha proteins[46456]
Класс: все альфа белки - Fold: SAM domain-like[47768]
4-5 helices; bundle of two orthogonally packed alpha-hairpins; involved in the interactions with DNA and proteins
Укладка: SAM-домен подобная, 4-5 спиралей, узел из двух ортогональных "альфа-шпилек" (16 суперсемейств имеют такую укладку) - Superfamily: C-terminal domain of RNA polymerase alpha subunit[47789]
contains one classic and one pseudo HhH motifs
Суперсемейство: С-концевой домен РНК-полимеразы, альфа-субъединица (содержит 1 семейство) - Family: C-terminal domain of RNA polymerase alpha subunit [47790]
Protein: Regulatory protein Spx from Bacillus subtilis (участок цепи a:1-114)
- Class: Alpha and beta proteins (a/b) [51349]
Mainly parallel beta sheets (beta-alpha-beta units)
Альфа и бета белки, в основном параллельные бета-листы - Fold: Thioredoxin fold [52832]
core: 3 layers, a/b/a; mixed beta-sheet of 4 strands, order 4312; strand 3 is antiparallel to the rest
Укладка: "Тиредоксиновая" (такую укладку имеют 2 суперсемейства - Superfamily: Thioredoxin-like [52833]
Суперсемейство: тиредоксин-подобные - Family:ArsC-like [69518]
Pfam 03960
- Class: Alpha and beta proteins (a/b) [51349]
Классификация доменов согласно CATH.
CATH, в отличие от SCOP, выделяет в моем белке 1NZJ два домена:

Однако оба домена еще не классифицированы (CATH пишет "This domain is in the HOMCHECK_REVIEW flow stage and therefore has not yet been assigned to a homologous superfamily in CATH.")
В белке 1Z3E домены выделены так же, как в SCOP (но классификация для одного из них так же недоступна):
Домен 1: цепь A, остатки 0-118
Домен 2: цепь B, остатки 245-311. CATH Code: 1.10.150.20.2.1.1.1.1
- Классификация:
- Класс: 1 Mainly Alpha
- Архитектура: 1.10 Orthogonal Bundle
- Топология: 1.10.150 DNA polymerase; domain 1 (23 суперсемейства)
- Суперсемейство: 1.10.150.20 5' to 3' exonuclease, C-terminal subdomain (25 семейств)
Остальные уровни классификации имеют только номер.
Отличия SCOP и CATH.
Мне попались домены, описанные в SCOP, но не классифицированные в CATH.
Сравним данные по SCOP и CATH для С-концевого домена 1Z3E b:245-311, который классифицирован в обеих базах.
- Класс - в обеих базах альфа белки.
- Архитектура - только в CATH
- Укладка - ортогональный узел по обеим базам
- Суперсемейство - С-концевой домен; РНК-полимеразы по SCOP, экзонуклеазы - по CATH.
- границы в цепи - совпадают
Выравнивание доменов из одной топологии.
Топология (по CATH) Porin, два домена из разных суперсемейств:- Класс: 3 Alpha Beta
- Архитектура: 3.30 2-Layer Sandwich
- Топология: 3.30.70 Alpha-Beta Plaits
- Суперсемейство: 3.30.70.1110 CheY-binding domain of CheA. Chain A
- 3.30.70.1110.1.1.1.1.1
- PDB ID 1u0s chain A 175-260
- Суперсемейство: 3.30.70.1230 Adenylyl Cyclase, chain A
- 3.30.70.1230.1.1.1.1.1
- PDB ID 1fx2 chain A 888-1122
По данному выравниванию с помощью сервиса Geometrical core найдено геометрическое ядро с порогом 2 A:
| Pos. | 1FX2_A | 1U0S_A |
| 14 | THR901 | LYS177 |
| 15 | LEU902 | THR178 |
| 16 | ILE903 | PHE179 |
| 17 | PHE904 | TYR180 |
| 18 | THR905 | ILE181 |
| 43 | ARG930 | TYR197 |
| 46 | ARG933 | PHE200 |
| 47 | SER934 | HIS201 |
| 50 | GLY937 | GLU204 |
| 51 | ARG938 | GLU205 |
| 52 | TYR939 | LEU206 |
| 53 | LYS940 | LYS207 |
| 54 | CYS941 | CYS208 |
| 55 | TYR942 | GLU209 |
| 56 | GLU943 | VAL210 |
| 57 | VAL944 | VAL211 |
| 58 | LYS945 | ARG212 |
| 59 | THR946 | THR213 |
| 60 | VAL947 | ILE214 |
| 75 | ASP949 | VAL229 |
| 76 | SER950 | GLU230 |
| 77 | PHE951 | LEU231 |
| 78 | MET952 | PHE232 |
| 79 | ILE953 | VAL233 |
| 80 | ALA954 | ILE234 |
| 93 | GLU967 | ALA245 |
| 94 | LEU968 | LEU246 |
| 101 | HIS975 | ALA250 |
| 147 | ARG1020 | ARG254 |
| 149 | ARG1022 | ILE256 |
| 150 | VAL1023 | ILE257 |
| 151 | GLY1024 | LYS258 |
| 152 | ILE1025 | GLU259 |
Скрипт для PyMOL.
Полученное в PyMOL изображение наложения геометрического ядра:
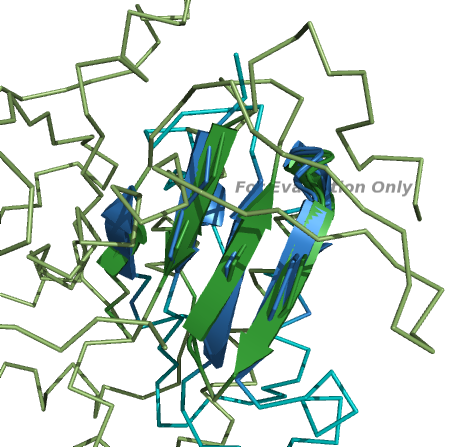
Видно, что у доменов из одной топологии действительно есть очень похожие элементы.