Подготовка файла координат и файла топологии.
В прошлом занятии был предоставлен gro файл с 38 молекулами этана. Создадим индекс файл котором будет группа из одной молекулы этана.make_ndx -f box_38.gro -o 1.ndx
Теперь создадим gro файл с одной молекулой и зададим ячейку.editconf -f box_38.gro -o et1.gro -n 1.ndx #зададим ячейку и расположим молекулу по центру ячейку editconf -f et1.gro -o et.gro -d 2 -c
Исправим файл топологии et.top из прошлого задания. В разделе [ molecules ] изменим количество молекул этана.Даны 5 файлов с разными параметрами контроля температуры:
be.mdp - метод Берендсена для контроля температуры.
vr.mdp - метод "Velocity rescale" для контроля температуры.
nh.mdp - метод Нуза-Хувера для контроля температуры.
an.mdp - метод Андерсена для контроля температуры.
sd.mdp - метод стохастической молекулярной динамики.
Скрипт по работе с 5ю системами: scr.bash Содержание скрипта:Сначала надо построить входные файлы для молекулярно-динамического движка mdrun с помощью grompp:
grompp -f ${i}.mdp -c et.gro -p et.top -o et_${i}.tpr # где i: be,vr,nh,an,sd см. выше список mdp файловДолжно получиться 5 tpr файлов. Теперь для каждого из них запустим mdrun.
mdrun -deffnm et_${i} -v -nt 1Теперь переходим к анализу результатов. Начнем с визуального анализа. Для каждой из 5 систем проведем конвертацию в pdb.
trjconv -f et_${i}.trr -s et_${i}.tpr -o et_${i}.pdbПолученные pdb-файлы:
be.pdb - молекула этана очень быстро вращается по оси С-С и поворачивается относительно середины этой связи. Заметны небольшие изменения длины связи С-С.
vr.pdb - молекула изменяется по-разному: то активно вращается по С-С связи, то начинает изменяться длина связи. Это не выглядит правдоподобным.
nh.pdb - молекула вращается вокруг своей оси. Видимого изменения длины связи нет.
an.pdb - молекула лишь немного "подрагивает" - есть изменение длины С-С связи, нет вращения. Похоже на поведение при низких температурах.
sd.pdb - молекула хаотично движется, есть вращение, изменение длины связи сложно проследить.
Сравним потенциальную энергию связи (Bond) и кинетическую энергию (Kinetic En.)для каждой из 5 систем.
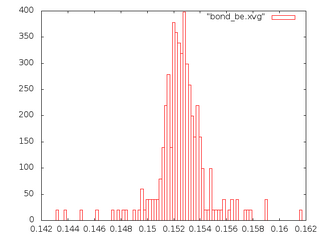
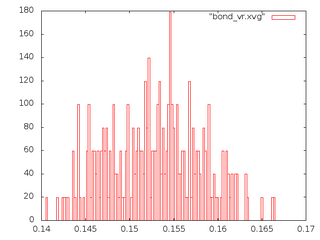
g_energy -f et_${i}.edr -o et_${i}_en.xvgРассмотрим распределение длинны связи С-С за время моделирования. Сначала создадим индекс файл с одной связью. В текстовом редакторе создадим файл b.ndx со следующим содержимым:
[ b ] 1 2
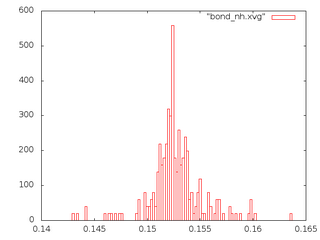
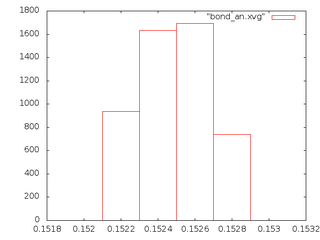
И запустим утилиту по анализу связей g_bond:g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
- Графики потенциальной энергии связи и кинетической энергии (слева, красным - потенциальная, зеленым - кинетическая) и распределения С-С связи (справа) для 5 систем:
Метод Энергии С-С be 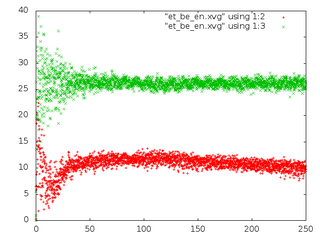
vr 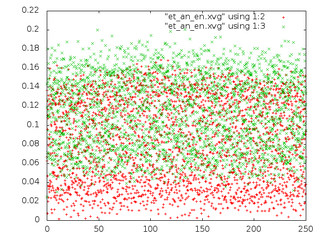
nh 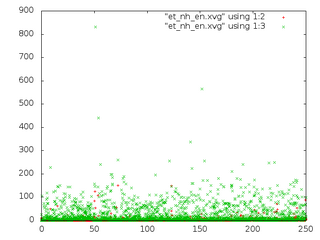
an 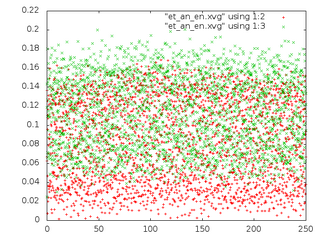
sd 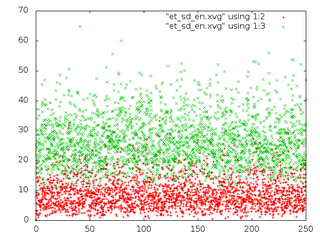
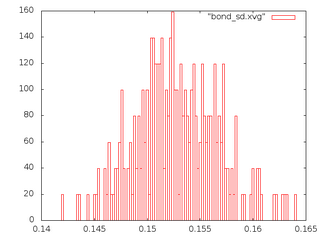
Сравнивая форму распределения Больцмана и графики выше можно заметить, что график распределения связей sd (Стохастическая молекулярная динамика) больше всего похож на график больцмановского распределения, и, возможно, этот метод позволяет наиболее реалистично поддерживать температуру в системе. Но в общих чертах ни один из методов не предлагает распределение, принципиально отличающеся от Больцмановского.