


MEME - это алгоритм поиска мотивов в биологических последовательностях белков и ДНК. С его помощью можно искать сайты связывания транскрипционных факторов у прокариот.
Исходными данными являлись файл с последовательностями промоторных областей некоторых генов E.coli и список экспериментально установленных сайтов связывания белка PurR.
При запуске MEME использовался режим Normal mode. В параметрах поиска была выставлена длина мотива равная 16 нуклеотидам. Запуск проводился два раза, в первом случае с параметром "One per sequence", во втором - "Zero or one per sequence" (поле "How do you expect motif sites to be distributed in sequences?" ). Выполнялся поиск одного мотива в каждой последовательности (поле "How many motifs should MEME find?") и была выставлена длина мотива в 16 нуклеотидов (поле "How wide can motifs be?").
В результате было получено несколько файлов, содержащих информацию о предположительных сайтах связывания PurR. Анализировались файлы в формате txt: meme_oops.txt для "One occurence per sequence" и meme_zoops.txt для "Zero or one occurence per sequence". Стоит отметить, что найденные мотивы и p-value в обоих файлах полностью идентичны.
Экспериментальные данные и предсказания были отмечены на последовательностях и сохранены в файле findings.doc. В первой строке каждой последовательности отмечена информация о том, на какой цепи был найден мотив, а так же опциональная информация для мотивов с p-value более 1.0e-4, которые не были отмечены в самих последовательностях. В файле синим отмечены сайты, установленные экспериментально, курсивом - предсказанные с параметром "One occurence per sequence" (11 мотивов, но 3 из них с p-value больше 10-4), а жирным шрифтом - с параметром "Zero or one occurence per sequence" (11 мотивов, но 3 из них с p-value больше 10-4).
Все найденные мотивы либо полностью совпали с предсказанными эксперементально, либо (для glnB и purA) были найдены точно, но с p-value более 1.0e-4, либо для folD был найден мотив de novo, но с p-value более 1.0e-4.
RegPredict - это веб сервер, разработанный для реконструкции и анализа регулонов в прокариотических геномах, использующий подходы сравнительной генмики. Статья о веб-сервисе вышла в 2010 году в журнале Nucleic Acid Res., в соавторах статьи - сотрудники факультета биоинженерии и биоинформатики, поэтому я остановилась именно на этом сервисе.
Регулон — это система генов, разбросанных по всему геному, но подчиняющихся общему регуляторному белку. Сервис имеет два модуля:
1) Поиск регулона на основании известной PWM. Принимает на вход следующие данные: позиционную матрицу (PWM) для исследуемого регуляторного фактора и набор близкородственных видов. На первой стадии выявляются потенциальные гены, перед которыми содержится сайт транскрипционного фактора. На второй стадии выявляются ортологичные гены внутри одного организма и между организмами. На основании полученных данных делается предсказание о регулонах в геномах.
2) De novo поиск регулона. Принимает на вход следующие данные: набор функционально связанных генов - потенциальных кандидатов, входящих в регулон. Этот модуль позволяет de novo выявить наличие мотивов в upstream области генов (перед геном), если таковые имеются и отсортировать их по информационному содержанию. Каждый мотив может быть протестирован с помощью модуля, описанного в п.1.
Для изучения работы сервиса мной был запущен пример, доступный для исследования - прекалькулированный список кластеров ко-регулируемых ArgR ортологичных оперонов для семейства гамма протеобактерий Shewanella. Пример иллюстрирует работу сервиса при de novo поиске регулонов. Были выбраны первые несколько ко-регулиремых оперонов, выбрана наиболее подходящая матрица мотива, и на основании этих данных были получены регулоны. Скриншоты графического интерфейса вы можете видеть на рис. 1 и 2.
Рис. 1. Выбор модуля
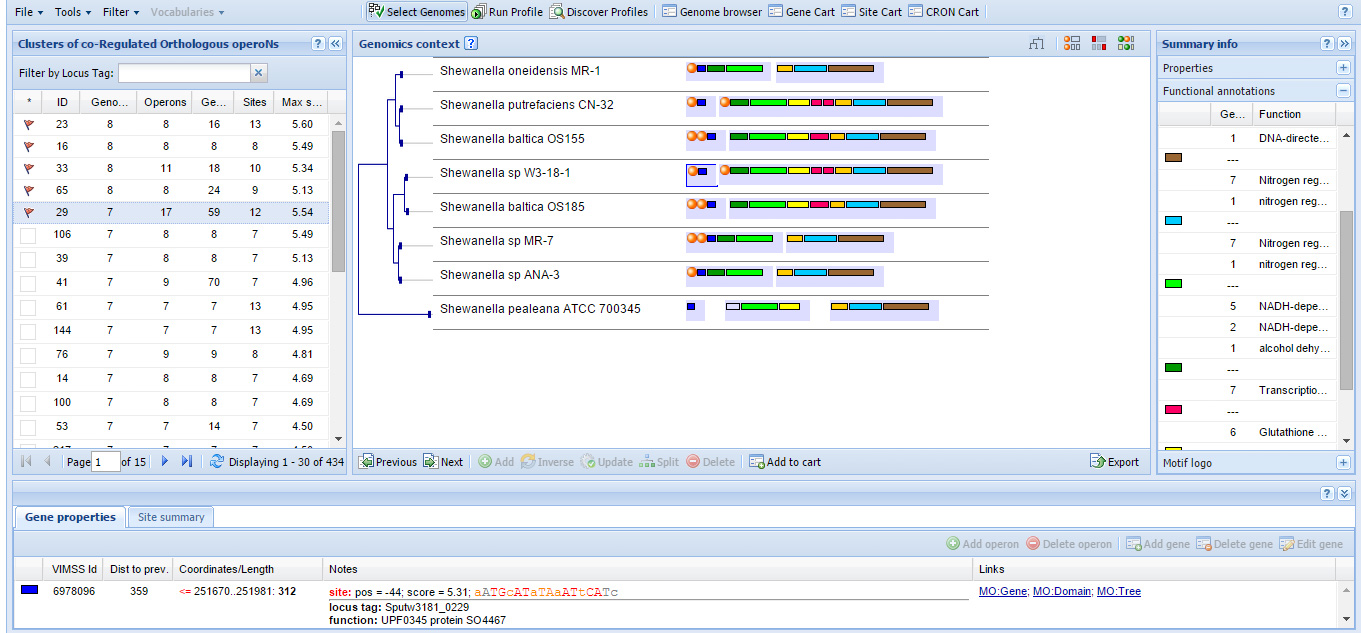
Рис. 1. Полученный результат