| Главная | Семестры | Обо мне | Ссылки |
|---|
Поиск регуляторных мотивов транскрипции в бактериальных последовательностях
В этом задании было необходимо найти регуляторный мотив (набор сайтов) в полученных последовательностях с помощью программы MEME.
Файл с последовательностями: загрузить
Файл с экспериментальными данными о мотивах: загрузить
Длина данных мне последовательностей 100 нуклеотидов, всего 13 последовательностей. Поиск был проведен с помощью MEME. Параметры: ‘Minimum length’ = 16; ‘Maximum length’ = 16; ‘Maximum number of motifs to find’ = 1. Программа запускалась дважды с разными количествами ожидаемых мотивов: ‘One per sequence’ или ‘Zero or one per sequence’. На рис. 1 показаны последовательности с выделенными мотивами.
Файл с размеченным выравниванием в формате .docx: загрузить

Рис. 1.Мотивы в последовательностях. Синим выделены экспериментально установленные сайты, серым – предсказанные MEME. Курсивом выделены мотивы, найденные MEME с параметром ‘One occurrence per sequence’, жирным – найденные MEME с параметром 'Zero or one occurrence per sequence'
Из полученных данных следует, что мотивы, предсказанные MEME с параметром ‘One occurrence per sequence’ полностью совпадают с экспериментальными. Лого найденного мотива показано на рис. 2. При заданном параметре ' Zero or one occurrence per sequence' мотив найден на обратной цепи и сдвинут относительно экспериментального на 1 нуклеотид вправо для прямой цепи и влево для обратной. На рис. 3 приведен лого этого мотива, а для наглядности на рис. 4 – лого обратно-комплементарного ему сайта. Если сравнить рис. 2 и рис. 4, то видно, что они отличаются сдвигом на 1 нуклеотид.
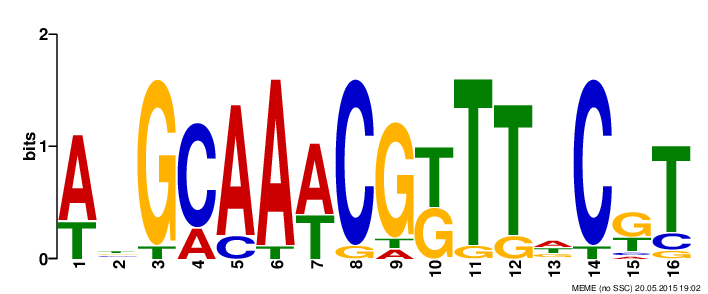
Рис. 2. Лого мотива, установленного экспериментально и найденного MEME с параметром ‘One occurrence per sequence’

Рис. 3. Лого мотива, найденного MEME с параметром 'Zero or one occurrence per sequence' на прямой цепи

Рис. 4. Лого мотива, найденного MEME с параметром ' Zero or one occurrence per sequence' на обратной цепи
Для трех последовательностей, у которых мотивы не подтверждены экспериментально, найдены мотивы при параметре ‘One occurrence per sequence’, а для двух из них – и при параметре ' Zero or one occurrence per sequence' . Но если для folD найденные мотивы различаются на один нуклеотид, то у rpiA мотивы перекрываются всего на 3 из 16 нуклеотидов, а для carA мотив обнаружен только при первом параметре. Мне кажется, что в случае folD можно утверждать о наличии мотива, а в случае rpiA и carA нельзя.
Сервис PePPER Prokaryote promoters
Работа выполнена вместе с Мошенским Денисом. Ссылка на его страницу: http://kodomo.fbb.msu.ru/~loven-doo/term4/pr9.html
PePPER - это веб-сервер, разработанный для поиска в ДНК прокариот сигнальных последовательностей: промоторов, регулонов и сайтов связывания транскрипционных факторов. Оригинальная статья разработчиков находится в открытом доступе. Ее можно скачать здесь. Для подробного описания был выбран сервис Prokaryote promoters.
Известно, что у прокариот существуют консервативные участки в области промотора на -35 и -10 позициях. В данном сервисе поиск этих консервативных последовательностей проводится по отдельности. Допустимая длина спейсера между ними составляет 16-18 нуклеотидов. Не очень понятно, на основании чего были выбраны такие значения длины спейсера, ссылок на другие публикации авторы не приводят. Соответственно, длина последовательности на выходе составляет 28-30 нуклеотидов. Также эта программа способна предсказывать промоторы, у которых есть только -35 или только -10 последовательность.
В данном алгоритме не производится поиск сайтов связывания сигма-факторов, хотя они тоже относятся к области промотора. Авторы выделили поиск этих сайтов в отдельный сервис.
Интерфейс сервиса Prokaryote promoters показан на рис. 5. На вход программа принимает последовательность ДНК, без формата, либо в формате FASTA. Можно подать несколько последовательностей, тогда поиск промоторов будет осуществлен в каждой из них независимо. Поэтому если требуется, например, найти промоторы в нескольких последовательностях из одного генома, то их нужно объединить в одну. Второе окно сервиса предназначено для поиска промоторов между известными генами и принимает на вход таблицу генов. Необходимые данные: название гена (видимо, условное обозначение гена, для того чтобы привязать к нему промотор), старт, конец, ориентация ("+" или "-"). По-видимому, это окно не работает, так как при вводе в него данных в указанном формате программа выдает такой же результат, что и без них.
Рис. 5. Стартовая страница сервиса Prokaryote promoters
В результате работы программы выводится таблица, фрагмент которой представлен на рис. 6. Колонки таблицы: номер последовательности, ее описание из fasta файла, сама последовательность, координаты начала и конца (в прямом направлении), предсказанный старт транскрипции, название найденного мотива, ориентация, длина последовательности и вес. Также эту таблицу предлагается скачать в виде текстового файла.
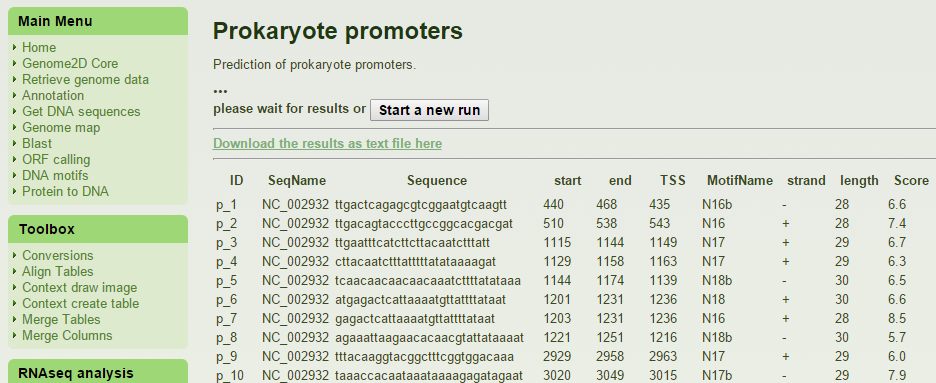
Рис. 6. Результат работы программы PePPER prokaryote promoters. Представлен фрагмент таблицы, содержащий первые 10 предсказанных последовательностей
При запуске данной программы на целом геноме бактерии даже предсказанные промоторы с большим весом находились на больших расстояниях от ближайших генов (> 200 нуклеотидов). Это является слишком большой длиной 5' нетранслируемой области для прокариот. Предсказание промоторов является очень сложной задачей, так как связывающая -10 последовательность может быть расширена до 8 нуклеотидов: добавляются два, со стороны старта трансляции, а -35 область тогда вообще не связывается. К тому же есть разные факторы, делающие связывание с -35 областью неспецифичным.
Также программа была протестирована на объединённых последовательностях из одного генома длинной 100 нуклеотидов каждая, находящихся перед старт-кодонами генов с Uniprot protein evidence 1. В результате для входных 39 последовательностей было предсказано 4 промотора. Такой результат может быть связан с оперонами, в которых не каждый ген имеет свой промотор. Однако объяснением такого результата может являться, возможно, ошибочный расчет веса, что влечет за собой отбрасывание правильных вариантов. Еще стоит принять во внимание установленные пороги для длин спейсеров, так как в некоторых организмах они могут быть другими. Правильнее было бы, если пользователь их мог задавать сам. Тогда при запуске этой программы несколько раз с разными порогами, возможно, можно было бы получить более точные результаты.