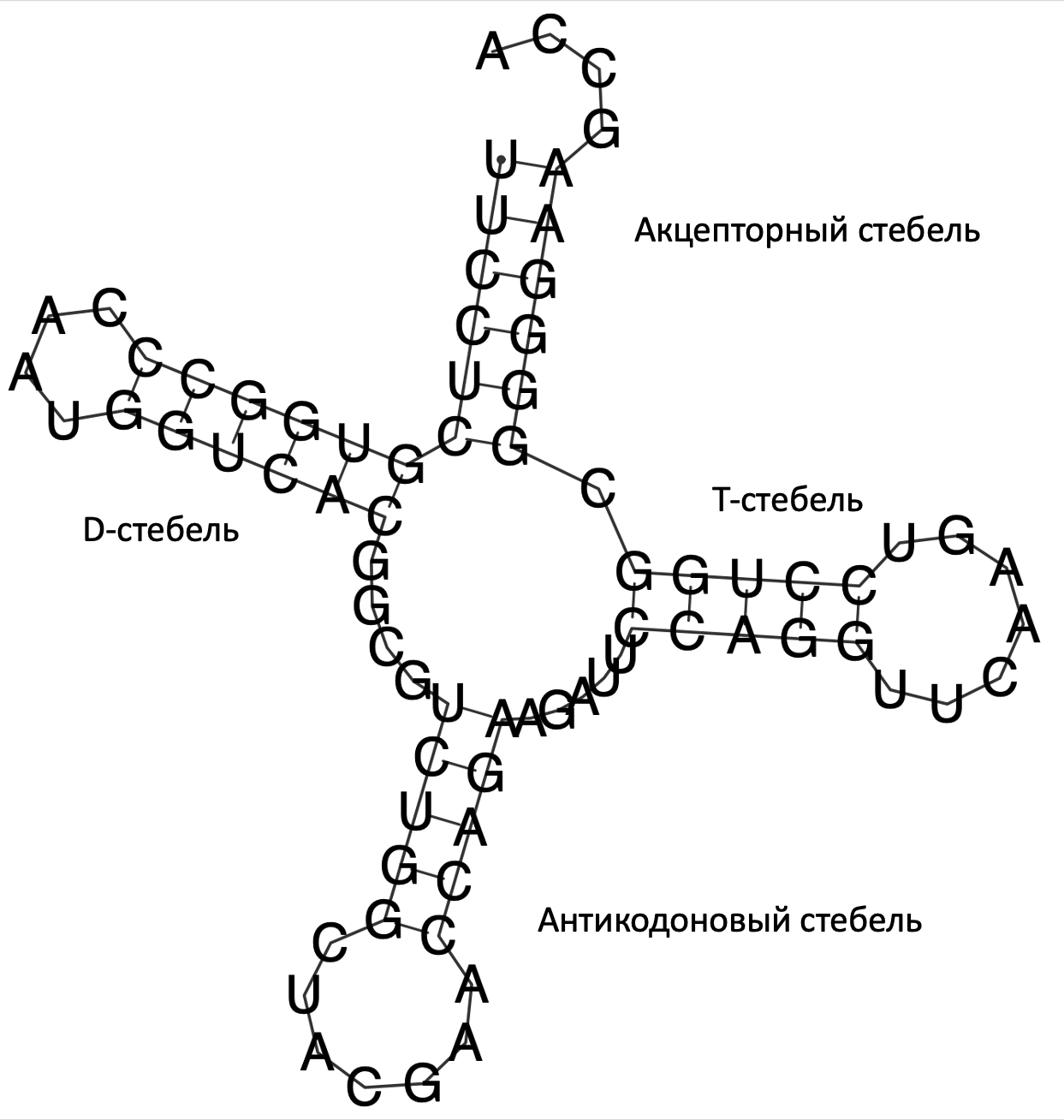
С помощью алгоритма Зукера было получено следующее изображение (справа рис 1).
В таблице 1 (ниже) приведено сравнение результатов работы трёх программ.
Для предсказания инвертированных участков тРНК с помощью программы einverted была скачана последовательность тРНК в формате fasta.
В последовательности был неканонический нуклеотид I (инозин), и программа einverted не работала. Поэтому была совершена замена I на похожий на него А.
tatyana.ponomareva@kodomo:~/term3/block1/pr2$ einverted rcsb_pdb_1F7U.fasta
Были подобраны следующие параметры:
Gap penalty [12]: 10
Minimum score threshold [50]: 0
Match score [3]: 3
Mismatch score [-4]: -4
Результат можно найти здесь
Найденные стебли совпадают с выдачей программы find_pair, однако einverted однозначно менее удобная в использовании.
| С помощью алгоритма Зукера было получено следующее изображение (справа рис 1). В таблице 1 (ниже) приведено сравнение результатов работы трёх программ. |

|
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 901-907, 966-972 (7 пар) | - (не смог найти нужные пары) | 6 из 7 |
| D-стебель | 949-953, 961-965 (5 пар) | 5 из 5 предсказано | 4 из 5 |
| Антикодоновый стебель | 939-944, 926-931 (6 пар) | 5 из 6 пар предсказано | 5 из 6 |
| T-стебель | 910-913, 922-925 (4 пары) | - | 2 из 4 |
| Общее число канонических пар нуклеотидов | 17 | 10 предсказано | 17 предсказано |
Программа nucplot не работала для заданного файла 1TRO, поэтому я взяла другой белок 1HW2. Файл pdb был переведен в нужный формат с помощью команды:
tatyana.ponomareva@kodomo:~/term3/block1/pr4$ remediator --pdb --old 1hw2.pdb > 1hw2_old.pdb
Далее программа выдала следующую визуализацию контактов между ДНК и белком (рис 2 и 3).
nucplot 1hw2_old.pdb
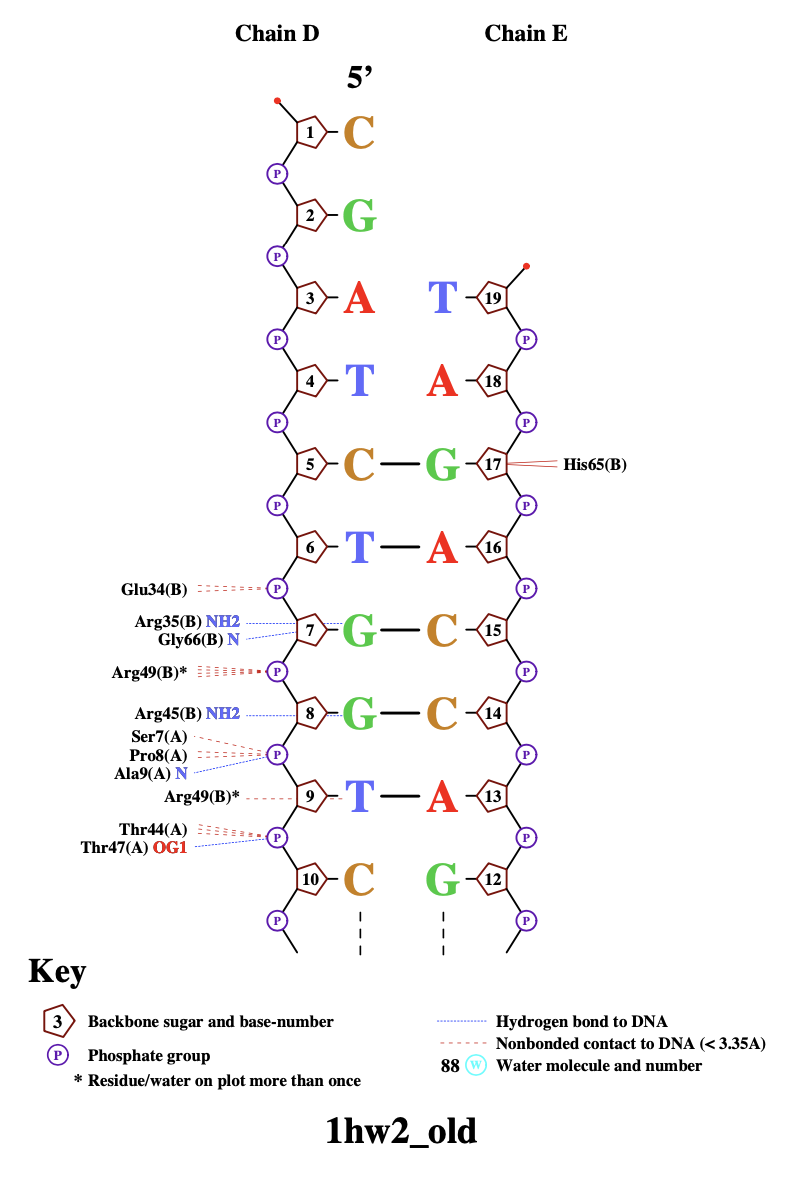
|
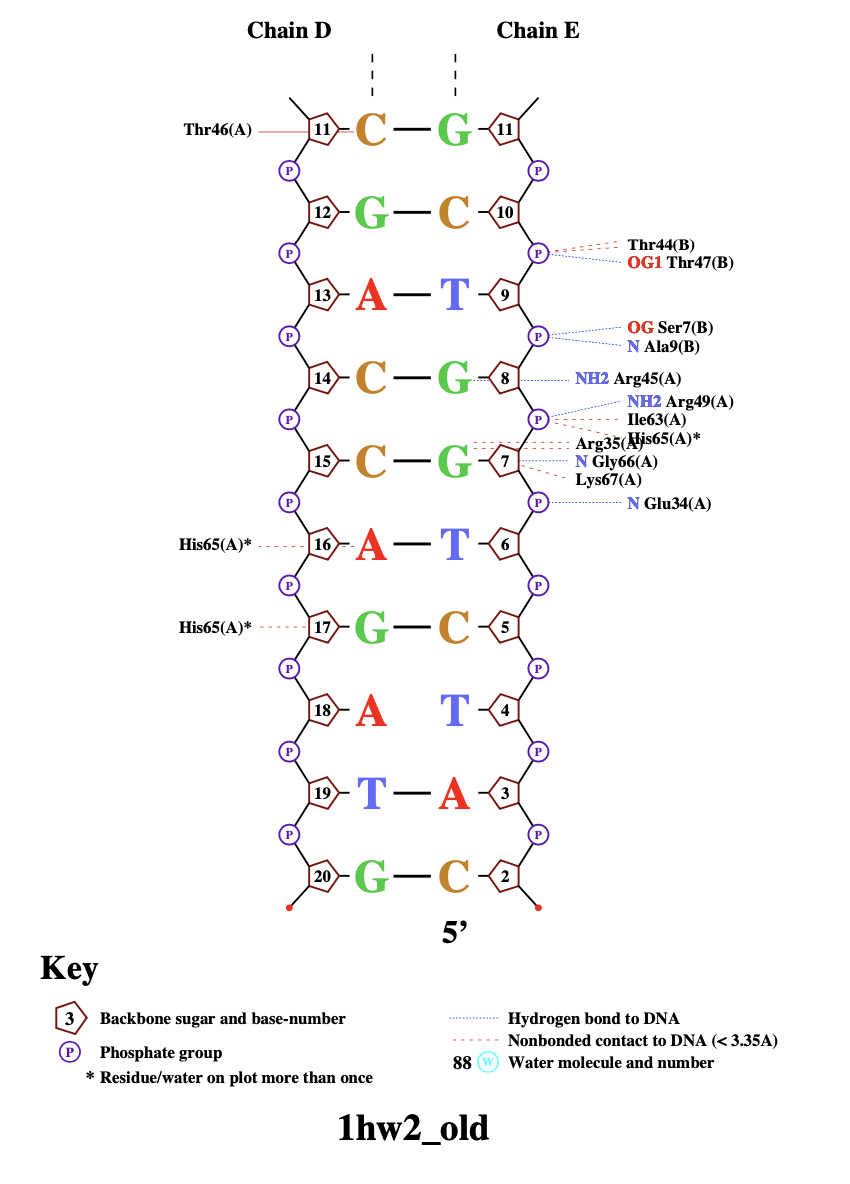
|
В скрипте 1 выполнены следующие упражнения по заданию множеств атомов:
Определите множество атомов кислорода 2'-дезоксирибозы (set1).
Определите множество атомов кислорода в остатке фосфорной кислоты (set2).
Определите множество атомов азота в азотистых основаниях (set3).
Скрипт 2 дает последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1, затем set2 и set3.
Далее с помощью скрипта 3 была построена таблица 2, показывающая количество контактов разного типа в исследуемом комплексе.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 3 | 22 | 25 |
| остатками фосфорной кислоты | 6 | 5 | 11 |
| остатками азотистых оснований со стороны большой бороздки | 1 | 1 | 2 |
| остатками азотистых оснований со стороны малой бороздки | 5 | 10 | 15 |
Можно проследить, что в целом преобладают неполярные контакты.
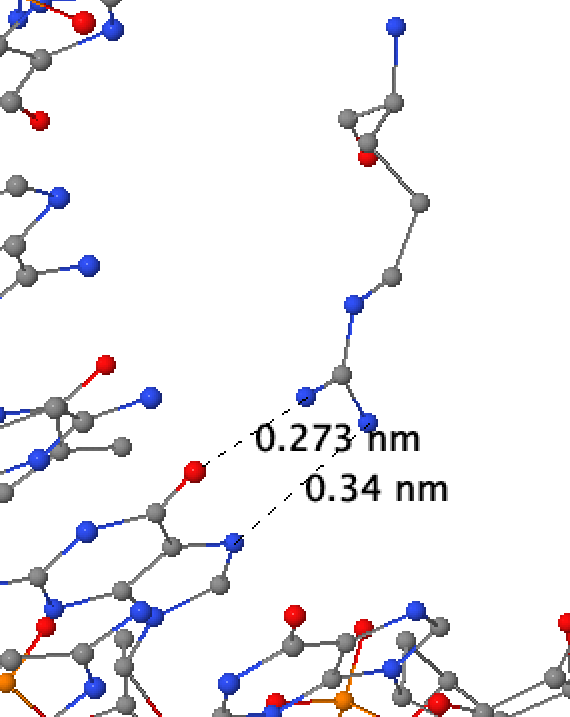
С помощью выдачи nucplot были определены 2 особенных амикокислотных остатка:
С помощью Jmol была выполнена визуализация расположения и контактов данных аминокислот (см рисунки 4, 5).
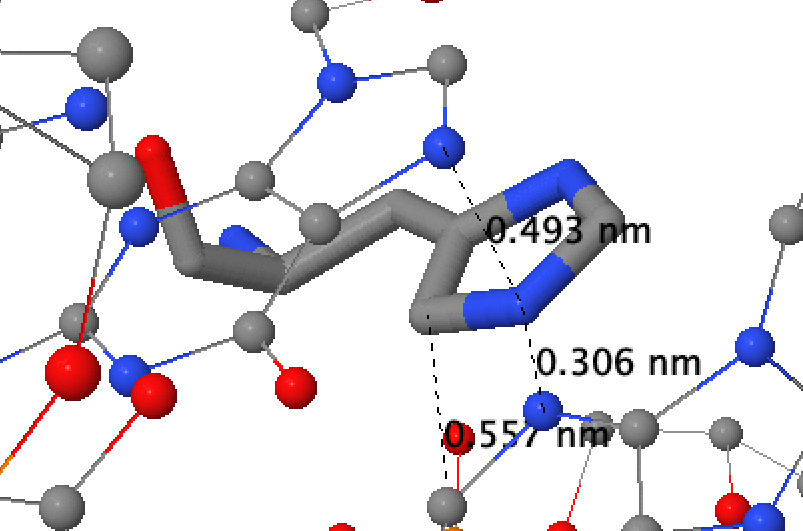
|
|