
Файл со всеми протеомами: см
Файл с выдачей BLAST: см
Находки из выдачи BLAST

С помощью JalView последовательности были скачаны и выравнены алгоритмом Muscle. Далее выравнивание было импортировано в MEGA и алгоритом Neighbor joining было построено дерево.
дерево в формате nwk без длин ветвей
дерево в формате nwk с длинами ветвей
Было получено следующее дерево:
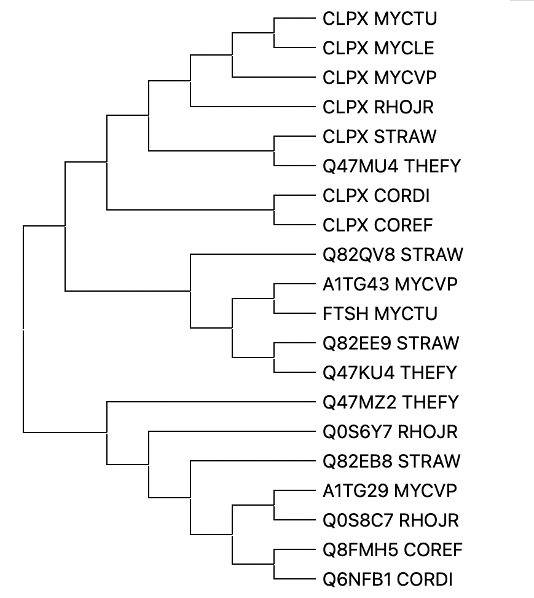

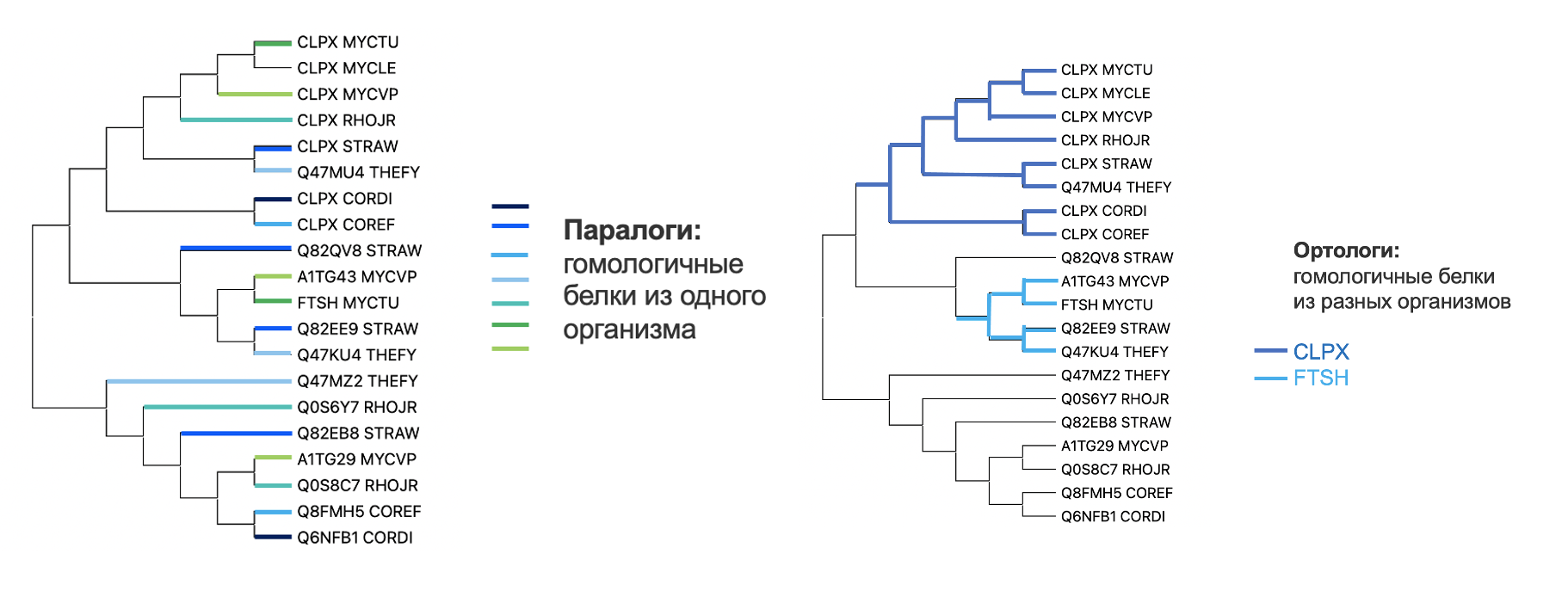
В таблице ниже приведены названия белков, указанных в дереве:
| Q47MU4 | ATP-dependent Clp protease ATP-binding subunit ClpX |
| Q82QV8 | Putative AAA family ATPase |
| A1TG43 | ATP-dependent zinc metalloprotease FtsH |
| Q82EE9 | ATP-dependent zinc metalloprotease FtsH |
| Q47KU4 | ATP-dependent zinc metalloprotease FtsH |
| Q47MZ2 | ATPase |
| Q0S6Y7 | Chaperone protein ClpB |
| Q82EB8 | Putative ATP-dependent Clp protease |
| A1TG29 | ATPase AAA-2 domain protein |
| Q0S8C7 | ATP-binding subunit of ATP-dependent Clp protease |
| Q8FMH5 | Putative endopeptidase Clp ATP-binding chain C |
| Q6NFB1 | ATP-dependent Clp protease ATP-binding subunit |
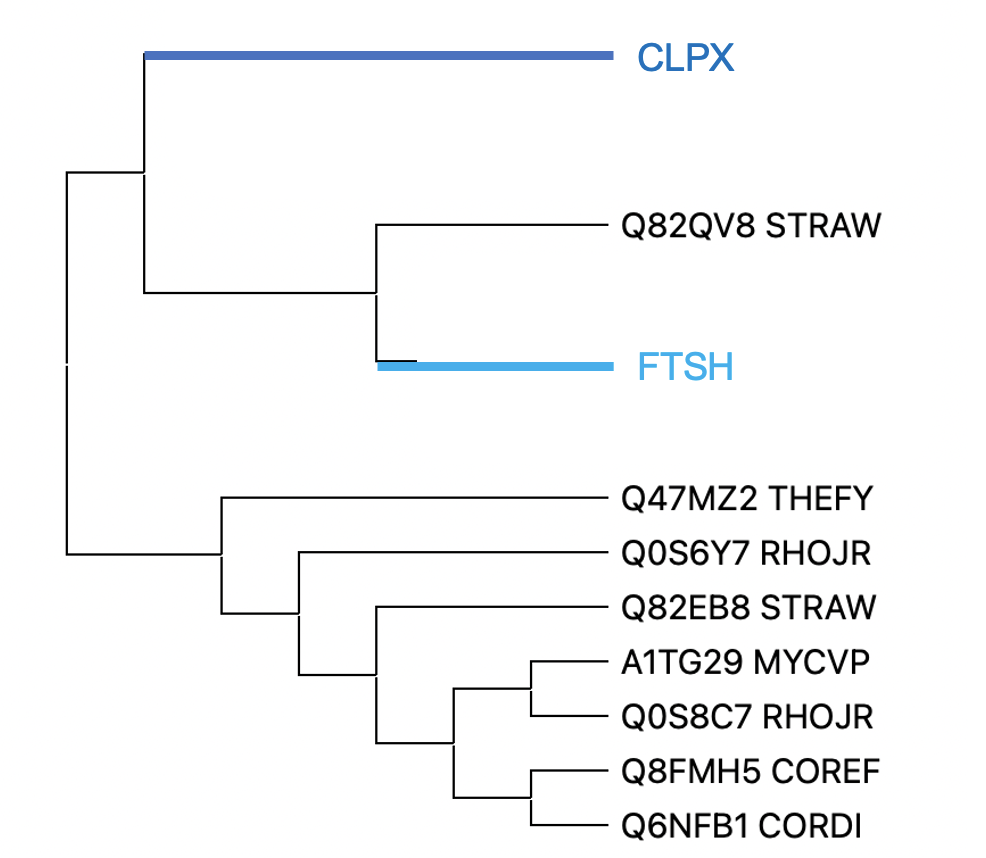
В таблице ниже приведена расшифровка рисунка 5:
| белок | полное название | у каких бактерий? | у всех бактерий? | соответствует ли реконструированная филогения белка филогении бактерий? |
| ClpX | АТФ-связывающая субъединица Clp протеаз | MYCTU MYCLE MYCVP RHOJR STRAW THEFY CORDI COREF | да | да |
| FtsH | АТФ-зависимая цинковая металлопротеаза | MYCTU MYCVP STRAW THEFY | нет | ветвь, содержащая STRAW THEFY, соответсвует филогении бактерий, также сохраняется ветвь с MYCTU MYCVP |