

| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
| Biosynthetic arginine decarboxylase (*) | SPEA_BACSU | SPEA_ECOLI | 53.5 | 10.2% | 18.7% | 482 | 20 |
| Trigger factor | TIG_BACSU | TIG_ECOLI | 606.5 | 31.6% | 48.6% | 86 | 6 |
| Septum site-determining protein MinD | MIND_BACSU | MIND_ECOLI | 598.5 | 43.1% | 66.3% | 14 | 4 |
(*) У белка SPEA_BACSU ID: Arginine decarboxylase, для белка SPEA_ECOLI ID приведен в таблице
1) Скачивание списка иидентификаторов (ID) всех аннотированных (Reviewed) записей, чей идентификатор кончается на _ECOLI:
infoseq sw:"*_ECOLI" -only -name > ecoli.txt
2) Проверка трех выбранных белков на сходство мнемоники с белками штамма 168 сенной палочки:
infoseq sw:"*_BACSU" -only -name | grep 'SPEA'
infoseq sw:"*_BACSU" -only -name | grep 'MIND'
infoseq sw:"*_ BACSU " -only -name | grep 'TIG'
3) Выравнивание последовательности каждой пары программой needle при параметрах по умолчанию:
needle sw:spea_bacsu sw:spea_ecoli spea.needle -auto
needle sw:tig_bacsu sw:tig_ecoli tig.needle -auto
needle sw:mind_bacsu sw:mind_ecoli mind.needle -auto
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
| Biosynthetic arginine decarboxylase (*) | SPEA_BACSU | SPEA_ECOLI | 66.0 | 17.8% | 33.0% | 124 | 15 | 78,6% | 54,3% |
| Trigger factor | TIG_BACSU | TIG_ECOLI | 610.0 | 36.5% | 55.3% | 26 | 4 | 93,9% | 89,4% |
| Septum site-determining protein MinD | MIND_BACSU | MIND_ECOLI | 601.5 | 43.6% | 67.0% | 12 | 3 | 98,9% | 99,7% |
(*) У белка SPEA_BACSU ID: Arginine decarboxylase, для белка SPEA_ECOLI ID приведен в таблице
water sw:mind_bacsu sw:mind_ecoli mind.water -auto
water sw:tig_bacsu sw:tig_ecoli tig.water -auto
water sw:spea_bacsu sw:spea_ecoli spea.water -auto
infoseq sw:SPEA_ECOLI -only -name -accession -length
infoseq sw:SPEA_BACSU -only -name -accession -length
infoseq sw:TIG_BACSU -only -name -accession -length
infoseq sw:TIG_ECOLI -only -name -accession -length
infoseq sw:MIND_BACSU -only -name -accession -length
infoseq sw:MIND_ECOLI -only -name -accession -length
Coverage
SPEA_ECOLI: 644-288+1=357 357/658*100%=54,3%
SPEA_BACSU: 402-18+1=385 385/490*100%=78,6%
TIG_BACSU: 398/424*100%=93,9%
TIG_ECOLI: 386/432*100%=89,4%
MIND_BACSU: 265/268*100%=98,9%
MIND_ECOLI: 269/270*100%=99,7%
В настоящей работе были выровнены человеческий муцин-1 и гомолог клеточного рецептора 1 вируса гепатита А у мыши.
| Protein Name 1 | Protein Name 1 | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
| Mucin-1 | Hepatitis A virus cellular receptor 1 homolog | MUC1_HUMAN | HAVR1_MOUSE | 52.0 | 5.0% | 6.8% | 1138 | 18 |
| Protein Name 1 | Protein Name 2 | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
| Mucin-1 | Hepatitis A virus cellular receptor 1 homolog | MUC1_HUMAN | HAVR1_MOUSE | 81.0 | 24.5% | 33.8% | 48 | 10 | 15,4% | 62,6% |
Результаты глобального и локального выравниваний подтверждают гипотезу о негомологичности выбранных белков:
Для множественного выравнивания была выбрана биосинтетическая аргининдекарбоксилаза 7 организмов, включая кишечную палочку и сенную палочку (ID всех семи белков и полное название белка представлены в таблице 5).
Далее в программе Jalview по ID были найдены последовательности. Выравнивание проводилось с помощью команд в меню Web service → Alignment. Окрашивание было произведено по проценту идентичности Colour → Percentage Identity.
Полученный результат можно посмотреть здесь .
| Protein Name | How many proteins SPEA_*? | Chosen proteins |
| Biosynthetic arginine decarboxylase | 97 | SPEA_BACSU; SPEA_SALDC; SPEA_MARHV; SPEA_ECOLI; SPEA_SHEWM; SPEA_SHEPW; SPEA_SALA4 |
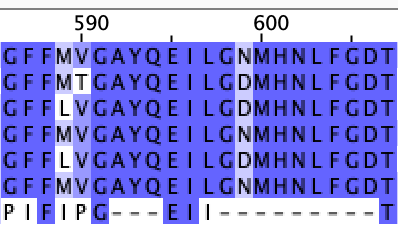
Большая часть белков (6/7) выровнялись достаточно хорошо, так как имееют много совпадений на всей протяженности выравнивания. Лишь белок сенной палочки SPEA_BACSU имеет крайне мало совпадений с остальными и скорее всего не приходится им гомологом. На рисунках 1,2,3 представлены наиболее консервативные участки: столбцы 250-258; столбцы 284-291; столбцы 591-598 и 600-667 соответсвенно. Нетрудно заметить, что самая нижняя последовательность (SPEA_BACSU) даже в этих участках сильно отличается от верних шести. Кроме того, имеется большо количество участков, на которых только у SPEA_BACSU произошли инсерции (например, столбцы 212-216, 341-344).

|

|

|
В качестве гомологичного белка к пектинэстеразе А бактерии Dickeya dadantii была выбрана пектинэстераза А гриба Aspergillus subgen. Nidulantes.
Результаты выравнивания (см таблицы ниже) подтверждают гипотезу о гомологичности выбранных белков:
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
| Pectinesterase A | PMEA_DICD3 | PMEA_EMENI | 304.0 | 27.1% | 40.7% | 116 | 14 | 98,6% | 98,2% |
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
| Pectinesterase A | PMEA_DICD3 | PMEA_EMENI | 301.0 | 26.6% | 39.9% | 121 | 16 |
Если не добавлять -auto при запуске программ needle и water, они требуют указать два параметра
water sw:tig_bacsu sw:tig_ecoli tig2.water
Gap opening penalty [10.0]: штраф за открытие гэпа (по умолчанию 10.0)
Gap extension penalty [0.5]: штраф за продолжение гэпа (по умолчанию 0.5)
На примере программы water попробуем изменить штрафы. Когда были увеличены оба стандартных параметра, исследуемые в предыдущих задачах значения (длина, идентичность,количество гэпов и тд) уменьшились:
| BEFORE | Gap_penalty: 10.0 | Extend_penalty: 0.5 | Length: 405 | Identity: 148/405 (36.5%) | Similarity: 224/405 (55.3%) | Gaps: 26/405 ( 6.4%) | Score: 610.0 |
| AFTER | Gap_penalty: 100.0 | Extend_penalty: 10.0 | Length: 323 | Identity: 117/323 (36.2%) | Similarity: 185/323 (57.3%) | Gaps: 2/323 ( 0.6%) | Score: 350.0 |