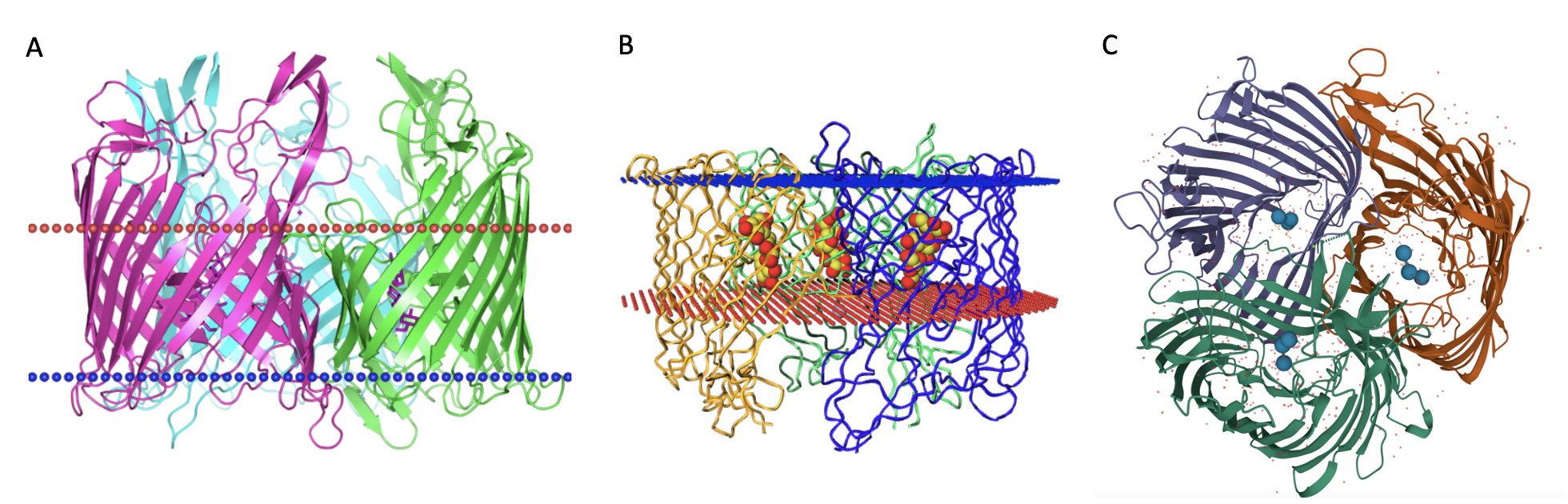
В базе данных OPM был выбран представитель суперсемейства Sugar porins, а именно трансмембранный β-листовой белок Maltoporin бактерии Salmonella enterica. Подробную информацию о нем можно найти ниже в таблице.
| Тип | Трансмембранный белок |
| Класс | Трансмембранный бета-бочонок |
| Суперсемейство | Sugar porins |
| Семейство | Malptoporin-like proteins |
| Организм, обладающий данным белком | Salmonella enterica |
| Локализация | Наружная мембрана грам-отрицательных бактерий |
| Толщина гидрофобной части белка в мембране | 24.5 Å |
| Угол наклона | 0° |
| Топология | субъединица A |
| Число трансмембранных структур | 54 |
| Среднее количество остатков в одном β-тяже белка | 9 |
| Число субъединиц | 3 (A, B и C) |
| Uniprot | LAMB_SALTY |
| PDB | 2MPR |
Координаты трансмембранных сегментов
A - Tilt: 3 - TM segments: 1(3-13),2(39-48),3(59-68),4(75-88),5(99-104),6(125-132),7(138-146),8(170-179),9(186-194),10(212-221),11(227-235),12(279-288),13(294-303),14(315-324),15(330-339),16(353-362),17(371-380),18(417-426)
B - Tilt: 3 - TM segments: 1(3-13),2(39-48),3(59-68),4(75-88),5(98-104),6(125-132),7(138-146),8(170-179),9(186-194),10(212-221),11(227-235),12(279-288),13(294-303),14(315-324),15(330-339),16(353-362),17(371-380),18(417-426)
C - Tilt: 2 - TM segments: 1(3-13),2(39-48),3(59-68),4(75-88),5(98-104),6(125-132),7(138-146),8(170-179),9(186-194),10(213-221),11(227-235),12(279-288),13(294-303),14(315-324),15(330-339),16(353-362),17(371-380),18(417-426)

На сервисе DeepTMHMM для выданного α-спирального белка и для выбранного β-листового белка были предсказаны трансмембранные участки. Результат можно найти ниже в файлах и на рисунке 2.
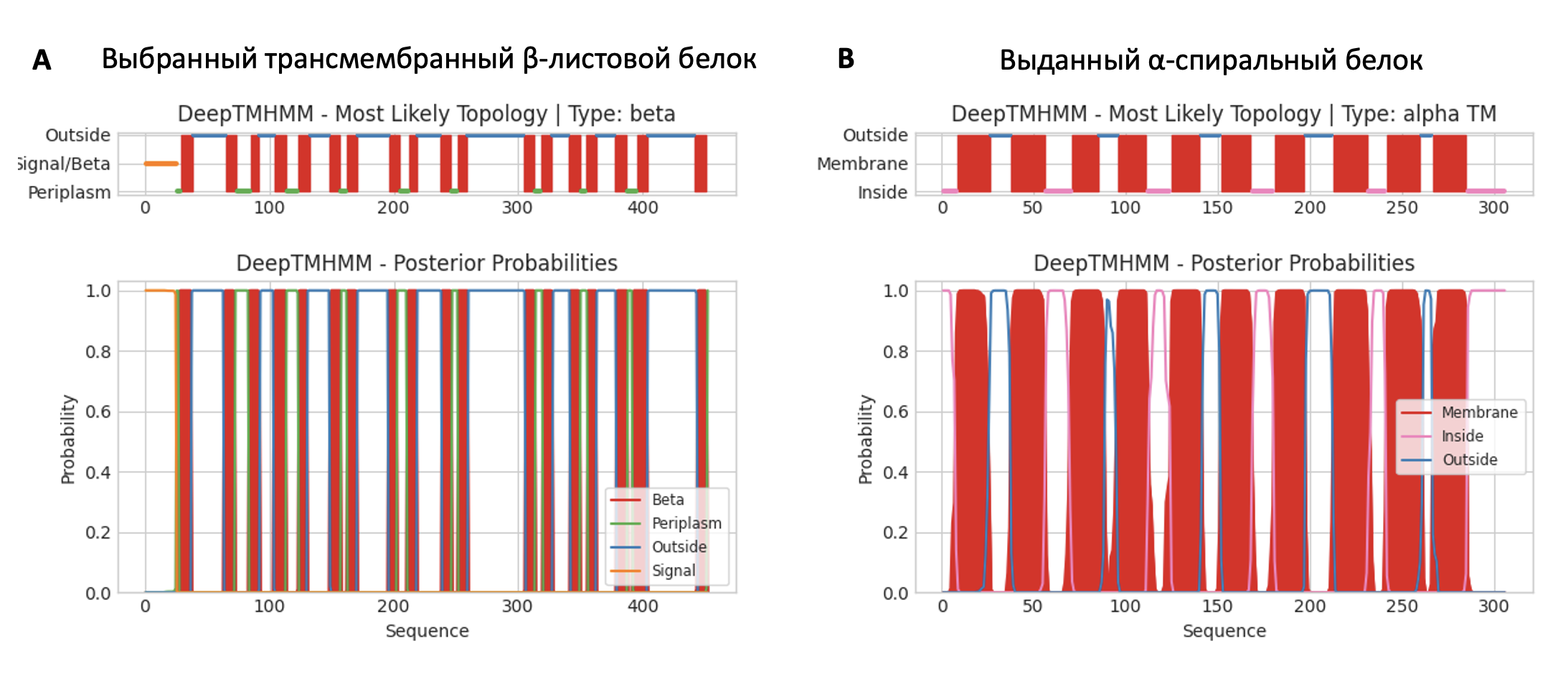
Пояснения к графикам
По оси Х: координаты аминокислотных остатков
По оси У: вероятность того, что данный остаток принадлежит к определённой топологии.
Разными цветами показано расположение участков:
красный - в мембране
розовый - внутри клетки (со стороны цитоплазмы)
голубой - вне клетки
зелёный - в периплазме
оранжевый - сигнал для периплазматической локализации
Обсуждение полученных результатов
Для β-листового белка было предсказано 18 трансмембранных участков,а для выданного α-спирального белка - 10 трансмембранных участков.
Целью данного блока стало предсказание топологии для выданного альфа-спирального белка Uncharacterized transporter YdfC с помощью сервиса PPM 3.0 на сайте OPM.
Параметры
Количество мембран - 1
Тип мембраны - внутренняя мембрана грам-положительной бактерии ( Bacillus subtilis - организм, обладающий исследуемым белком - грам-положительная бактерия)
Топология (N-конец) - внутри (опираемся на данные DeepTMHMM - файлы с результатом).
Allow curvature - нет
Загружаем PDB файл и не учитываем гетероатомы
Результат работы программы представлен в таблице 2 ниже.
| Тип | Трансмембранный белок |
| Класс | альфа-спиральный |
| Название | Неописанный транспортер YdfC |
| Организм, обладающий данным белком | Bacillus subtilis (strain 168) |
| Локализация | Мембрана грам-положительной бактерии |
| Толщина гидрофобной части белка в мембране | 32.1 ± 1.2 Å |
| Угол наклона | 6 ± 2° |
| ΔGtransfer | -85.5 kcal/mol |
| Число трансмембранных структур | 10 |
| Среднее количество остатков в одном а-спирали белка | 20 |
| PDB | Результат |
Координаты трансмембранных сегментов
A 1( 6- 30), 2( 35- 57), 3( 66- 93), 4( 94- 112), 5( 122- 145), 6( 147- 169), 7( 178- 196), 8( 210- 234), 9( 239- 260),10( 267- 284)

Сопоставив данные, полученные в результате работы альтернативных программ, можно отметить следующее:
1. Было найдено равное количество трансмембранных участков
2. Среднее количество остатков в одной a-спирали белка едино
4. Координаты трансмембранных сегментов отличаются не очень сильно
DeepTMHMM
A 1( 8- 25), 2( 38- 56), 3( 70- 88), 4( 96- 111), 5( 125- 141), 6( 152- 168), 7( 181- 197), 8( 213- 233), 9( 242- 261),10( 267- 285)
PPM
A 1( 6- 30), 2( 35- 57), 3( 66- 93), 4( 94- 112), 5( 122- 145), 6( 147- 169), 7( 178- 196), 8( 210- 234), 9( 239- 260),10( 267- 284)
Если обратиться к модели AlphaFold исследуемого белка (рис 4), несложно заметить, что практически весь белок за исключением его концевой части (местами не имеющей вторичной структуры) предсказан с крайне высокой степенью достоверности. Поэтому это не должно было повлиять на работу программы PPM.

Таким образом, предказания полученные программами можно считать достаточно качественным.
Поиск выбранного белка осуществлялся на сайте TCDB по АС P26466. К сожалению, ничего найдено не было. Видимо, запись о данном белке еще не создали.
output: Sorry! p26466 is not included in TCDB
А неописанный транспортер YdfC в базе данных нашелся
РезультатНайденный код: 2.A.7.3.32
2 - электрохимический потенциалзависимый транспортер
А - транспортный белок, образующий пору
7 - Суперсемейство транспортеров лекарств и метаболитов