
В таблице 1 представлено таксономическое положение Dickeya dadantii.
| Kingdom: | Bacteria |
| Phylum: | Proteobacteria |
| Class: | Gamma Proteobacteria |
| Order: | Enterobacterales |
| Family: | Pectobacteriaceae |
| Genus: | Dickeya |
| Species: | D. dadantii |
Dickeya dadantii до 2005 года существовала под названием Erwinia chrysanthemi.
Эта подвижная неспорообразующая грамотрицательная бацилла с загругленными концами и многочисленными жгутиками. Как и все члены семейства Pectobacteriaceae, изучаемая бактерия является факультативным анаэробом. Dickeya dadantii содержит много пектиназ, с помощью которых она может мацерировать клеточные стенки растений, вызывая некроз тканей и мягкую (мокрую) гниль. Вследствие прогрессирующей мацерации тканей растения бактерия получает доступ к многочисленным питательным веществам и активно размножается.
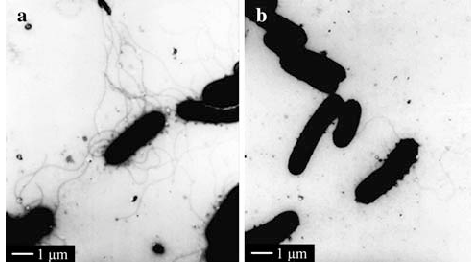
На рисунке 1 под буквой а представлена Dickeya dadantii. Отчетливо видны многочисленные жгутики и очевиден средний размер бациллы: длина бактерии приблизительно равна 3 μm.
На рисунке 2 показана мокрая плесень лука, вызванная данной бактерией. Сочные чешуи размягчились и приобрели желтую окраску.

|

|
| Susceptible Families | Examples of specific species affected |
| Solanaceae | peppers, potato, eggplant, tomato, tobacco |
| Convolvulaceae | sweet potato |
| Brassicaceae | broccoli, radishes |
| Apiaceae | celery, carrot |
| Poaceae | sugar cane, sorghum, rice |
| Bromeliaceae | pineapple, urn plant |
| Asparagaceae | asparagus |
| Amaryllidaceae | onions |
1. Dickeya dadantii является патогеном многих экономически важных сельскохозяйственных культур. Для разработки средств защиты растений нужно понять какие белки работают в клетке, на какие биохимические процессы бактерии можно воздействовать. Секвенирование сильно облегчает поиск этой информации.
2. Бактерия содержит аспариганазу, которая используется в сочетании с другими химиотерапевтическими средствами для лечения острого лимфобластного лейкоза. Следовательно, аспарагиназу Dickeya dadantii можно использовать в случае развития у пациентов аллергии на аспаргиназу Escherichia coli.
3. В связи в увеличением интереса к возоблонвляемым топливным ресурсом и способностью бактерии разрушать пектиновые вещества Dickeya dadantii активно изучается с целью дальнейшего использования данной бактерии в производстве этанольного топлива.
В таблице 3 можно найти общую информацию о белке.
| Раздел UniProtKB | Swiss-Prot |
| UniProt ID | PMEA_DICD3 |
| UniProt AC | P0C1A9; E0SAZ5; P07863; |
| EMBL | L07644; AAA24852.1; CP002038; ADM99554.1; |
| PDB ID | 2NSP, 2NST, 2NT6, 2NT9, 2NTB, 2NTP, 2NTQ |
| Length | 366 AA |
| Molecular weight | 39,373 KDa |
| Recommended Name | Full=Pectinesterase A; Short=PE A; EC=3.1.1.11; |
| Alternative name | Full=Pectin methylesterase A |
Реакция, которая катализируется пектинэстеразой А, выглядит так: [(1->4)-alpha-D-galacturonosyl methyl ester](n) + n H2O = [(1->4)-alpha-D-galacturonosyl](n) + n H(+) + n methanol 3
|
|
+ | n |
|
= |
|
+ | n |
|
+ | n |
|
Пектинэстераза А характерна для многих растений ( Arabidopsis thaliana, Oryza sativa subsp. japonica) и бактерий (Escherichia coli , изучаемая Dickeya dadantii). Lygus hesperus-это единственное описанное многоклелочное животное, которое содержит изучаемый белок. Lygus hesperus - насекомое, которое питается такими растениями, как хлопок, люцерна, клубника.
| What are we looking for? | Request + link | Total amount | Amount of Reviewed (Swiss-Prot) |
| all pectinesterases in all organisms | name:pectinesterase | 25,214 | 118 |
| all pectinesterases A in all organisms | name:pectinesterase name:a | 164 | 9 |
| all proteins in Dickeya dadantii | organism:"dickeya dadantii" | 12,812 | 103 |
| all pectinesterases in Dickeya dadantii | name:pectinesterase organism:"dickeya dadantii" | 5 | 2 |
| all pectinesterases A in Dickeya dadantii | name:pectinesterase name:a org$ | 1 | 1 |
| all pectinesterases in Escherichia coli | name:pectinesterase organism:"$ | 293 | 0 |
| all pectinesterases A in Escherichia coli | name:pectinesterase name:$ | 0 | 0 |
| all pectinesterases B in Escherichia coli | name:pectinesterase name:$ | 0 | 0 |
| all proteins in Escherichia coli | organism:"escherichia coli" | 2,130,102 | 23,166 |
| all pectinesterases A in family Pectobacteriaceae | name:pectinesterase name:a ta$ | 3 | 2 |
| all pectinesterases A in order Enterobacterales | name:pectinesterase name:a taxo$ | 16 | 2 |
| all pectinesterases A in class Gammaproteobacteria | name:pectinesterase name:a t$ | 29 | 2 |
| all pectinesterases A in phuylum Proteobacteria | name:pectinesterase name:a taxono$ | 39 | 2 |
| all pectinesterases A in kingdom Bacteria | name:pectinesterase name:a taxonomy:bac$ | 140 | 2 |
| all pectinesterases in family Pectobacteriaceae | name:pectinesterase taxonomy:pectobact$ | 78 | 4 |
| all pectinesterases in order Enterobacterales | name:pectinesterase taxonomy:enterobacte$ | 880 | 4 |
| all pectinesterases in class Gammaproteobacteria | name:pectinesterase taxonomy:gammapro$ | 1,121 | 4 |
| all pectinesterases in phuylum Proteobacteria | name:pectinesterase taxonomy:prot$ | 1,412 | 6 |
| all pectinesterases in kingdom Bacteria | name:pectinesterase taxonomy:bacteria | 5,419 | 6 |
| all pectinesterases in Eurariota | name:pectinesterase taxonomy:eukaryota | 19,699 | 112 |
| all pectinesterases in Metazoa | name:pectinesterase taxonomy:metazoa | 51 | 1 |
С помощью команд EMBOSS было однозначно опредлелено количество участков с указанными формами.
entret 'sw:PMEA_DICD3' -filter | grep 'FT' | grep -v 'FT ' | tr -s ' ' ',' | cut -d ',' -f 2 | grep 'STRAND' | wc -l
entret 'sw:PMEA_DICD3' -filter | grep 'FT' | grep -v 'FT ' | tr -s ' ' ',' | cut -d ',' -f 2 | grep 'HELIX' | wc -l
entret 'sw:PMEA_DICD3' -filter | grep 'FT' | grep -v 'FT ' | tr -s ' ' ',' | cut -d ',' -f 2 | grep 'TURN' | wc -l
entret 'sw:PMEA_DICD3' -filter | grep 'FT' | grep -v 'FT ' | tr -s ' ' ',' | cut -d ',' -f 2 | grep 'DISULFID' | wc -l
Пектинэстераза А содержит 29 бела-тяжей, 7 альфа-спиралей, 1 поворот, 1 дисульфидный мостик.

В таблице 5 представлены сокращения в строках FT текстового файла Uniprot, особенности последовательности, соответсвующие данным аббревиатурам, и примеры участков исследуемого белка с такими особенностями.
| Abbreviation | Sequence feature | Amino-acid residues of Pectinesterase A, notes, evidences |
| 1)SIGNAL | signal sequence | 1..24 /evidence="ECO:0000250" |
| 2)DISULFID | Вisulfide bridges | 192..212, /evidence="ECO:0000269|PubMed:17717531" |
| 3)MUTAGEN | Mytation | 153 /note="Q->A: Strong decrease in affinity for substrate." /evidence="ECO:0000269|PubMed:17717531" |
| 3)MUTAGEN | Mytation | 177 /note="Q->A: Strong decrease in catalytic activity." /evidence="ECO:0000269|PubMed:17717531" |
| 3)MUTAGEN | Mytation | 178 /note="D->A: Loss of activity." /evidence="ECO:0000269|PubMed:17717531" |
| 4)STRAND | beta-sheet | 29..32 /evidence="ECO:0007744|PDB:2NSP" |
| 5)HELIX | helix | 45..50 /evidence="ECO:0007744|PDB:2NSP" |
| 6)TURN | turn is an element of secondary structure in proteins where the polypeptide chain reverses its overall direction | 86..88 /evidence="ECO:0007744|PDB:2NSP" |
Крайне интересно проанализировать, как сильно точечные мутации влияют на работу энзима. Например, при замене полярного глутамина на алифатический аланин в 153 положении сильно снижается сродство фермента к субстрату. В случае той же замены в 177 положении происходит сильное уменьшение каталитической активности (что предсказуемо, так как аминокислоту в 177 положении называют transition state stabilizer), а когда отрицательно заряженная полярная аспарагиновая кислота в 178 положении меняется на неполярный незаряженный аланин, фермент полностью теряет активность, поскольку аспарагиновая кислота здесь образует активный центр фермента.
Запись в UniProt об изучаемом белке менялась 77 раз.
Сравнение 1 (18-APR-2006) и 77 (07-APR-2021) версии:
Для всех трех кластеров изучаемый белок - репрезентативная последовательность, но он является белком сидом (seed) только в 1 кластере с максимальным процентом идентичности (100%) и лишь 2 записями, следовательно, исследуемый фермент организма Dickeya dadantii наиболее хорошо аннотирован, однако не имеет самую длинную аминокислотную последовательность среди подобных белков у близких организмов.
В таблице 6 видно что размеры кластеров довольно небольшие, и после анализа самого крупного (размер: 255 белков) можно сделать вывод о том, что белок не является очень распространенным, присуетсвует только в нескольких порядках бактерий:
В таблице 6 представлены кластеры белка Pectinesterase A.
| Cluster ID | Cluster name | Size | Lenght | Identity |
| UniRef90_P0C1A9 | Cluster: Pectinesterase A | 31 | 366 | 90% |
| UniRef100_P0C1A9 | Cluster: Pectinesterase A | 2 | 366 | 100% |
| UniRef50_P0C1A9 | Cluster: Pectinesterase A | 255 | 366 | 50% |
На основании поисковых запросов в Uniprot (таблицы 7,8) можно сделать вывод о том, что протеом Dickeya dadantii описан и изучен намного хуже протеома классической Escherichia coli. Всего лишь 2% записей о белках исследуемой бактерии аннотированы вручную, в то время как практически весь протеом Escherichia coli (98%) проверен человеком. Это говорит не только о меньшем интересе ученых к исследуемой бактерии, но и о меньшей достоверности записей. Поэтому сравнивая количество записей о белках, выполняющих определнную функцию, у Escherichia coli и у Dickeya dadantii следует принимать во внимание тот факт, что их число у изучаемой бактерии может быть меньше в силу отсутствия достоверной информации и полного понимания процессов метаболизма этого организма.
В настоящей работе был проведен анализ трансмемранных белков и ферментов у вышеуказанных бактерий (см таблица 6,7). Количество ферментов, как и трансмембранных белков, у Escherichia coli и у Dickeya dadantii примерно одинаково, что неудивительно, поскольку Escherichia coli необходимы разнообразные ферменты для расщепления органических соединений в кишечнике человека и трансмембранные белки для транспорта этих органических соединений, а Dickeya dadantii использует энзимы для разрушения тканей растений, вызывая гниль. По этим же причинам и у обеих бактерий была найдена целлюлаза - фермент, расщепляющий основной компонент клеточной стенки - целлюлозу, причем у Dickeya dadantii записей больше, возможно, потому что Dickeya dadantii паразитирует исключительно на растениях, а в кишечнике человека есть большее разнообразие питательных соединений. Далее были изучены оксидазы. Их количество у Escherichia coli несильно больше, чем у Dickeya dadantii, у обеих бактерий имеются оксидазы типа bo, но у кишечной палочки, в отличие от исследуемой бактерии, имеются еще и оксидазы типа bd. Вероятно, это связано с тем, что в кишечнике выделяется большое количество сероводорода, который подавляет работу цитохромоксидазы bo, и Escherichia coli жизненно необходимы дополнительные оксидазы типа bd, которые более устойчивы к действию H2S.4 А раз у Dickeya dadantii оксидазы типа bd отсутсвуют, значит, в процессе жизнедеятельности этот газ она не выделяет.
Поисковые запросы в UniProtKB
| Organism | Dickeya dadantii (strain 3937) | Escherichia coli (strain K12) |
| Proteome ID | UP000006859 | UP000000625 |
| Proteins | 4532 | 4437 |
| Swiss-Prot, % | 77, 1,7% | 4389, 98,9% |
| Transmembrane proteins, % | 893, 19,7% | 956, 21,5% |
| Enzymes, % | 1602, 35,3% | 1687, 38% |
| Cellulase, % | 2, 0,04% | 1 0,02% |
| Oxidase, % | 22, 0,49% | 32 0,72% |
| Oxidase bd | 0 | 6 |
| Oxidase bo | 3 | 4 |
| ec:* AND organism:"Escherichia coli (strain K12) [83333]" AND proteome:up000000625 |
| ec:* AND organism:"Dickeya dadantii (strain 3937) (Erwinia chrysanthemi (strain 3937)) [198628]" AND proteome:up000006859 |
| annotation:(type:transmem) AND organism:"Dickeya dadantii (strain 3937) (Erwinia chrysanthemi (strain 3937)) [198628]" AND proteome:up000006859 |
| annotation:(type:transmem) AND organism:"Escherichia coli (strain K12) [83333]" AND proteome:up000000625 |
| name:cellulase AND organism:"Escherichia coli (strain K12) [83333]" AND proteome:up000000625 |
| name:cellulase AND organism:"Dickeya dadantii (strain 3937) (Erwinia chrysanthemi (strain 3937)) [198628]" AND proteome:up000006859 |
| name:oxidase AND organism:"Escherichia coli (strain K12) [83333]" AND proteome:up000000625 |
| name:oxidase AND organism:"Dickeya dadantii (strain 3937) (Erwinia chrysanthemi (strain 3937)) [198628]" AND proteome:up000006859 |
| name:oxidase name:bd AND organism:"Escherichia coli (strain K12) [83333]" AND proteome:up000000625 |
| name:oxidase name:bd AND organism:"Dickeya dadantii (strain 3937) (Erwinia chrysanthemi (strain 3937)) [198628]" AND proteome:up000006859 |
| name:oxidase name:bo AND organism:"Escherichia coli (strain K12) [83333]" AND proteome:up000000625 |
| name:oxidase name:bo AND organism:"Dickeya dadantii (strain 3937) (Erwinia chrysanthemi (strain 3937)) [198628]" AND proteome:up000006859 |
1.Scientific classification of bacterium link
2.Electron microscopy of Dickeya dadantii link
3.Uniprot link
4.Oxidases of Escherichia coli link