EMBOSS

|

|

|

|

|

|

|

|

|
Программа getorf пакета EMBOSS
С помощью системы SRS была получена запись D89965.entret банка данных EMBL. Эта запись содержит информацию о мРНК (длиной 448 нуклеотидов), кодирующей белок RSS из желудка серой крысы Rattus norvegicus
Далее с помощью программы getorf требовалось получить набор трансляций всех открытых рамок данной последовательности, которые одновременно определены при использовании стандартного генетического кода, имеют длину не менее 30 аминокислотных остатков, начинаются со старт-кодона (то есть с начала последовательности) и заканчиваются стоп-кодоном (до конца последовательности). Для этого использовалась команда:
Программа нашла 9 рамок считывания. Выводной файл программы можно посмотреть здесь. Пятая найденная рамка идентична кодирующей последовательности в записи D89965.entret:
MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM
AVTAYAYYSCHELTPWLRIQSTNPVQKYGA
Данная запись EMBL ссылается на файл Swiss-Prot с идентификатором P0A7B8. Это файл с записью о субъединице HslV АТФ-зависимой протеазы Escherichia coli (strain K12). При помощи команды needle было построено выравнивание последовательности из P0A7B8 с найденными рамками. Выяснилось, что девятая рамка считывания совпадает с геном E.coli:
HSLV_ECOLI 1 MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGG 50
|||||||||||||||||||||||
D89965_9 1 ---------------------------MKGNVKKVRRLYNDKVIAGFAGG 23
HSLV_ECOLI 51 TADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVAD 100
||||||||||||||||||||||||||||||||||||||||||||||||||
D89965_9 24 TADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVAD 73
HSLV_ECOLI 101 ETASLIITGNGDVVQPENDLIAIGSGGPYAQAAARALLENTELSAREIAE 150
|||||||||||||||||||||||||
D89965_9 74 ETASLIITGNGDVVQPENDLIAIGS------------------------- 98
HSLV_ECOLI 151 KALDIAGDICIYTNHFHTIEELSYKA 176
D89965_9 98 -------------------------- 98
Такому результату можно дать логичное объяснение: при отборе клеток желудка и секвенировании мРНК в образец могли попасться бактерии, обитающие в пищеварителном тракте крысы. Полученную последовательность занесли в базу данных EMBL, где, как известно, может храниться непроверенная информация, и эта ошибка была вначале незамечена. Но когда несоответствие было замечено, к старой записи EMBL добавили ссылку на правильную запись SwissProt (которая по определению проверенная экспертом и является достоверной).
Файлы-списки
Для выполнения задания необходимо было получить список всех записей алькогольдегидрогеназ из Swiss-Prot. Это было сделано с помощью команды seqred sw:adh*_* adh.fasta. Результат можно посмотреть здесь.
Следующим шагом было получение файла с универсальными адресами (USA) этих последовательностей с помощью команды infoseq -only -usa adh.fasta -out usa.txt. Результат можно посмотреть здесь.
Далее следовало получение укороченного списка универсальных адресов - только тех, что были выданы мне, то есть CUPNH, APTAU, DRODI, RAT, AMYME, DROSE, DROAN. Для этого нужно было воспользоваться командой grep:
Результатом является вот этот txt-файл.
Последним этапом было получение fasta-файла с последовательностями из списка. Для этого была использована следущая команда:
Итоговый документ с результатом можно посмотреть по этой ссылке.
Случайная модель для оценки достоверности выравнивания
Для оценки достоверности вывода о гомологии последовательностей на основе их выравнивания было проведено сравнение со случайной моделью.
Мною были выбраны две алкогольдегидрогеназы из моего списка: ADH1_RAT (субъединица А алкогольдегидрогеназы серой крысы Rattus norvegicus) и ADH_DROAN (алкогольдегидрогеназа дрозофилы Drosophila ananassae).
Затем было получено 100 перемешиваний последовательности ADH_CUPNH.fasta (алкогольдегидрогеназа Cupriavidus necator) с помощью программы shuffleseq. Результат показан здесь.
С помощью команды water были построены локальные выравнивания ADH1_RAT с ADH_DROAN и 100 перемешиваниями ADH_CUPNH. Затем с помощью команды grep были извлечены веса всех выравниваний. Вес выравнивания с ADH_DROAN равен 58.0. Веса с преремешиваниями показаны на рис.1.
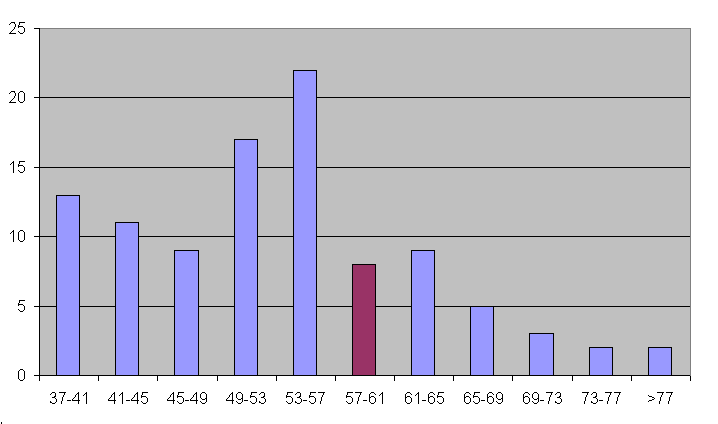
Рис.1. Гистограмма распределения весов выравниваний со случайными последовательностями. Красным выделен столбец, содержащий в себе выравнивание с ADH_DROAN.
Из рисунка 1 видно, что вес выравнивания с ADH_DROAN незначительно отличается от наиболее часто встречающихся весов выравниваний со случайными последовательностями. Таким образом судить о достоверности выравнивания затруднительно. Возможно это связано со слишком дальним родством рассмотренных организмов.
Команды, использованные в данном упражнении можно увидеть здесь
Последнее изменение: 22.10.2014