|
|
Построение множественного выравнивания. Pfam.
Для начала мне надо было составить fasta-файл из 7-8 белков таких, что coverage(%)∈[70;90],
ID(%)∈[50;60], E-value<0.00001. Я попыталась использовать RefSeq, но у всех полученных белков
coverage>90, поэтому я перешла к базе SwissProt. Ни в Blast, ни в PsiBlast у меня не попалось
ни одного белка с ID>39%, что поделать, пришлось выбирать с самыми большими ID из предложенных.
Я выбрала последовательности с AC: O34508.1, A0QRG0.1, Q9CBB2.1, Q8P3K2.1, Q0QWS4.1, P65426.1,
Q9RNZ9.2. (fasta-файл.)
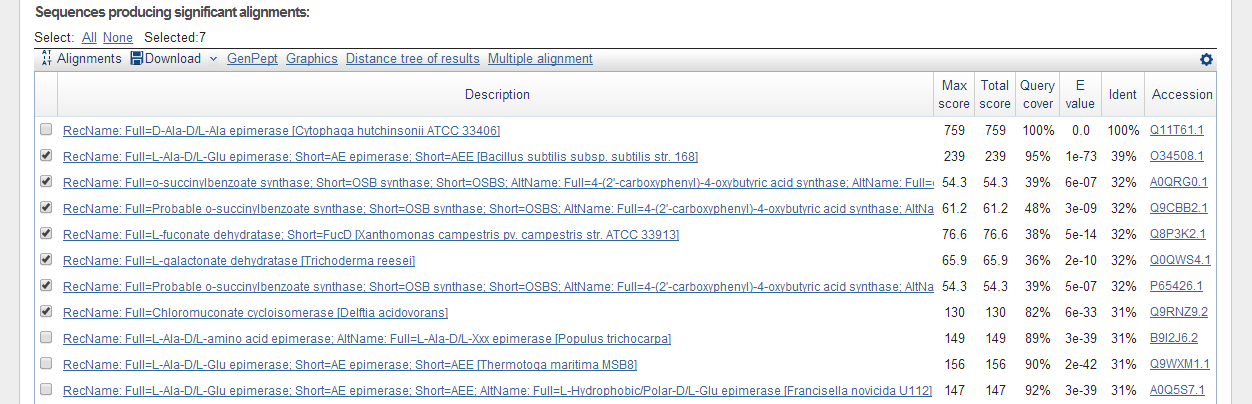
Рис. 1. Выбранные белки.
C помощью программы muscle на сервере kodomo я построила множественное выравнивание выбранных
белков. Для этого я ввела команду: muscle -in seq.fasta -out alseq.fasta.
(Полученный файл.) Само выравнивание
представленно на рис. 2.
Рис. 2. Множественное выравнивание (muscle).
Затем я построила множественное выравнивание выбранных
белков с помощью программы mafft на сервере kodomo.
Для этого я ввела команду: muscle seq.fasta > alseq2.fasta.
(Полученный файл.) Само выравнивание
представленно на рис. 3.
Рис. 3. Множественное выравнивание (mafft).
Далее для сравнения полученных выравниваний я решила использовать снова программу muscle.
Для этого я ввела запрос: muscle -profile -in1 alseq.afa -in2 alseq2.afa -out bothalseq.afa,
полученный результат (Полученный
файл.) на рис. 4. Я несколько опасалась, что, отступив от начальных указаний, я получу плохо
совпадающие выравнивания, однако этого не произошло. Вы можете видеть хорошее совпадение раскрашенных
участков во всех позициях (последовательности в выравниваниях перемешаны по-разному). Это может говорить
о том, что выбранные последовательности действительно гомологичны и их выравнивания не сильно зависят
от матрицы.
Рис. 4. Множественное выравнивание (muscle).
Далее с помощью сайта Pfam нужно было найти домены Pfam-семейств исходной последовательности. Было найдено
два Pfam-A совпадения, их можно видеть на рис. 5.
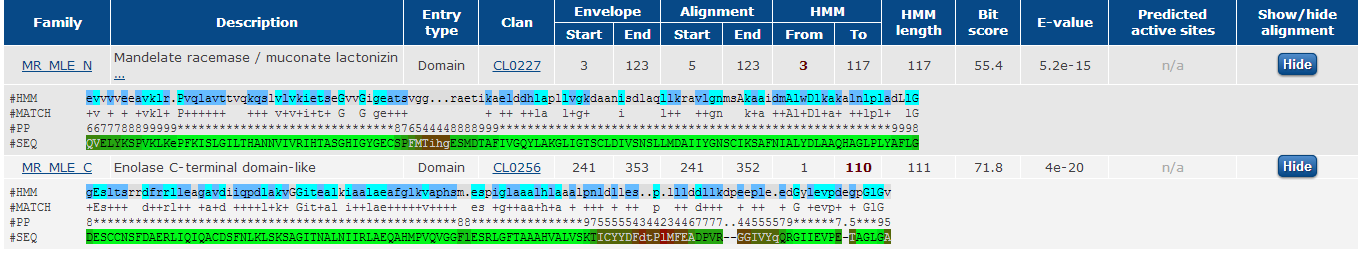
Рис. 5. Результат поиска.
Ссылка на проект.
|




