|
|
EMBOSS
Первым делом нужно было получить файла с записью
D89965 банка EMBL. Для этого я воспользовалась сервером kodomo, введя следующий запрос (c автоматическим параметром выхода):
entret embl:d89965
В этой записи содержится полная последовательность и аннотации гена, кодирующего мРНК Серой крысы (Rattus norvegicus).
Соответствующий белок участвует в трансдукции RT-сигнала желудке крысы.
Далее нужно было разобраться в программе getorf и вывести в
файл все
возможные открытые рамки считывания, удовлетворяющие трем
условиям: не менее 30 нуклеотидов, от старт-кодона до стоп-кодона и должны быть определены при использовании стандартного
генетического кода. Строка запроса выглядела так (с автоматическим параметром выхода;
90, а не 93, так как команда сама не учитывает стоп-кодон):
getorf embl:d89965 -find 1 -minsize 90
В результате 5 рамок, из которых 2 на комплементарной цепи. Среди них есть и та, которая указана в CDS, только в случае CDS
у нее границы 163..435 (до начала стоп-кодона), а в файле с рамками - 163..432 (до начала последней аминокислоты).
Затем я с помощью все той же программы entret получила
файл записью из банка Swiss-Prot,
на которую была ссылка в записи из EMBL. Для этого я ввела следующий запрос:
entret sw:p0a7b8
Полученная последовательность содержит в себе последовательность из 5-й рамки считывания (из полученных ранее), что видно
здесь. Строго говоря, я не знаю, как эта рамка
считывания попала в файл, так как заканчивается она там, где заканчивается цепь ([294 - 1]), а не на стоп-кодоне. Зато это хорошо
объясняет продолжение новополученной последовательности в ту сторону за нашу рамку считывания. Что касается участка перед началом -
тоже ничего удивительного. Настоящий старт - кодон тоже не попал в последовательность, с которой мы работали изначально, но попал
другой метионин из середины, который и был принят за старт-кодон.
Далее надо было скачать в файл все доступные в Swiss-Prot
последовательности алкогольдегидрогеназ. Для этого я использовала команду seqret (fasta-формат устанавливается автоматически).
Строка запроса:
seqret sw:adh*_* adh.fasta
C помощью запроса:
infoseq sw:adh*_* -only -usa > sw-usa
я получила еще один файл, который содержит только список
универсальных адресов USA в Swiss-Prot.
Затем мне надо было создать список только из тех кодов, которые
соответствуют выданным мне организмам. Для этого я воспользовалась следующей командой:
grep -E 'ENTHI|DROFL|METVS|SCHPO|ZEALU|CERRO|PERMA' 'sw-usa' > list
А потом на основе этого списка получить файл в fasta-формате
с последовательносями этих алкогольдегидрогеназ. Строка запроса:
seqret @list list.fasta
Для дальнейших экспериментов из предложенных организмов я выбрала Zea luxurians (вид кукурузы) и Drosophila flavomontana
(дрозофила), так как это достаточно далекие виды. Перемешивала я последовательность кукурузы, создав специально отдельный
файл с ее последовательностью под названием "ZEALU",
вот результат . Строка запроса:
shuffleseq -shuffle 100 ZEALU adh1_zealu.fasta
Затем собственно выравнивание. Я сделала сначала отдельно 100 случайных, а потом одно неслучайное, чтобы было виднее, что происходит.
"DROFL" - файл с последовательностью дрозофилы. Мои запросы (штрафы за
гепы стандартные):
water DROFL adh1_zealu.fasta test
water DROFL ZEALU test2
Чтобы собрать все счета в новом файле "score", я выполнила
следующие запросы:
grep Score test|sed 's/# Score //' > score
echo '60.0' >> score
Последним ходом я прибавила к списку счет "настоящего" выравнивания.
Для наглядности результаты представлены на рис. 1. По оси Y счета выравниваний. Для удобства все счета отсортированы
по возрастанию. Красным выделено "настоящее" выравнивание. Можно видеть, что против ожиданий "настоящее" выравнивание имеет счет
меньше некоторых случайных. Это связано с тем, что я взяла очень далекие организмы, поэтому их алкогольдегидрогеназы выравниваются
довольно плохо. Тем не менее "настоящее" все-таки одно из лучших, так как какая-никакая гомология все же присутствует.
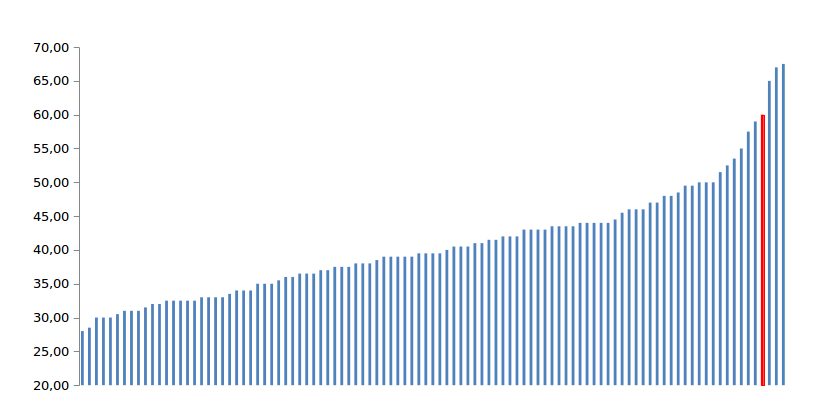
Рис 1. Сравнение выравниваний.
|
