|
|
Онлайн BLAST
Для начала мне надо было найти с помощью megablast организм (бактерию), частью генома которого является выданная мне
последовательность из 300 нуклеотидов. Для поиска я пользовалась параметрами по умолчанию, установив только в качестве базы для поиска
refseq_genomic, в поле Organism: bacteria (taxid:2), и убрала галочку с пункта "Low complexity regions". Megablast
удобен как раз для такого рода поисков, откуда взят участок генома.
Процесс поиска происходил порядка 10 минут. В результате была найдена бактерия Candidatus Blochmannia floridanus. Эта
бактерия является эндосимбионтом некоторых клеток муравьев. Хромосома одна, кольцевая.
Затем мне надо было выбрать такой белок человека, для которого идентификатор в Swiss-Prot и моя фамилия начинаются с максимального
количества одинаковых букв. Для этого я ввела следующий запрос:
infoseq sw:kh*_human -only -name -desc -out proteins
Таких белков оказалось 8 (список) и ни один из них не начинается с
"kha", так что я выбрала прямо первый "KHK_HUMAN". Чтобы получить
файл с самой последовательностью я выполнила следующую команду:
seqret sw:khk_human -auto
Потом с помощью сайта ENA найти гомологов выбранного белка в геноме Африканского слона (Loxodonta africana). Для поиска
нужна нуклеотилная последовательность. Чтобы ее добыть я сначала ввела такой запрос:
entret sw:khk_human -auto
Затем из полученного файла узнала идентификатор
последовательности мРНК в EMBL и загрузила еще один файл,
в котором есть уже нужная нуклеотидная последовательность, такой командой:
entret embl:x78678 -auto
Поиск на сайте ENA выдал всего три результата (рис.1). Как можно заметить, наилучшая находка имеет e-value 10-116,
длину 344 и identity 88%, что говорит о достаточно близком родстве. Найденный ген в геноме слона имеет координаты [40628806..40634573],
всего 2 интрона.
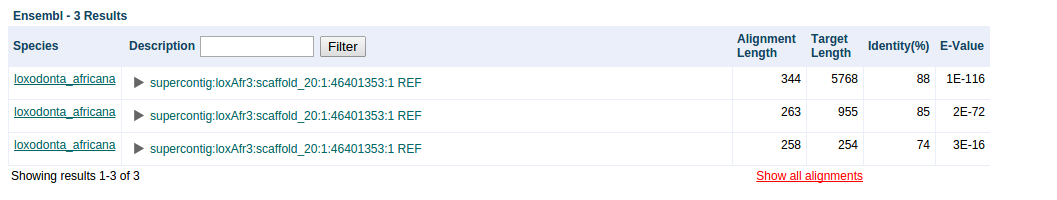
Рис 1. Результат поиска.
Для выполнения последующих заданий мне нужно было выбрать последовательность, кодирующую тРНК из моей бактерии
(Cytophaga hutchinsonii). Воспользовавшись указанием, я скачала
файл, относящийся к моей бактерии, с расширением .frn.
Из него я выбрала последовательность тРНК (переносящую аланин) и поместила в отдельный
файл. Затем я выяснила название порядка, к которому относится
моя бактерия, - Cytophagales.
Далее поиск гомологов того же порядка тремя разными способами:
Способ I
Использовать алгоритм megablast. Параметры я использовала, как в первом задании, но поиск не по всем бактериям, а только по
соответствующему порядку. В результате нашлась только оригинальная последовательность (разумеется, e-value < 10-3). Это
можно объяснить тем, что настолько близких гомологов, чтобы их нашел megablast, у этой тРНК нет.
Способ II
Использовать алгоритм blastn с параметрами по умолчанию. Нашлось 95 совпадений, из которых 69 с e-value < 10-3.
Этот способ в отличие от первого все-таки является настоящим способом поиска гомологов.
Способ III
Использовать алгоритм blastn с длиной слова = 7, match/mismatch = 1/-1 (максимально чувствительные параметры,
доступные на сайте). Находок 87, среди которых, например нет самых верхних (по e-value) в способе II. С e-value < 10-3
- 64. Различия не очень существенные и чем вызваны, я не знаю. Я ожидала отличия в другую сторону, раз уж более
чувствительная система.
|
