| Главная |
| Семестр 1 |
| Семестр 2 |
| Семестр 3 |
| Семестр 4 |
| О себе |
| Ссылки |
Предсказание генов у прокариот
Для аннотации был дан контиг из желудка Wallabia bicolor. Проведем предсказание двумя различными способами и сравним результаты.
Способ I
Будем предсказывать гены по открытым рамкам считывания. Применим программу getorf для поиска искомых рамок. Установим порог в 60 триплетов, такие рамки вероятнее будут соответствовать каким-то генам. Строка запроса:getorf kontig -table 11 -find 1 -minsize 180 -out kontig.orfНашлось 18 рамок считывания (файл). Теперь узнаем, какие из предполагаемых генов имеют в базе Swiss-prot преполагаемых гомологов. Для поиска я использовала blastp, установила в качестве базы "swissprot", а в поле "Organism" "Bacteria (taxid:2)", так как в желудке с большой вероятностью могли попасться только бактерии. Результаты поиска я представила в Таблице 1, оставив только те результаты, в которых e-value<0.001, а перекрывание (если не скраю контига) не менее 80%. Первая строка под вопросом: перекрывание маленькое, но близко к краю, зато e-value очень хорошее.
| 1776 | 2657 | ? | - | описание отсутствует |
| 768 | 1769 | 334 | - | амидотрансфераза |
| 222 | 764 | 181 | - | регулятор транскрипции |
| 1 | 222 | 74 | - | аминотрансфераза |
Способ II
Теперь воспользуемся программой GeneMark. Нашлись те же рамки, однако с чуть сдвинутыми границами. Так же граница одной из по предположению программы лежит за началом контига, а у меня на первом нуклеотиде. Права, вероятно, программа, но я так написала, потому что все условия были выполнены и для указанного мной участка. Почему сдвинуты границы, я не понимаю. Подробные результаты в таблице 2.
| <1 | 222 | ? | - |
| 219 | 701 | 483 | - |
| 765 | 1745 | 981 | - |
| 1773 | 2582 | 810 | - |
Pdf-файл с результатами работы программы. В этом файле приведен график кодирующего потенциала (вертикальная ось). По горизонтальной оси номера нуклеотидов. Жирной черной линией выделены те нуклеотиды, которые по мнению GeneMark составляют совой ген, что так же видно и по их высокому кодирующему потенциалу. Я предполагаю, что провалы в графиках близко к краям гена могут объяснить различие между рамками из первого способа и второго. Ниже я приведу фрагменты графика.
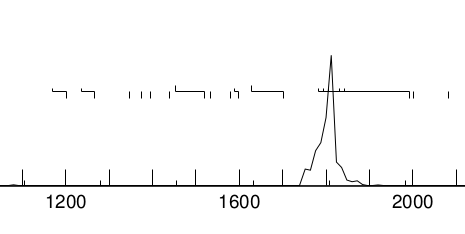 Рис 1. Участок графика с предположительно некодирующей последовательностью нуклеотидов. |
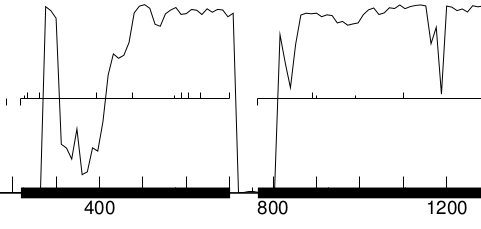 Рис 2. Участок графика с предположительно кодирующими последовательностями нуклеотидов. |
В моем случае при изменении эвристических параметров фактически ничего не изменилось. Только тот ген, у которого координата начала была "<1" стала просто "1". Выходные файлы: обычный, .pdf.