|
|
Восстановление предкового состояния доменной архитектуры
Часть 1. Выбор объекта изучения и построение выравнивания.
У моего белка ничего подходящего не нашлось, поэтому я почти случайно наткнулась на в итоге выбранный домен (PF00049).
Это семейство инсулина,
в которое входит подсемейство инсулина и инсулиноподобых факторов роста и подсемейство релаксина. Все они обладают широкой гормональной
активностью в организме. Ссылка результата на сайте Pfam.
Этот домен содержит 5 архитектур, но одна представленна всего одной последовательностью. На рис 1 показаны все, но для дальнейшего
исследования я выбрала первую и вторую архитектуры.

Рис 1. Схемы строения архитектур, содержащих домен инсулина.
Я скачала выравнивание всех последовательностей, открыла в Jalview, раскрасила по консервативности и прикрепила одну
3D структуру, проект тут.
Затем с помощью скрипта с сайта kodomo "swisspfam-to-xls.py" и команды:
python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -m AC -o PF00049
я получила файл с инфомацией о
доменной структуре каждой из последовательностей. Файл "AC" содержит ID домена.
Из базы данных Uniprot с помощью файла
со списком идентификаторов был скачен файл с записями
о последовательностях. С использованием еще одиного скрипта с сайта kodomo "uniprot-to-taxonomy.py" и команды:
python uniprot-to-taxonomy.py -i PF00049_uniprot -o PF00049_taxonomy
был получен файл с данными о таксономии.
На основе этих данных была получена таблица. На листе
"whole table" отмечены выбранные последовательности. В качестве таксона я выбрала царство Metazoa, а в качестве подтаксонов
типы Chordata и Arthropoda. Когда я выбирала последовательности, я обнаружила, что во второй архитектуре меньше
представителей типа Arthropoda, чем нужно, но переделывать все я уже не стала (а замена архетиктуры на другую не помогает).
Получилось 26 последовательностей первой архитектуры и 29 - второй. Но дальше началось самое интересное.
Вторая архитектура по описанию и данным из базы содержит два домена инсулина, я рассчитывала сравнить их друг с другом и инсулином из
первой архитектуры, но как оказалось, это не два домена, а две половинки одного с небольшим наложением. После соединения
последовательностей выравнивание стало приличным. В нем меньше последовательностей второй архитектуры, чем планировалось изначально,
так как часть почему-то не нашлась в базе. Тут финальное
выравнивание и проект.
Ко всем именам последовательностей прибавлено в начале 1_A_/1_С_/2(1)_A_/2(1)_C_, что обозначает, к каким
архитектурам (1 - первая, 2(1) - вторая) и типам (A - Arthropoda, C - Chordata) относится домен.
На основании этого было построено дерево методом наибольшего правдоподобия, посчитан бутстрэп ветвей, чувствительность к гэпам была
снижена до нуля, укоренение было проведено с помощью программы figtree. Результат на рис 2.
Скобочная формула дерева по ссылке.
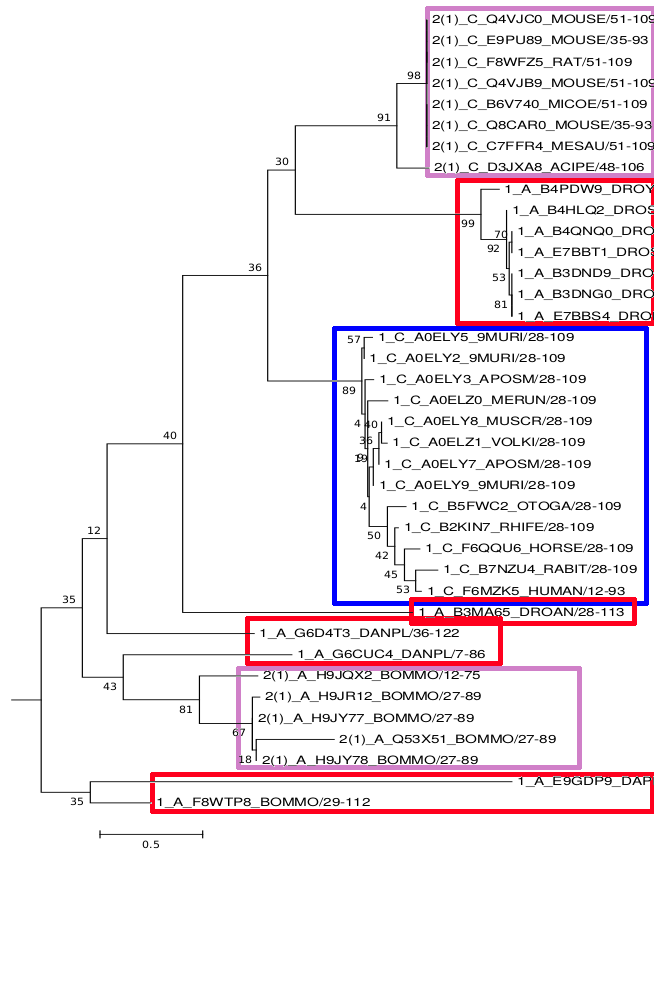
Рис 2. Филогенетическое дерево домена инсулина (PF00049). Построено методом Maximum Likelihood.
Дерево получилось с низкими бутстрэпами ветвей, но в целом правдоподобное. Только последовательности 1_A_* оказались разбросаны
по всему дереву. Однако все же не перемешались с другими кладами, а только в нетривиальных местах ответвились (красные рамки).
Последовательности второй архитектуры четко отделились друг от друга и оказались в разных частях дерева (фиолетовые рамки), а
Хордовые с первой архитектурой выделились четко в свою кладу (синяя рамка), их (1_С_*) я и выбрала в качестве подсемейства
для дальнейших заданий.
Выравнивание выбранных последовательностей тутU.
С сайта Uniprot был скачен файл со всеми
последовательностями домена инсулина (PF00049). Затем с помощью следующих команд на сервере kodomo:
hmm2build hmm2build_result subfamily.fasta
hmm2calibrate hmm2build_result
hmm2search hmm2build_result uniprot-PF00049.fasta > hmm2seach_result
был получен файл с результатом поиска по профилю
последовательности. Всего было найдено 1375 последовательностей с e-value от 6.6E-66 до 9.9.
На основе этих данных была составленна таблица
и постороена ROC-кривая (рис 3, первый лист таблицы).
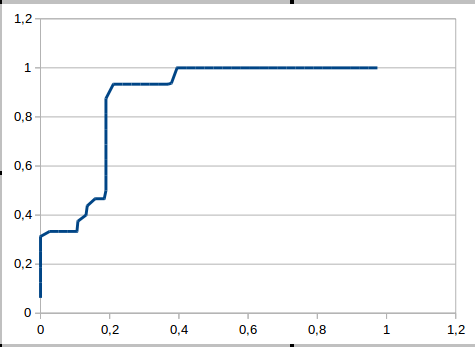
Рис 3. ROC-кривая.
При выбранном пороге E-value 6.9E-066 чувствительность 0.875, а специфичность 0.811. Результат в Таблице 1.
Таблица 1. Результаты поиска по профилю при выбранном пороге E-value.
| На самом деле |
Принадлежит подсемейству |
Не принадлежит подсемейству |
Сумма |
| Выше порога по профилю |
14 |
7 |
21 |
| Ниже порога по профилю |
1 |
30 |
31 |
| Сумма |
15 |
37 |
52 |
|


