|
|
Реконструкция филогении по нуклеотидным последовательностям. Паралоги.
Для этого задания мне нужны были последовательности 16s рРНК. Я их скачала из базы данных Silva. Затем построила
выравнивание
программой Muscle и дерево (рис 1).
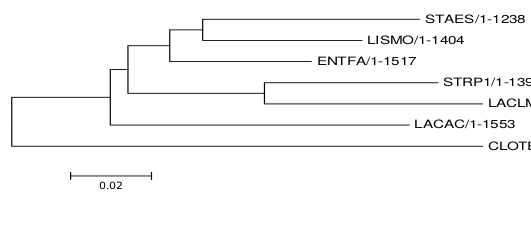
Рис 1. Дерево, построенное на основе нуклеотидных последовательностей 16s рРНК.
Если сравнивать дерево с правильным из первого задания, то оно получилось совсем недостоверным. Только две правильных нетривиальных
ветви.
Следующая часть посвящена анализу гомологов белка CLPX_BACSU в протеоме все тех же последовательностей. Для этого был проведен
локальный бласт по скаченным с сервера kodomo протеомам бактерий. В результате найдено было 13 совпадений с приличным e-value.
Дерево (рис 2) было построено методом UPGMA. На нем черной рамкой выделены гомологи, одинаковые белки в разных видах. Но разделились
они в этих круппах не так, как в правильном дереве. Кружком отмечено вероятное место разделения путей эволюции белков в результате
видообразования. Подчеркнуто три разных белка одного вида бактерии. Так как два из них разошлись после отделения от третьего,
их можно назвать паралогами.
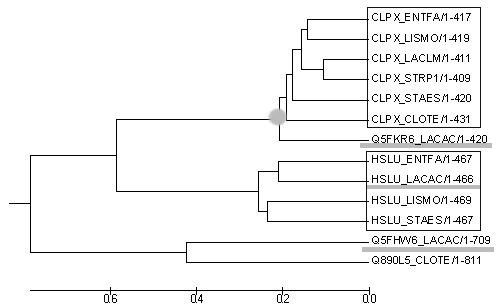
Рис 2. Дерево гомологов белка CLPX_BACSU.
|

