| Главная |
| Семестр 1 |
| Семестр 2 |
| Семестр 3 |
| Семестр 4 |
| О себе |
| Ссылки |
Ферменты и метаболические пути. База данных KEGG
Задание 1.
Для простоты я выбрала из предоставленных ферментов уже известный мне 1.1.1.1 (алкоголь дегидрогеназа).
С помощью сервиса ENZYME можно найти следующую информацию по ферменту, каждой из цифр EC-кода и ферментативной активности.
Название: Alcohol dehydrogenase
Альтернативное название: Aldehyde reductase (говорит о том, что фермент катализирует и обратную реакцию образования спирта из альдегида)
Катализируемая реакция:
An alcohol + NAD(+) <=> an aldehyde or ketone + NADHКофакторы: Zn(2+), Fe cation
A secondary alcohol + NAD(+) <=> a ketone + NADH
С помощью таблицы 1 покажем, что означают цифры EC-кода (полезный сайт):
| Класс | 1 | Оксидоредуктазы | Oxidoreductases |
| Подкласс | 1 | Действие на группу CH-OH донора | Acting on the CH-OH group of donors |
| Подподкласс | 1 | Использует NAD+ или NADP+ как акцептор | With NAD+ or NADP+ as acceptor |
| Порядковый номер | 1 | Алкоголь дегидрогеназа | Alcohol dehydrogenase |
Задание 2.
В базе данных KEGG я нашла карту метаболического пути для метаболизма простых эфиров (пункт 6).
Этот путь по данным KEGG связан с метоболизмом глицерофосфолипидов через 1-ацилглицерон-3-фосфат.
Затем надо было выбрать три организма из доступных в базе данных KEGG, приведу данные о них в таблице 2.
| Бактерии | Delftia acidovorans | карта | 2.5.1.26 3.1.1.4 |
| Археи | - | - | - |
| Эукариоты | Ursus maritimus (Белый медведь) |
карта | 2.5.1.26 3.1.1.4 3.1.3.4 GGT 2.8.2.11 2.7.8.1 2.7.8.2 LPCAT LPCAT2 3.1.1.47 3.1.4.4 LPCAT4 3.1.4.39 3.3.2.5 ENPP6 |
Можно заметить, что даже у млекопитающих не содержится весь путь, изображенный на карте, у бактерий лишь малая часть, а у Архей совсем нет необходимых ферментов.
Задание 3.
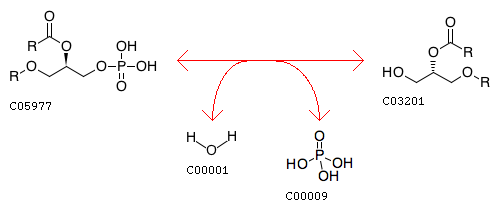
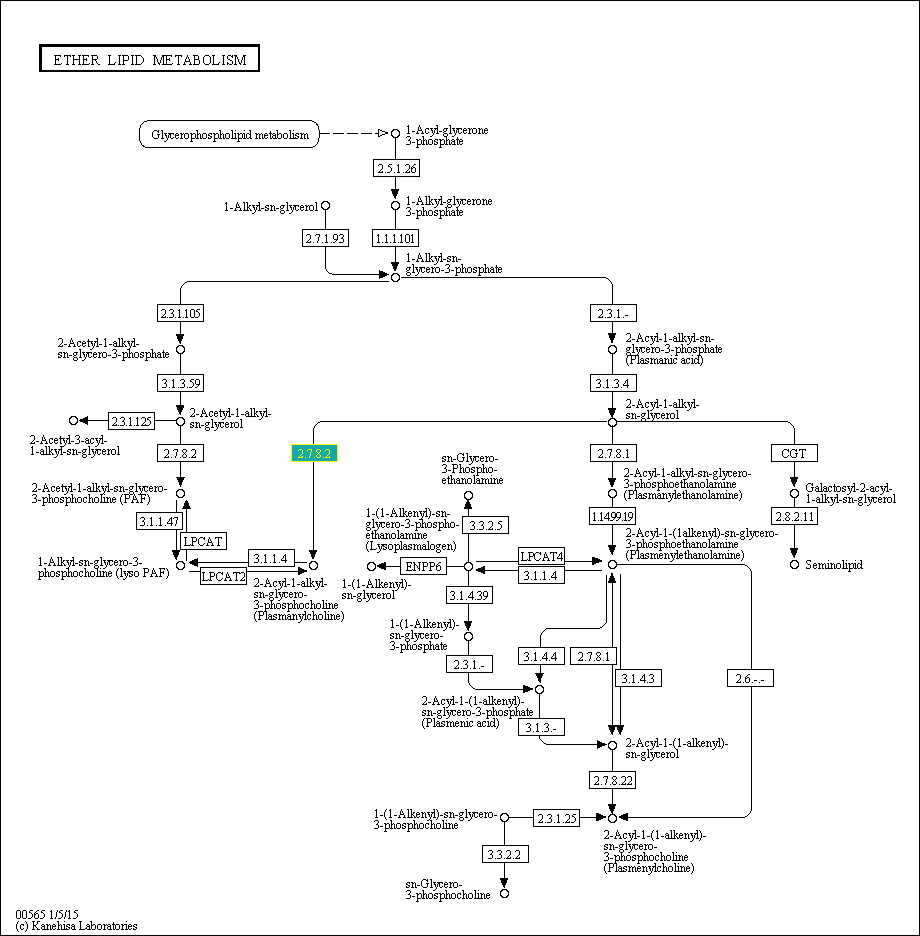
Я выбрала произвольную реакцию. Ее катализирует фермент 3.1.3.4, идентификатор реакции: R04162. Ссылка на страницу с реакцией тут, сама реакция на рис 2 и выделена на рис 1.
Рис 1. Карта метаболического пути с выделенной реакцией.
Рис 2. Катализируемая реакция.
Задание 4.
Для этого задания я решила выбрать другой фермент 2.7.8.2. При наведении нашлось две группы рядов: K00994 (79 представителей) и K13644 (73 представителя). Выбранная реакция показана на рис 3.
Рис 3. Положение выбранного фермента (выделен цветом) на карте метаболического пути.
Последовательностей из базы Uniprot нашлось для K00994 -- 32, а для K13644 -- 29. Затем я построила множественное выравние с помощью Muscle (вот файл) и открыла в Jalview (проект).
На первый взгляд белки выравнены хорошо. Есть несколько белков, у которых просто отсутствуют некоторые части, но в других местах они имеют достаточно консервативные участки.
Последнее -- надо было построить филлогенетическое дерево этих белков.
Можно обратить внимание на то, что дерево не распалось на две группы, соответствующие ортологическим рядам, что говорит о том, что эти белки родственны в той или иной мере друг другу. При этом ветви, отделяющие более мелкие группы, таких три группы, состоят только из представителей одного ряда. Есть одно исключение: белок из ряда K00994 попал в группу из представителей другого ряда, хотя сразу от них отделился. Достоверность попадания его с ними в одну группу 42, а вот уже достоверность отделения на следующем шаге 100. Мне кажется, что это, вероятно, ошибка, так как иначе вторая достоверность полная, а первая -- меньше половины.