| Главная |
| Семестр 1 |
| Семестр 2 |
| Семестр 3 |
| Семестр 4 |
| О себе |
| Ссылки |
Поиск сигналов
Задание 1.
В этом задании нужно было сравнить предсказанные регуляторные мотивы последовательностей программой MEME с эксперимнтальными данными, которые нам были выданы заранее. Результат можно посмотреть тут.
Мне были выданы фрагменты длины 200 нуклеотидов. В MEME я установила минимальный и максимальный размер мотива 16 нуклеотидов и запустила один раз с параметром «Zero or one per sequence» и один - с «One per sequence».
В результате последовательности нашлись у всех в обоих случаях. В 9 случаях из 12 они пересеклись с экспериментальными данными по 15 нуклеотидам. Если в параметрах выставить не 16, а 15 нуклеотидов, то это как раз и оказываются соответствующие пересечения, что говорит о том, что эти 15 нуклеотидов дают хороший сигнал. Последний нуклеотид в экспериментальных данных всегда выше по цепи оказывается, а в предсказанных - ниже. Для двух последовательностей нечего сравнивать (у них и p-value на один порядок хуже, чем нужно), а в одной предсказанный мотив оказался далеко от экспериментального (возможно, там несколько мотивов).
Можно заметить, что найденные мотивы полиндромы не полностью, зато очень похожи друг на друга. LOGO для найденных мотивов показано на рис 1.
Рис 1. LOGO, построенное программной MEME для найденных мотивов.
Задание 2.
Задание выполнено вместе с Ксенией Худяковой.
Мы выбрали сервис rVista. Это программа, осуществляющая поиск регуляторных элементов в геноме человека. Она основывается на предсказании сайтов связывания транскрипционных факторов и сравнении этих последовательностей с другими организмами. На вход сервису подаются парные выравнивания, сделанные с помощью blastz или zPicture. Так же можно найти уже сделанные парные выравнивания в базах данных ECR и GALA. По функциям rVista похож на базу данных TRANSFAC, но последняя основывается только на предсказании с использованием матрицы весов, в то время как rVista сочетает этот подход с оценкой консервативности кандидатов. Благодаря этому сильно снижается количество ложноположительных результатов.
Схема работы программы такова: 1. Построение выравнивания человек-мышь нужного участка с помощью программы AVID.
2. Предсказание программой Match (основывается на библиотеке TRANSFAC Professional 9.2) возможных сайтов связывания на обеих последовательностях независимо. После чего отбираются только те сайты, которые находятся друг напротив друга в выравнивании (возможен сдвиг не более чем на 6 оснований и не более одного гэпа любой длины внутри сайта).
3. Следующий шаг - оценка консервативности участка, в котором находится предполагаемый сайт связывания транскрипционного фактора. Если его консервативность выше 80% и его размер больше 24 п.н., то сайт, находящийся в этом участке, заносится в список консервативных сайтов.
4. Если загружаемая последовательность аннотирована, то rVista выдаёт только те, консервативные сайты, которые находятся в некодирующих областях. Если последовательность не аннотирована, то выданы будут все консервативные сайты.Сейчас используют версию сервеса rVISTA 2.0. Статья, посвященная ему, опубликована в 2004 году и находится сейчас в открытом доступе по ссылке.
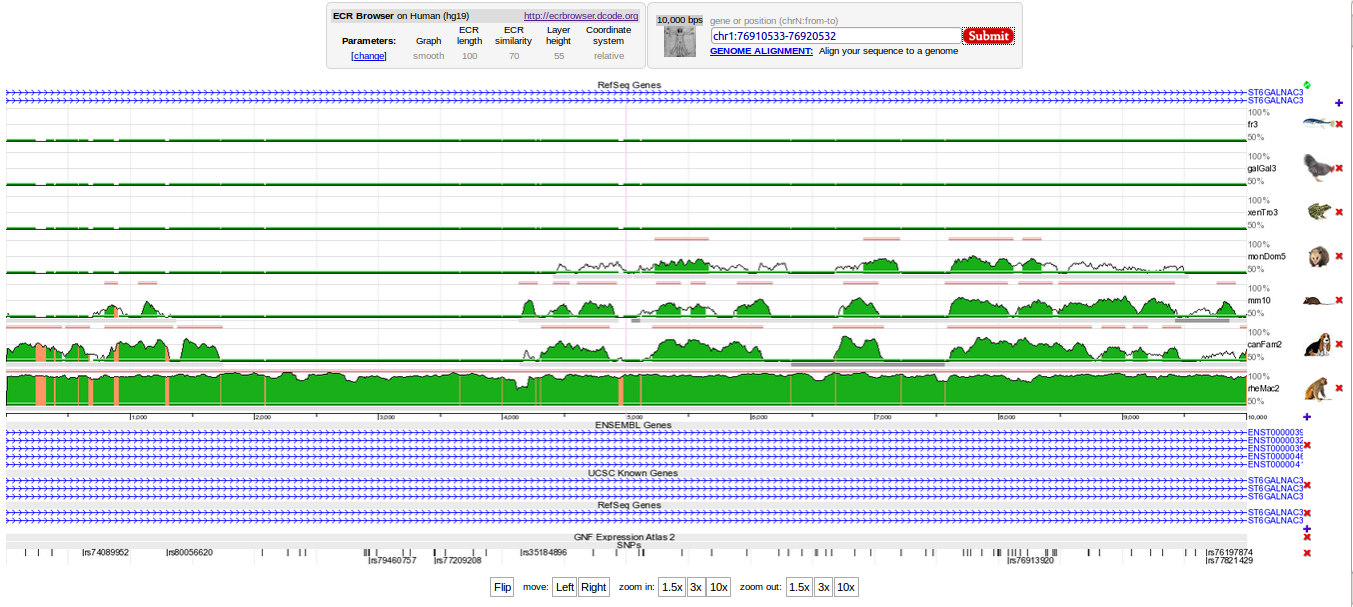
Далее покажем на примере типичное пользование программой. Мы взяли выравнивание, о котором говорится в статье про эту программу. Оно получено с помощью ECR Browser для человеческого локуса NKX2.5 (рис 2). Я выбрала участок выравнивания с геномом мыши и нажала на "conserved transcription factor binding sites (TFBS)".
Рис 2. Результат работы ECR Browser.
Затем надо было выбрать какие-нибудь (или все) предполагаемые транскрипционные факторы. Из статьи мы уже знали, что нам нужны транскрипционный фактор Smad.
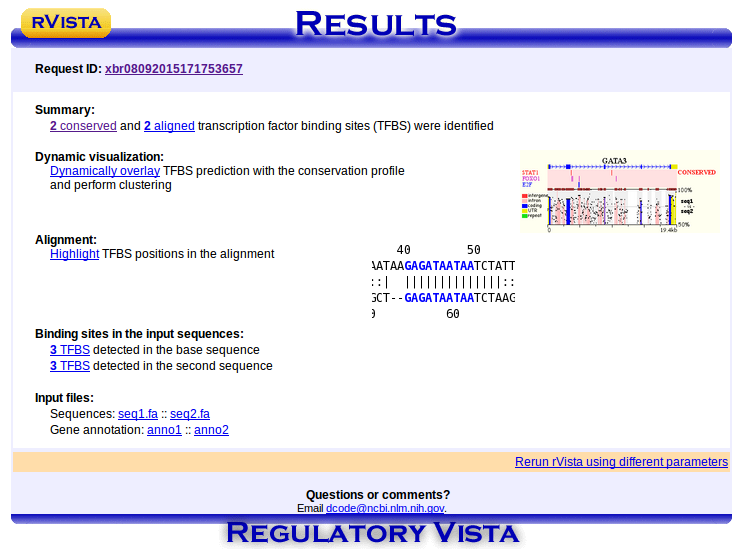
Программа rVista выдала различные данные (рис 3). Сверху можно увидеть ID запроса. В дальнейшем по нему можно будет восстановить результат. Затем ссылки на выровненные и консервативные транскрипционные факторы. Следующим пунктом идет ссылка на интерактивную базу данных сайтов связывания транскрипционных факторов. В строке про выравнивания, если перейти по ссылке, можно воспользоваться визуализацией выравнивания. Далее можно посмотреть отдельно для каждой цепи. А последней строкой предлагается скачать или посмотреть файлы с результатами. Подробная удобная инструкция есть по этой ссылке.
Рис 3. Результат работы rVista.