|
|
Совмещение структур
Совмещение структур
Для поиска структурных гомологов и построения совмещения была выбрана РНК-метилтрансфераза из Yersinia pestis
(PDB 3N4J). Результат работы программы PDBeFold на рис. 1, галочкой отмечены структурные гомологи, которые я затем использовала.
У них RMSD в диапазоне от 0.8 до 3 и процент совпадения последовательностей от 50 до 90.
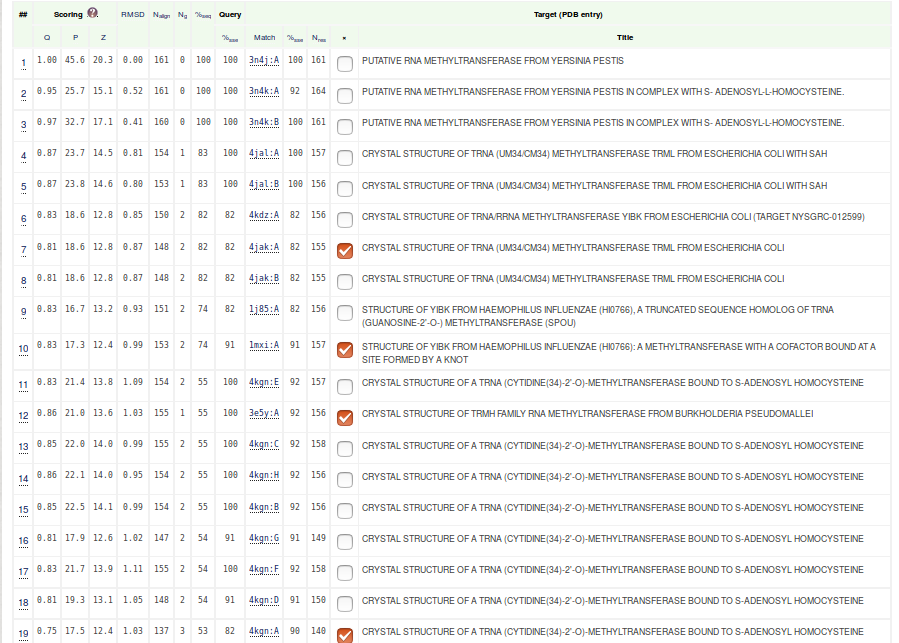
Рис 1. Структурные гомологи РНК-метилтрансферазы 3N4J.
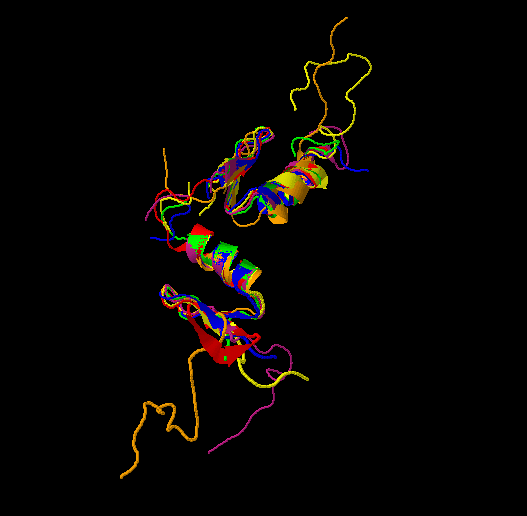
PDBeFold выдает структурное выранивание в формате Rasmol и fasta. На рис. 3. показано это выравнивание полученное с помощью
PyMol. Видно, что структуры достаточно похожи, хотя у одной нет двух α-спиралей и у двух есть дополнительный
β-лист (рис. 2).
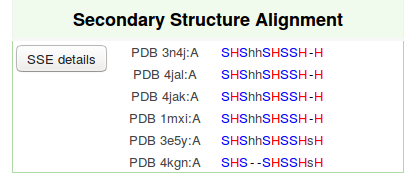
Рис 2. Элементы вторичной структуры гомологов.

|
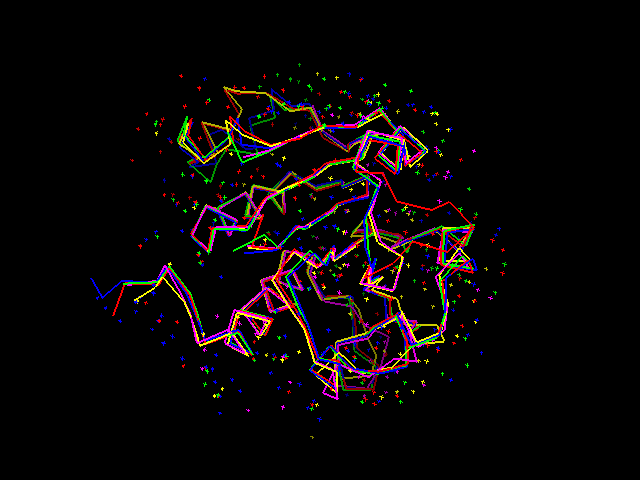
|
| Рис 3. Белок 3N4J и его структурные гомологи. |
С помощью Jalview я построила выранивание последовательностей этих белков (Muscle whith Default) (рис. 4 левый, нижнее
выравнивание) и открыла структурное выравнивание (рис. 4 левый, верхнее) для сравнения. В целом они похожи, но я выбрала
подчеркнутое место, где в двух нижних структурах серины и глицины выравнены по-разному. Это же место я посмотрела в
структурном выравнивании (рис. 4 правый), по которому видно, что правильнее было бы совместить серин с серином и соответствующий
глицин с глицином.
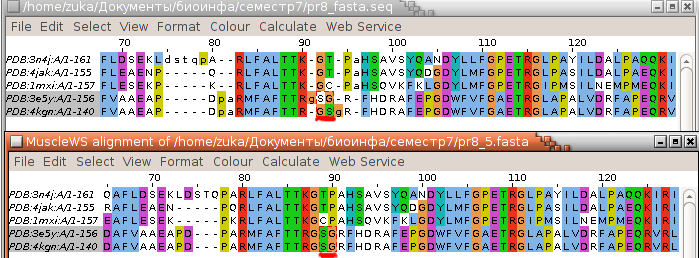
|
|
| Рис 4. Выравнивания по последовательности и структурное (слева) и совмещение выделенного
участка (справа). |
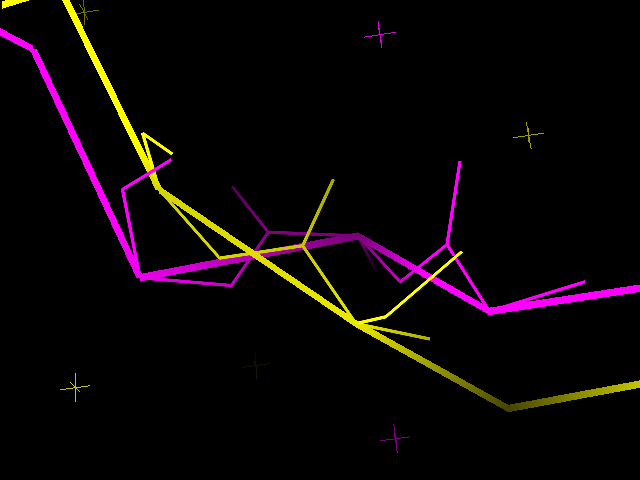
Гибкое выравнивание
Я взяла один из примеров, приведенных на сервере Posa: выранивание
доменов, содержащих цинковые пальцы, различных белков (2drp:A; 2d9h:A; 2adr:A; 4gzn:C; 1x6e:A; 2cot:A). По данным
Posa при гибком выравнивании совпало 49 аминокислот (рис. 5 слева). При этом при жестком выравнивании совпадений
обнаружено не было (рис. 5 справа).
|

|
| Рис 5. Гибкое выравнивание цинковых пальцев (слева) и жесткое выравнивание (справа). |
|







