Нуклеотидный BLAST
1. Определение функции и таксономии нуклеотидной последовательности
Для того, чтобы предположить функцию нуклеотидной последовательности, полученной мною при расшифровке хроматограммы, и таксономическое положение организма, геному которого она принадлежит, мною был выбран blastn, так как заранее про последовательность совсем ничего не известно.
Параметры запуска были следующие: база данных - nr/nt (т.к непонятно, есть ли интересующая нас последовательность среди аннотированных), word size изменён на минимальный возможный (7) для того, чтобы повысить чувствительность поиска. Также были исключены лишь предсказанные последовательности (Exclude Models (XM/XP)). Остальные же параметры установлены по умолчанию. Выдачу программы можно посмотреть по ссылке.
Исходя из найденных последовательностей, искомая последовательность с высокой вероятностью кодирует гистон H3. Также, судя по всему, наша последовательность является лишь частью гена, кодирующего H3 (на это указывает подпись partial во всех находках).
Находки принадлежат очень разным организмам. Так, в выдаче среди даже первых десяти находок встречаются, например, последовательности таких далёких организмов, как Ophelia limacina и Asterias rubens - представителей первично- и вторичноротых соответственно. Вполне объяснимо, почему так произошло: гистоны - сильно консервативные белки, ровно как и кодирующие их нуклеотидные последовательности.
Из-за такого многообразия сложно вполне точно определить таксономию организма, геному которого принадлежит наша последовательность. Я выбрала 10 лучших находок и посмотрела для них вкладку по таксономии. С большой вероятностью последовательность, полученная при анализе хроматограммы, принадлежит организму из семейства кольчатых червей Opheliidae (5 находок из топ 10). Для определения вида я скачала выравненные последовательности и визуализировала их в Jalview. Результат представлен на Рис.1. Можно заметить, что вторая последовательность, принадлежащая Ophelia limacina и являющаяся первой в списке находок, "выбивается" из выравнивания. Следовательно можно предположить, что наша последовательность вряд ли принадлежит особи этого вида. В остальных же последовательностях число замен равномерно одинаково, поэтому видовую принадлежность определить затруднительно.

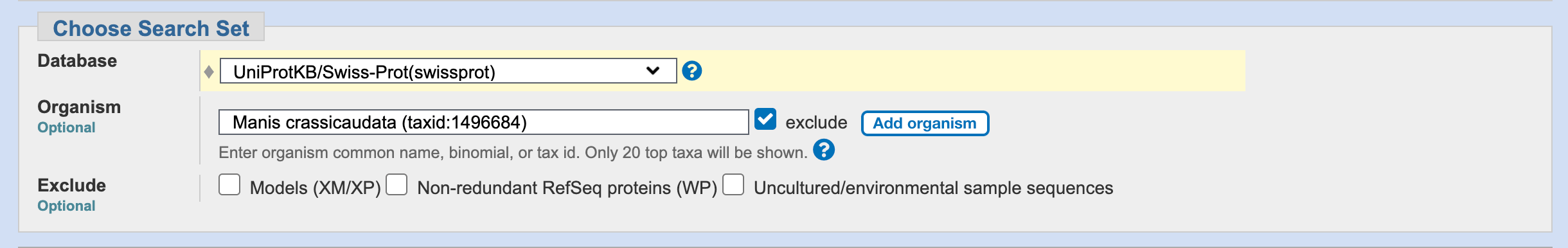
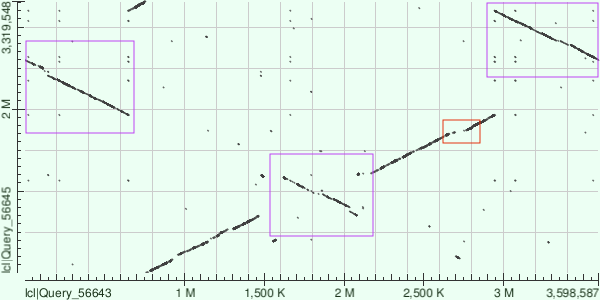

Рисунок 1. Выравнивание последовательностей, принадлежащих представителям семейства Opheliidae из топ 10 находок.
2. Поиск генов белков в неаннотированной нуклеотидной последовательности
Для данного задания я выбрала контиг Manis crassicaudata длиной около 50 тысяч нуклеотидов. Далее для определения функции и таксономии последовательности был произведен поиск в blastx, так как именно он позволяет по нуклеотидным последовательностям найти белки.
Параметры запуска были следующие: значение word size было выбрано наименьшее (2) для увеличения чувствительности поиска, были исключены последовательности, относящиеся к Manis crassicaudata, а также для поиска была выбрана база данных SwissProt, чтобы данные с большей вероятностью были достоверные (см. Рис.2). Выдачу программы можно посмотреть по ссылке.

Рисунок 2.
У находок достаточно низкий e-value, уровень покрытия около 1% (что неудивительно, так как размер контига весьма значительный), а процент индентичности в некоторых находках достигает 100%. Все найденные последовательности кодируют белок CASPR - мембранный белок, экспрессируемый в нейронах и, веротяно, участвующий в их миелинизации [Rios, J C et al., 2000]. С большой вероятностью можно предположить, что в искомой последовательности закодирован именно этот белок.
3. Интепретация карты локального сходства гомологичных хромосом двух бактерий
Для этого задания мною были выбраны сборки родственных бактерий Acinetobacter baylyi и Acinetobacter soli .
Чтобы построить карту локального сходства, я воспользоволась megablast (так как цель - сравнить близкие последовательности) с параметрами по умолчанию. Выдача доступна по ссылке.

Рисунок 3. Карта локального сходства хромосом Acinetobacter soli и Acinetobacter baylyi
На полученном графике из крупных перестроек хорошо заметны инверсии (выделены фиолетовым). Также присутствуют непротяженные индели (пример выделен красным). Точками показаны короткие неконсервативные участки.