Что такое филогенетическое дерево
Список выбранных бактерий:
| Название | Мнемоника |
| Bacillus subtilis | BACSU |
| Clostridium tetan | CLOTE |
| Enterococcus faecalis | ENTFA |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus acidophilus | LACAC |
| Lactobacillus delbrueckii | LACDA |
| Listeria monocytogenes | LISMO |
| Staphylococcus epidermidis | STAES |
- Cкобочная формула
(((((BACSU, GEOKA), LISMO), STAES), ((LACDA, LACAC), ENTFA)), CLOTE); - Изображение:
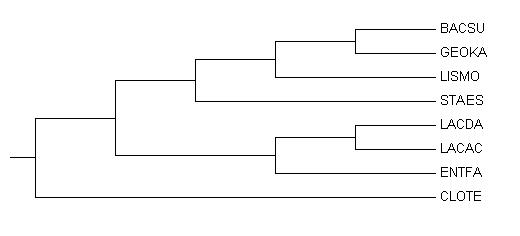
- Cписок нетривиальных ветвей как разбиений множества листьев
1.{BACSU, GEOKA} vs {LISMO, STAES, LACDA, LACAC, ENTFA, CLOTE};
2.{BACSU, GEOKA, LISMO} vs { STAES, LACDA, LACAC, ENTFA, CLOTE};
3.{BACSU, GEOKA, LISMO, STAES} vs { LACDA, LACAC, ENTFA, CLOTE};
4.{LACDA, LACAC} vs {BACSU, GEOKA, LISMO, STAES, ENTFA, CLOTE};
5.{LACDA, LACAC, ENTFA} vs {BACSU, GEOKA, LISMO, STAES, CLOTE};
Реконструкция филогенетических деревьев
- Таксономия выбранных мной бактерий, найденная с помощью таксономического сервиса NCBI http://www.ncbi.nlm.nih.gov/taxonomy/:
Bacillus subtilis :
cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus;
Clostridium tetan :
cellular organisms; Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium
Enterococcus faecalis:
cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus
Geobacillus kaustophilus:
cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus
Lactobacillus acidophilus:
cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus
Lactobacillus delbrueckii:
cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus
Listeria monocytogenes:
cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria
Staphylococcus epidermidis:
cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus
Первая ветка соответствует таксону Bacillaceae, третья – Bacillales, четвертая- Lactobacillus а пятая - Lactobacillales. Втора ветвь ничего существенного не отделяет.Я выбрала семейство белков LEPA (Фактор элонгации трансляции 4) и создавала выравнивание с помощью программы muscle
- Скобочная формула дерева, созданного программой fprotpars(программа выдала только одно дерево):
(((((((LISMO,ENTFA),STAES),BACSU),GEOKA),CLOTE),LACDA),LACAC); - Изображение :
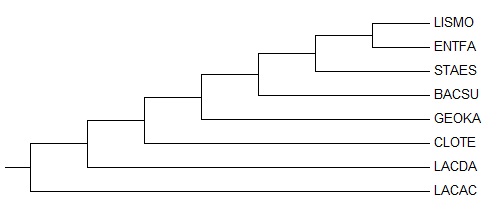
Замечание: У этого дерева с "правильным" совпадает только одна ветвь -
{LACDA, LACAC} vs {BACSU, GEOKA, LISMO, STAES, ENTFA, CLOTE}; - Матрица расстояний, полученная программой fprotdist:
----------- ---LACAC--- ---LACDA--- ---CLOTE--- ---STAES--- ---ENTFA--- ---LISMO--- ---BACSU--- ---GEOKA--- LACAC 0.000000 0.120965 0.591230 0.529388 0.408304 0.472016 0.470013 0.437770 LACDA 0.120965 0.000000 0.607378 0.544579 0.459744 0.480280 0.475593 0.488149 CLOTE 0.591230 0.607378 0.000000 0.574039 0.477361 0.448340 0.427136 0.391489 STAES 0.529388 0.544579 0.574039 0.000000 0.293306 0.283476 0.281272 0.291492 ENTFA 0.408304 0.459744 0.477361 0.293306 0.000000 0.188812 0.250032 0.246539 LISMO 0.472016 0.480280 0.448340 0.283476 0.188812 0.000000 0.174179 0.224005 BACSU 0.470013 0.475593 0.427136 0.281272 0.250032 0.174179 0.000000 0.169548 GEOKA 0.437770 0.488149 0.391489 0.291492 0.246539 0.224005 0.169548 0.000000
Оценка ультрамеричности:- LACAC=A, LISMO=B, BACSU=C
d(A,C)=0.470013; d(A,B)=0.472016; d(B,C)=0.174179;
в(A,C) практически равно d(A,B) (отклонение на 0.002003) и эти расстояния больше третьего - ENTFA=A, LACAC=B, LACDA=C
d(A,C)=0.459744; d(A,B)=0.408304; d(B,C)=0.120965;
В этом случае отклонение от ультрамеричности больше (d(A,C) отличается от d(A,B) больше, чем на 0.05)
Оценка аддитивности:- GEOKA=A, LACAC=B, LACDA=C, CLOTE=В
d(A,B)+d(C,D)=1.045148;
d(A,C)+d(B,D)=1.079379;
d(A,D)+d(C,B)=0.512454;
Две суммы из трех почти равны и больше третьей
- LACAC=A, LISMO=B, BACSU=C
- Деревья, построенные программой fneighbor
Алгоритм Neighbor-Joining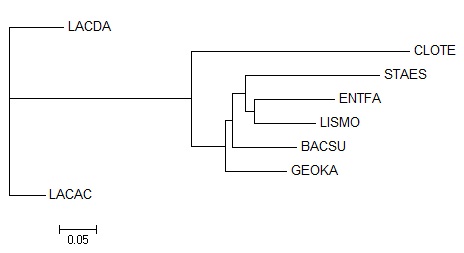
Алгоритм UPGMA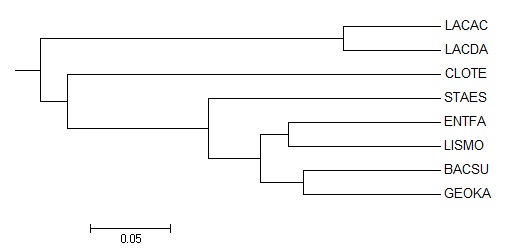
- Сравнение деревьев
Деревья, полученные с помощью алгоритмов Neighbor-Joining и UPGMA совпадают тремя ветвями:
{LACAC, LACDA} vs {CLOTE, STAES, ENTFA, LISMO, BACSU, GEOKA};
{LACAC, LACDA, CLOTE} vs {STAES, ENTFA, LISMO, BACSU, GEOKA};
{ENTFA, LISMO} vs {CLOTE, STAES, LACAC, LACDA, BACSU, GEOKA};
При этом неукорененное дерево ( алгоритм Neighbor-Joining) совпадает с "правильным" только одной ветвью
{LACAC, LACDA} vs {CLOTE, STAES, ENTFA, LISMO, BACSU, GEOKA};
А укорененное - двумя
{LACAC, LACDA} vs {CLOTE, STAES, ENTFA, LISMO, BACSU, GEOKA};
{BACSU, GEOKA} vs {CLOTE, STAES, ENTFA, LISMO, LACAC, LACDA};
Получилось, что программа fneighbor в данном случае не намного точнее, чем fprotpars.