Построение дерева по нуклеотидным последовательностям
Задание: построить филогенетическое дерево тех же бактерий, что в предыдущих заданиях, используя последовательности РНК малой субъединицы рибосомы (16S rRNA)
- Последовательности 16S рибосомной РНК добывала с помощью банка EMBL
и программы seqret embl:AC -sask, где АС - acception number записи EMBL, описывающий
соответствующий геном бактерии.
Организм AC Координаты вырезанных участков LACAC CP000033 1632689-1634260 (COMPLEMENT) LACDA CR954253 45160-46720 BACSU AL009126 30279-31872 ENTFA AE016830 248466-249987 GEOKA BA0000043 10421-11973 LISMO CP002004 241587-243142 STAES AE015929 1598006-1599559 (COMPLEMENT) CLOTE AE015927 8715-10223 (COMPLEMENT) - Выровняла последовательности с помощью программы muscle
- Построила дерево с помощью программы fdnaml. Она выдала
неукорененное дерево с длиннами ветвей:
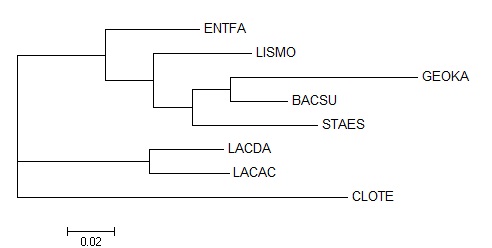
Полученное дерево оказалось "лучше"деревьев, построенных по белку Lepa, и имеет три ветви, общих с "правильным":
{LACDA,LACAC} против всего остального;
{GEOKA,BACSU} против всего остального;
{GEOKA, BACSU,STAES,LISMO} против всего остального;
Задание: найти в своих бактериях достоверные гомологи белка CLPX_BACSU. Построить дерево этих гомологов. Считая дерево реконструированным верно, указать несколько пар ортологов и несколько пар паралогов.
Была сделана база данных для бласта из файла proteo.fasta, затем по этой базе данных были найдены гомологи белка CPLX_BACSU (evalue - 0.001) Затем были отобраны белки, принадлежащие выбранным мной бактериям. Дерево построено программой fprotpars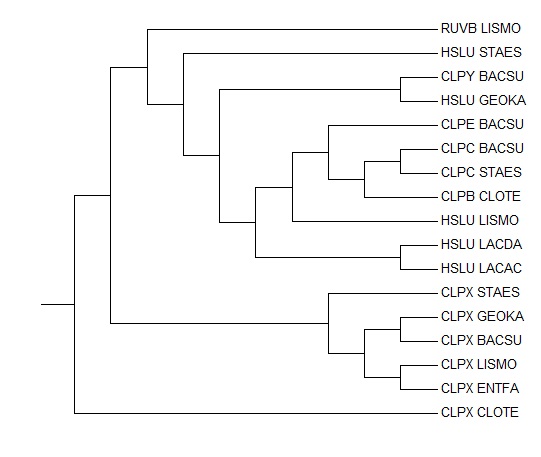
- Примеры ортологов:
- CLPC_BACSU & CLPC_STAES
- HSLU_STAES & HSLU_GEOKA
- Примеры паралогов:
- CLPE_BACSU & CLPY_BACSU
- CLPB_CLOTE & CLPX_CLOTE
- CLPC_STAES & CLPX_STAES
- Примеры ортологов:
