Суть задания ознакомиться с программой ZDOCK и сделать
предсказание о свзывании фрагмента антитела (VHH domain)
c панкреатической альфа-амилазой.
Файлы:
amilase.pdb
camelid.pdb
uniCHARMM
Сначала были добавлены водороды:
pdb2gmx -f amilase.pdb -o amilase_h.pdb -p -ignh pdb2gmx -f camelid.pdb -o camelid_h.pdb -p -ignh
С помощью утилиты mark_sur проведен препроцессинг файлов pdb.
mark_sur amilase_h.pdb amilase_h_m.pdb mark_sur camelid_h.pdb camelid_h_m.pdbРезультаты:amilase_h_m.pdb, camelid_h_m.pdb. Утилита mark_sur добавляет информацию о радиусе, заряде и типе атомов. Для дальнейшей работы из выходных файлов были удалены строчки MODEL и TER. И, собственно, запущен докинг:
zdock -R amilase_h_m.pdb -L camelid_h_m.pdbРезультирующий файл - zdock.out. Для работы утилиты zrank необходимо, чтобы в pdb-файлах перед строчками, описывающими атомами была хотя бы одна пустая строка. Так же нужно удалить вторую строчку из zdock.out.
zrank zdock.out 1 2000Результат - таблица, где напротив каждой модели указан ее вес.
Создание pdb-файлов моделей:
create.pl zdock.outДля работы этой утилиты нужен файл create_lig. Так же понадобилось удалить третий столбец в первой строчке файла zdock.out. Тогда результатом работы create.pl является 1999 pdb-файлов моделей. Я открыла 5 случайных файлов, в которых связывание белков достаточно разнообразны. По оценкам zrank score было отобрано 3 файла, имеющих наименьшую энергию. Изображения можно будет увидеть ниже. Затем, с помощью g_rms, было посчитано rmsd каждой моели относительно известной структуры 1kxt. (Очень важно перед началом работы удалить водороды из pdb файлов)
for i in {1..2000}; do sed /^$/d ../complex.${i}.pdb > ${i}.pdb;
babel -d -ipdb ${i}.pdb -opdb ${i}_noh.pdb ;
echo -e "1\n1\n" | g_rms -f ${i}_noh.pdb -s ../kxt.pdb -o rmsd${i};
echo -n "${i} " >> RMS;
grep ' -1.0000000' rmsd${i}.xvg >> RMS;
done
№ zrank score RMSD ################################## 1588 -28,661 2,5344827 1252 -27,5357 1,6861778 610 -25,5514 1,9159631 1879 0 1,4554459 1114 0 1,4688028 1182 0 1,4694291Первые три структуры имеют лучшую энергию, а вторая тройка - самое минимальное квадратичное отклонение от 1kxt.
Изображения:
| 1588 | 1252 | 610 |
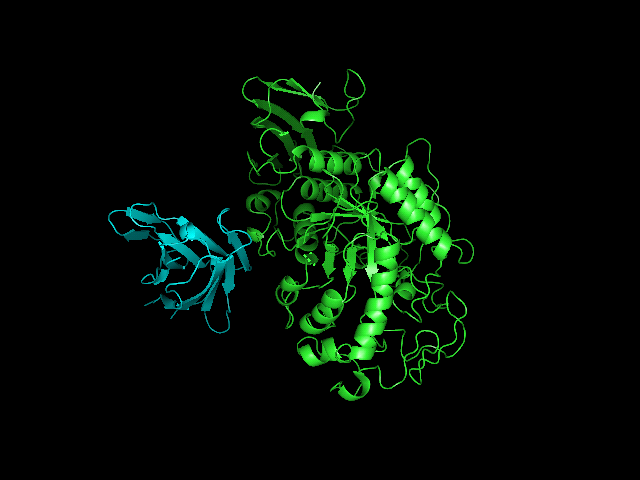
|
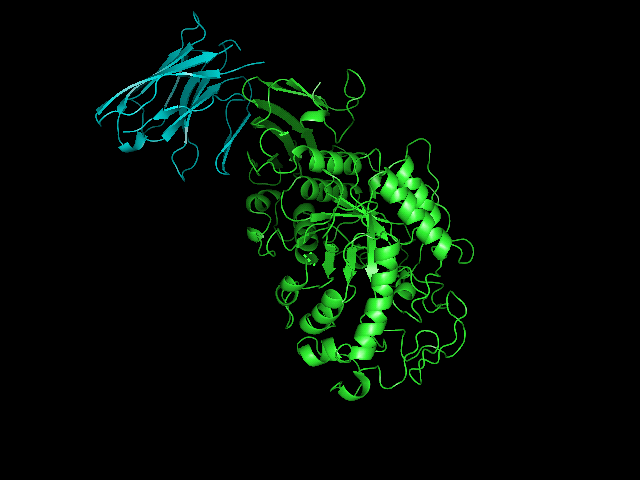
|
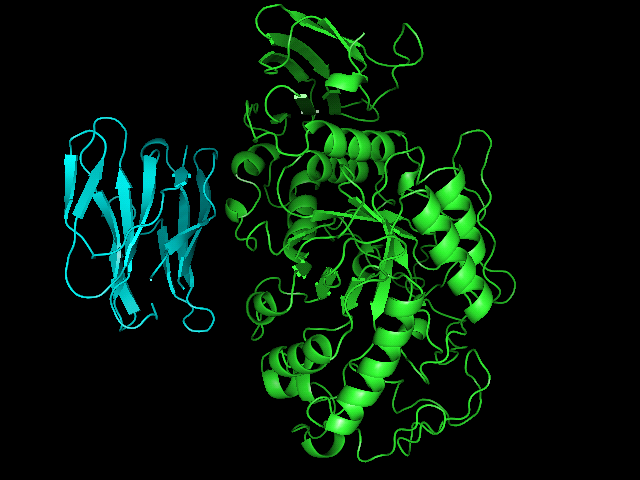
|
| 1879 | 1114 | 1182 |
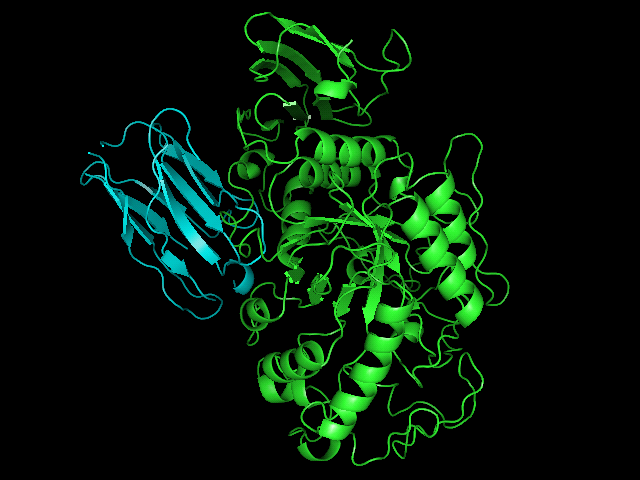
|
Очень странная структура записана в этом файле. | Очень странная структура записана в этом файле. |
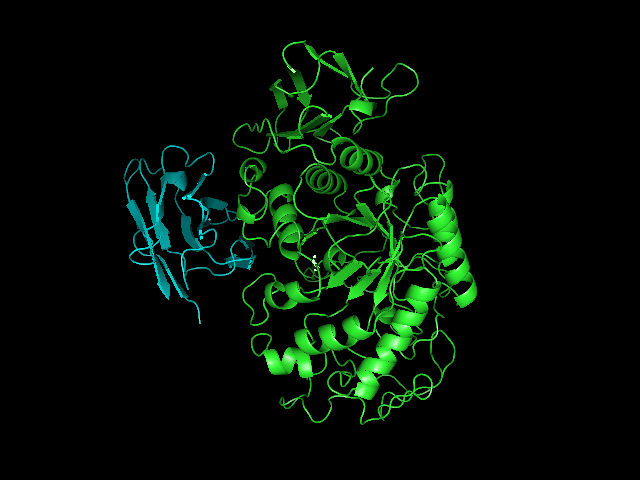 Видно, что на рефересную структуру больше всего похожи 610 и 1879 модели.
Хотя вариация достаточно большая. 114 и 1182 модели вообще какие-то странные и не отображают
структуры комплекса.
Вообще, можно сказать, что резуьтаты получились не очень хорошие,
возможно, имеет смысл использовать -D для докинга с помощью zdock
Видно, что на рефересную структуру больше всего похожи 610 и 1879 модели.
Хотя вариация достаточно большая. 114 и 1182 модели вообще какие-то странные и не отображают
структуры комплекса.
Вообще, можно сказать, что резуьтаты получились не очень хорошие,
возможно, имеет смысл использовать -D для докинга с помощью zdock