Изучение результатов работы на суперкомпьютере
Визуальный анализ движения молекул
Конвертирование в pdb-формат:
trjconv -f dna_md.xtc -s dna_md.tpr -o dna_pbc_1.pdb -skip 20 -pbc mol
Полученный pdb файл
Переход из A в B форму по визуальному анализу происходит на 46 модели (t=9000.0000)
Определение средне-квадратичного отклонения в ходе моделирования. Из-за конформационного перехода сначала расчитано отклонение в ходе всей симуляции относительно стартовой структуры.
g_rms -f dna_md.xtc -s dna_md.tpr -o rms_1
И относительно каждой предидущей структуры на растоянии 400 кадров.
g_rms -f dna_md.xtc -s dna_md.tpr -o rms_2 -prev 400
Интересно, что ближе к концу отклонение не стало уменьшаться
(файл), что может говорить о том,
что переход из А в В форму произошел не до конца.
Определение изменения гидрофобной и гидрофильной поверхности в ходе конформационного перехода.
g_sas -f dna_md.xtc -s dna_md.tpr -o sas_dna.xvg
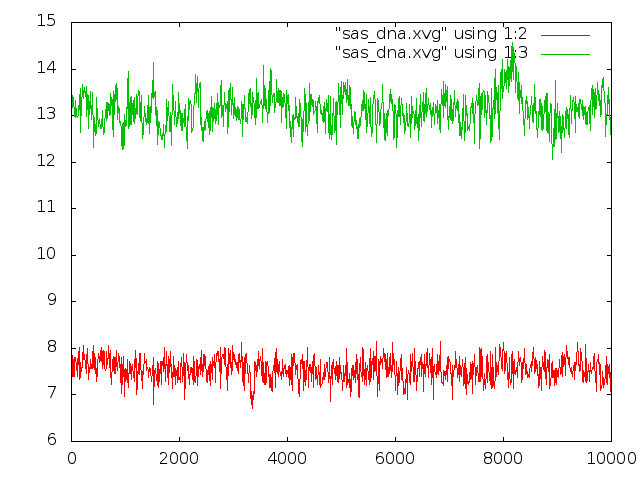 Видно, что гидрофобная (красный) поверхность остается примерно одинаковой на
протяжении всего моделирования, когда гидрофильная имеет пик ближе к концу.
Видно, что гидрофобная (красный) поверхность остается примерно одинаковой на
протяжении всего моделирования, когда гидрофильная имеет пик ближе к концу.
Расчёт колчества образуемых водородных связей.
Используемая команда:
g_hbond -f dna_md.xtc -s dna_md.tpr -num hbond_dna
Аналогичным образом было рассчитано количество водородных связей ДНК-Вода.
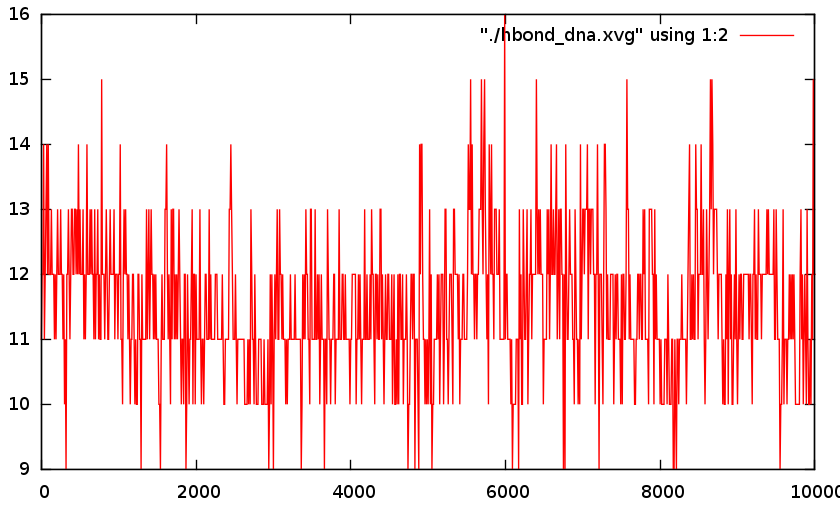
 На первом графике видно, что количество водородных связей меняется в процессе перехода,
но, в конце концов возвращается к исходному значению (около 12).
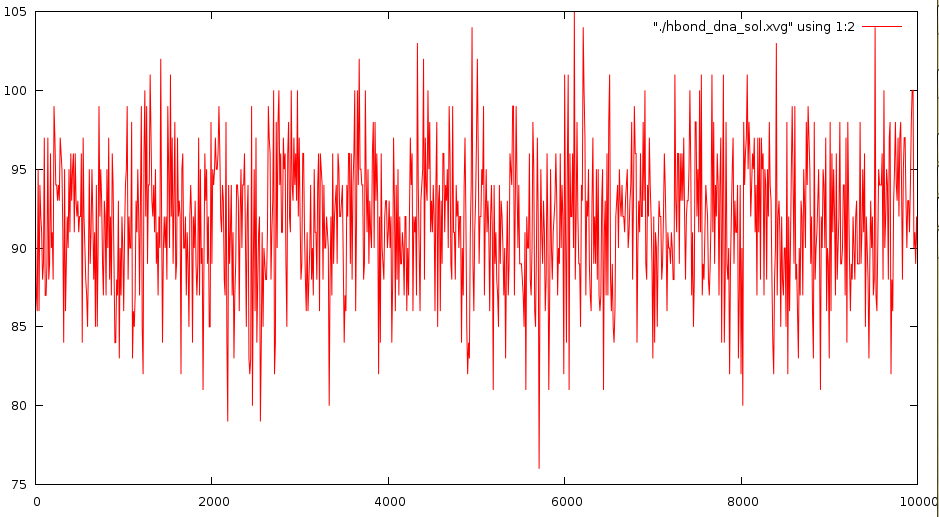
Второй график, показывающий количесвто водородных связей ДНК-вода,
имеет небольшую тенденцию к росту.
На первом графике видно, что количество водородных связей меняется в процессе перехода,
но, в конце концов возвращается к исходному значению (около 12).
Второй график, показывающий количесвто водородных связей ДНК-вода,
имеет небольшую тенденцию к росту.