На главную страницу четвертого семестра
Классификация функций. Ферменты.
Расшифровка кода фермента.
EC 1 - оксидоредуктазы
EC 1.2 - взаимодействующие с альдегидной или оксо-группой
EC 1.2.1 - С использованием в качестве акцептора НАД+ или НАДФ+
EC 1.2.1.12 - глицеральдегид-3-фосфатдегидрогеназа (фосфорилирующая) [glyceraldehyde-3-phosphate dehydrogenase (phosphorylating)]
Даты.
Использование средств специализированного ресурса Brenda для характеристики фермента EC 1.2.1.12.
Схема реакции:
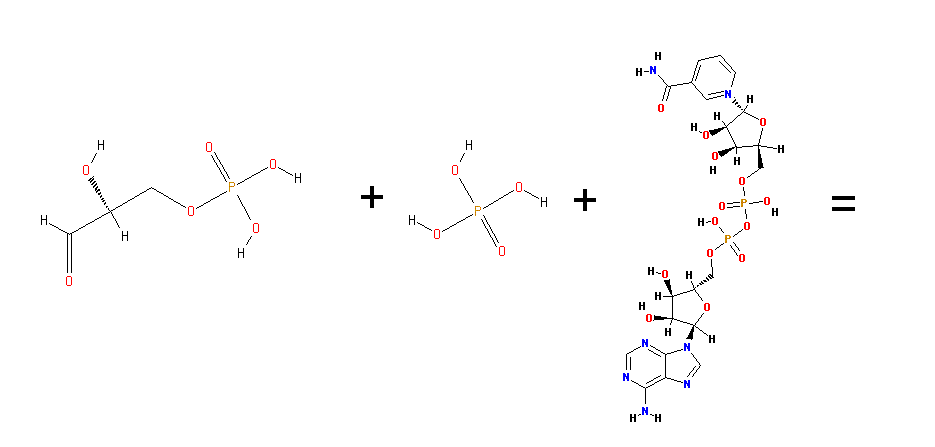
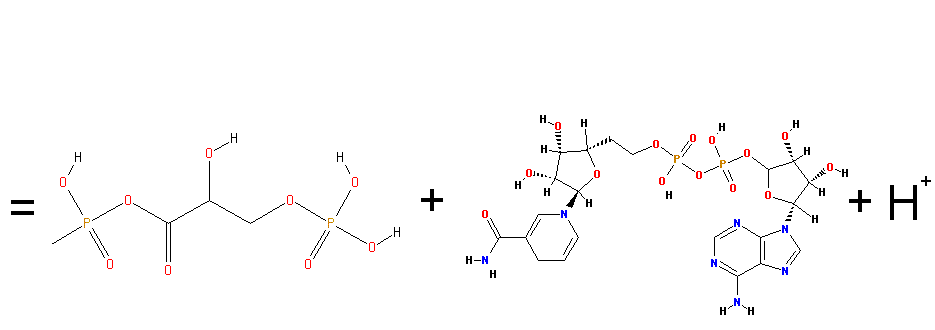
Общее число ферментов с данным кодом : 672
Оптимум pH лежит в интервале 7 - 10.
Примеры ингибиторов ферментов с данным кодом :
(NH4)2SO4 ; 2,3-Дифосфоглицерат ; 6-Фосфоглюканат ; АДФ ; АТФ ; CdCl2 ; Cu2+ ; CuSO4 и т.д.
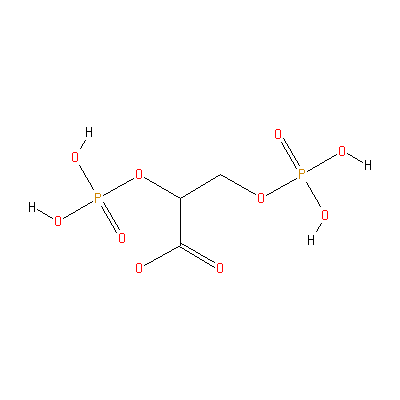 |
2,3-Дифосфоглицерат |
Примеры активаторов ферментов с данным кодом :
2,3-Димеркаптопропанол ; ацетон ; ЭДТА ; тетрабутиламмонийхлорид и т.д.
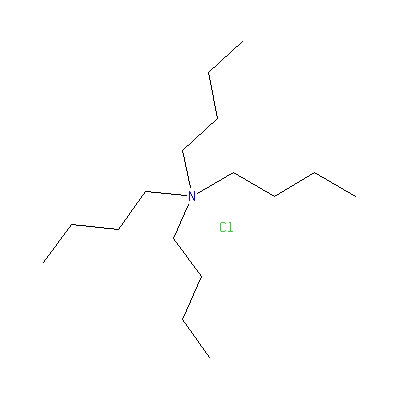 |
Тетрабутиламмонийхлорид |
- Болезни человека, так или иначе связанные с данным ферментом - адренолейкодистрофия(Adrenoleukodystrophy), гиперглицемия(Hyperglycemia)
- Субъединичная структура, характерную для данного типа ферментов - димер,тетрамер,октамер
- Пострансляционные модификации, характерные для данного типа ферментов - фосфопротеин, протеолитическая модификация
Сравнение ферментов с одинаковым кодом из эволюционно далеких организмов
По заданному коду EC 1.2.1.12 с помощью SRS в SWISS–Prot искались ферменты по трём хорошо изученным организмам - кишечной палочки Escherichia coli K-12, человека и Methanococcus jannaschii.
Запрос: Query "([swissprot-ECNumber:1.2.1.12*] & (([swissprot-ID:*_Ecoli*] | [swissprot-ID:*_Human*]) | [swissprot-ID:*_Metja*])) " found 4 entries
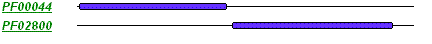
По данному запросу было найдено по 2 последовательности из кишечной палочки и человека.В каждой из них содержались домены с идентификаторами PF00044 и PF02800.По архее Methanococcus jannaschii ничего не было найдено,
поэтому поиск ферментов с данным кодом проводился по другим эволюционно далеким организмам в соответствии с запросом:
Query "(([swissprot-ECNumber:1.2.1.12*] ! [swissprot-Taxonomy:BACTERIA*]) ! [swissprot-Taxonomy:VERTEBRATA*]) " found 114 entries.
Было найдено 114 последовательностей с такими же доменами.Из низ был выбран белок G3P1_AGABI - фермент представителя базидиомицетов.Для дальнейшего сравнения
из всех 5 белков были выделены гомологичные домены PF00044 с помощью команды
extractseq <входящий файл> <исходящий файл> -regions <начало домена>—<конец домена>
Положение доменов PF00044 в белковых последовательностях и имена файлов с готовыми доменами:
| ID |
Положение домена |
Файл с доменом |
| G3P1_ECOLI |
2-149 |
1.fasta |
| G3P3_ECOLI |
2-150 |
2.fasta |
| G3PT_HUMAN |
76-224 |
3.fasta |
| G3P_HUMAN |
3-151 |
4.fasta |
| G3P1_AGABI |
2-150 |
5.fasta |
Все найденные белки идентичны по доменной организации:
Затем для программы needle был составлен скрипт, позволяющий получить значения Identity для попарных выравниваний:
needle 1.fasta 2.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
needle 1.fasta 3.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
needle 1.fasta 4.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
needle 1.fasta 5.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
needle 2.fasta 3.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
needle 2.fasta 4.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
needle 2.fasta 5.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
needle 3.fasta 4.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
needle 3.fasta 5.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
needle 4.fasta 5.fasta -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity
Выравнивания
На основе полученных данных была построена таблица значений Identity попарных выравниваний доменов
PF00044 следующих белков:
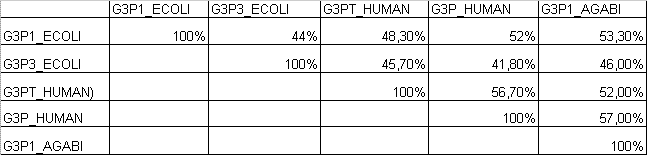
Полученные проценты Identity ( >40% ) позволяют предположить, что данные белки выполняют схожие функции
и у кишечной палочки, и у человека, и у гриба.Также видно, что у человека существует 2 белка с одинаковой функцией,но с Identity 56,7%.Можно предположить что
эти белки функционируют в разных органах.
Что можно найти в ENZYME?
ENZYME - база данных номенклатуры ферментов Швейцарского Института Биоинформатики.В ней, как написано
в пояснении, можно найти следующую информацию:
- Код EC
- Основное название
- Альтернативные названия (если есть)
- Каталитическая активность
- Кофакторы (если есть)
- Указание на записи банка данных Swiss-Prot, которые связаны с ферментом (если есть)
- Указание на болезни, связанные с ферментной недостаточностью (если есть)
На главную страницу четвертого семестра.