На главную страницу
Классификация функций. Ферменты.
Код фермента и его обозначения.
EC 1.3.3.4
EC 1 - оксидоредуктазы
EC 1.3 - действуют на CH-CH группы доноров
EC 1.3.3 - содержат кислород в качестве акцептора
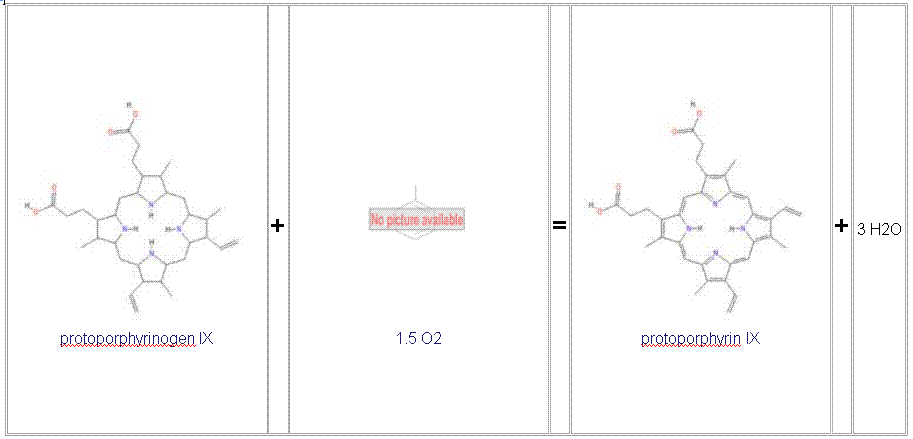
EC 1.3.3.4 - протопорфирогенная оксидаза
Характеристика фермента EC 1.3.3.4 с помощью специализированного ресурса Brenda.
Ниже приеден рисунок со схемой ферментативной реакции данного фермента.

Общее число ферментов с таким кодом равно 89.
У ферментов с данным кодом бывают нижеследующие ингибиторы и активаторы.
Билирубин.
Активаторы:
Структура запроса: ([swissprot-ECNumber:1.3.3.4*] & (([swissprot-ID:*_Ecoli*] | [swissprot-ID:*_Human*]) | [swissprot-ID:*_Metja*])) По данному запросу было найдено два белка из кишечной палочки и человека, а в археи такого не оказалось.
Сравнивались только домены Pfam.
| идентификатор | положение в последовательности |
| PF00258 | 5-56 |
| PF01593 | 12-471 |
Если белки имеют одинаковый ЕС номер, то это означает, что они действуют по одному и тому же механизму. Но найденные домены - негомологичны. Получается, что разные белки выполняют одну и ту же функцию. Возможно это проявление конвергенгенции в эволюции.
Нет смысла делать глобальное выравнивание. Локальное выравнивание программой было сделано программой water. Идентичность составляет 35.5%.
Такие результаты, и то что, данного белка не оказалось в археях могут говорить о том, что этот фермент хорошо эволюционировал и в таком виде имеется только у человека. Но в кишечной палочке имеется домен сходный(по механизму действия) с доменом у человека. При этом можно предположить, что тот участок, который у них совпадает(с идентичностью 35.5%) является важным и отвечает за основную функцию этого фермента.
Последовательности доменов извлекались с помощью нижеследующего скрипта:
seqret sw:P0ACB4 -sask
seqret sw:P50336 -sask
Вравнивание было получено следующим скриптом:
water hemg_ecoli.fasta ppox_human.fasta ecoli_human.water -gapopen 10.0 -gapextend 0.5
Ниже приведено выравнивание:
| локальное выравнивание |
# Program: water
# Rundate: Tue Mar 14 2006 18:04:01
# Align_format: srspair
# Report_file: human_ecoli.water
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: PPOX_HUMAN
# 2: HEMG_ECOLI
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 31
# Identity: 11/31 (35.5%)
# Similarity: 17/31 (54.8%)
# Gaps: 1/31 ( 3.2%)
# Score: 32.0
#
#
#=======================================
PPOX_HUMAN 64 VSELGLDSEVLPV-RGDHPAAQNRFLYVGGA 93
:.|||:.::|..| |.:.|..:|....|.||
HEMG_ECOLI 21 LKELGIQADVANVHRIEEPQWENYDRVVIGA 51
|