
Матрица попарных расстояний:
* была получена с помощью программы ednadist. Предварительно было построено выравнивание последовательностей , соответствующих листьям начального дерева.
6
A 0.0000 0.8631 1.6199 1.6920 1.6062 1.7719
B 0.8631 0.0000 1.5172 1.5668 1.3956 1.6062
C 1.6199 1.5172 0.0000 0.3470 0.4351 0.6504
D 1.6920 1.5668 0.3470 0.0000 0.4700 0.6619
E 1.6062 1.3956 0.4351 0.4700 0.0000 0.6355
F 1.7719 1.6062 0.6504 0.6619 0.6355 0.0000
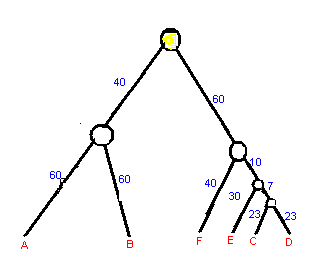
Реконструкция методом UPGMA:
*программа eneighbor
+-------------------------A
+---------------------4
! +-------------------------B
!
--5 +----------C
! +--1
! +-----2 +----------D
! ! !
+---------------------------3 +-------------E
!
+-------------------F
((A:0.43155,B:0.43155):0.36694,(((C:0.17350,D:0.17350):0.05278,
E:0.22628):0.09836,F:0.32463):0.47385)
Реконструкция методом Neighbor-Joining:
*программа eneighbor
+----C
+--2
+--3 +-----D
! !
! +-----E
!
--4-----------F
!
! +--------------A
+-------------------------1
+----------B
(((C:0.15563,D:0.19137):0.08246,E:0.19659):0.02708,F:0.39247,(A:0.50707,
B:0.35603):0.86503)
Реконструкция методом Maximum Likelihood:
*программа ednaml
+------D
+--------5
! +------C
+-----------4
! !
+---------------3 +--------F
! !
+---------2 +-----------E
! !
! +-------------B
--1
!
!
+--------------------A
(B:0.37622,(E:0.17030,(F:0.47787,(D:0.20197,C:0.16455):0.06927):0.06484):1.41918,
A:0.82072)
Реконструкция методом Parsimony:
*программа ednapars
+------D
+--------5
! +------C
+-----------4
! !
+-----------3 +--------E
! !
+--2 +-----------F
! !
! +-----------B
--1
!
!
+--------------A
(((F,(E,(D,C))),B),A)
| Ветвь | Исходное дерево модели |
UPGMA | NJ | ML | Parsimony |
| ABCDEF | |||||
| 110000 | + | + | + | + | + |
| 001100 | + | + | + | + | + |
| 110001 | + | + | + | - | + |
| 110010 | - | - | - | + | - |