Отобранные бактерии
| Название | Мнемоника |
| Bacillus subtilis | BACSU |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Finegoldia magna | FINM2 |
| Lactobacillus acidophilus | LACAC |
| Lactococcus lactis | LACLM |
| Listeria monocytogenes | LISMO |
| Staphylococcus aureus | STAA1 |
Скобочная формула дерева
((CLOTE,FINM2),((LACAC,(ENTFA,LACLM)),(SAA1,(LISMO,BACSU))));
Изображение дерева
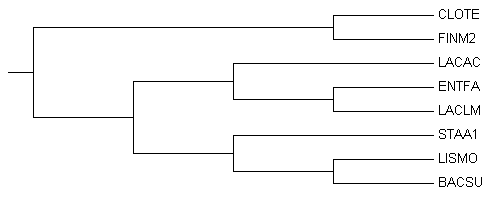
Ветви дерева
Дерево содержит 5 нетривиальных ветвей:
1) {CLOTE, FINM2} vs {LACAC, ENTFA, LACLM, STAA1, LISMO, BACSU}
2) {CLOTE, FINM2, LACAC, ENTFA, LACLM} vs {STAA1, LISMO, BACSU}
3) {LACAC, ENTFA, LACLM} vs {CLOTE, FINM2, STAA1, LISMO, BACSU}
4) {ENTFA, LACLM} vs {CLOTE, FINM2, LACAC, STAA1, LISMO, BACSU}
5) {CLOTE, FINM2, LACAC, ENTFA, LACLM, STAA1} vs {LISMO, BACSU}
Таксономия
Есть ветви, выделяющие порядки Clostridiales (CLOTE,FINM2), Lactobacillales (LACAC,ENTFA,LACLM), Bacillales (SAA1,LISMO,BACSU). И классы Bacilli и Clostridia.
Реконструкция дерева программой fprotpars
Выбранный белок - IF2. Выравниваю программой muscle.
Программа выдала одно дерево.
Скобочная формула:
(((BACSU,(STAA1,CLOTE)),(LISMO,(ENTFA,(LACLM,LACAC)))),FINM2);
Изображение:
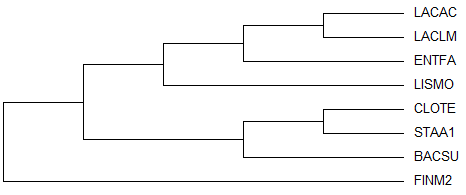
В правильном дереве CLOTE и FINM составляют кладу. fprotpars помещает CLOTE рядом с представителями отряда Bacillales, а LISMO рядом с представителями отряда Lactobacillales. В одной кладе, как и на правильном дереве оказались только Lactobacillales. Реконструкция дерева не очень хорошая.
Матрица расстояний
FINM2 LACAC CLOTE LACLM ENTFA BACSU LISMO STAA1
FINM2 0.000000 0.935808 0.656026 0.982581 0.877902 0.718460 0.793737 0.723231
LACAC 0.935808 0.000000 0.867984 0.876767 0.672759 0.711451 0.748716 0.728854
CLOTE 0.656026 0.867984 0.000000 0.846511 0.738504 0.650974 0.682834 0.652525
LACLM 0.982581 0.876767 0.846511 0.000000 0.566235 0.701363 0.662440 0.653701
ENTFA 0.877902 0.672759 0.738504 0.566235 0.000000 0.563546 0.513440 0.522920
BACSU 0.718460 0.711451 0.650974 0.701363 0.563546 0.000000 0.416163 0.414624
LISMO 0.793737 0.748716 0.682834 0.662440 0.513440 0.416163 0.000000 0.393263
STAA1 0.723231 0.728854 0.652525 0.653701 0.522920 0.414624 0.393263 0.000000
Расстояние между LACAC и FINM2 - 0.935808. Между CLOTE и FINM2 - 0.656026. Между CLOTE и LACAC - 0.867984. d(LACAC, FINM2) > d(CLOTE, FINM2). Если бы пространство было ультраметрическим, то d(FINM2, LACAC)=d(CLOTE, LACAC). Но в данном случае расстояния отклоняются от ультраметричности на 0.67824.
1) d(FINM2,CLOTE) + d(LACAC,ENTFA) = 1.328785;
2) d(CLOTE,ENTFA) + d(FINM2,LACAC) = 1.674312;
3) d(CLOTE,LACAC) + d(FINM2,ENTFA) = 1.745886;
Если предположить, что должны быть равны суммы 2) и 3), то отклонение от аддитивности - 0.071574.
Реконструкция дерева программой fneighbor
Neighbor-Joining
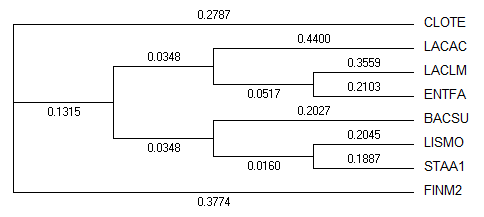
UPGMA

Дерево, построенное по алгоритму NJ будет похоже на правильное, если его укоренить так, чтобы CLOTE и FINM2 составляли одну кладу. Единственное отличие от "правильного": наличие ветви (LISMO, STAA1), тогда как в "правильносм" ветвь (LISMO.BACSU).
Дерево, построенное по алгоритму UPGMA имеет одну общую ветку с правильным деревом (FINM2, CLOTE). А представители класса Bacillales как бы происходят из класса Lactobacillales. Так происходит, вероятно, потому что UPGMA учитывает гипотезу молекулярных часов. А получается, что расстояние от ENTFA до BACSU почти такое же, как расстояние от ENTFA до (LISMO, STAA1), и эти расстояние больше, чем от BACSU до (ENTFA, LISMO). То есть, выполняется "ультраметричность".
Деревья, построенные программой fneighbor не имеют общих ветвей с деревьями, построенными с помощью fprotpars.



